https://doi.org/10.22319/rmcp.v14i4.6354
Artículo
Evaluación de resistencia a antibióticos en muestras de heces de terneros con diarrea en la región Cajamarca, Perú
Marco Antonio Cabrera González a
Héctor Vladimir Vásquez Pérez b
Carlos Quilcate-Pairazamán b
José Bazán-Arce a
Medali Cueva-Rodríguez a*
a Instituto Nacional de Innovación Agraria (INIA). Estación Experimental Baños del Inca. Dirección de Desarrollo Tecnológico Agrario, Jr. s/n Wiracocha, Baños del Inca, Cajamarca 06004, Perú.
b INIA. Dirección de Desarrollo Tecnológico Agrario, La Molina. Lima. Perú.
*Autor de correspondencia: mcuevar@unc.edu.pe
Resumen:
La diarrea está asociada con bacterias infecciosas que ocasionan mortalidad en terneros como Escherichia coli, representando un problema para los productores de leche y carne a nivel global, provocando grandes pérdidas económicas. En este estudio se evaluó la resistencia a cepas de E. coli aisladas de heces diarreicas de terneros recién nacidos de la región Cajamarca. Se recolectaron 52 muestras de heces de terneros de cinco provincias de la región Cajamarca para el aislamiento de E. coli en agar MacConkey con sorbitol. La identificación molecular de E. coli se realizó mediante la amplificación del gen uidA por PCR convencional y luego se evaluó la susceptibilidad/resistencia a antibióticos utilizando la metodología de Kirby-Bauer y el uso de discos de antibiótico con neomicina, tetraciclina, sulfametoxazol-trimetroprim y enrofloxacina. Los resultados fueron que el 96.15 % de cepas de E. coli fueron resistentes a tetraciclina, el 51.92 % a sulfametropim, el 26.92 % a neomicina y el 9.61 % a enrofloxacina. También se demostró que el 30.76 % presentaban resistencia a dos fármacos, el 19.23 % a tres fármacos y el 5.76 % a cuatro fármacos; se encontró diferencia significativa de resistencia a tetraciclina (P<0.0001). Se concluye que los terneros neonatos de la región Cajamarca que presentaban diarrea son portadores de E. coli resistentes a antibióticos, representando un problema para los criadores de ganado vacuno, ya que estas cepas pueden causar la muerte de los animales y contribuyen a la diseminación de la resistencia de antibióticos.
Palabras clave: Resistencia, Antibióticos, E. coli, Terneros, Diarrea.
Recibido: 17/11/2022
Aceptado: 05/05/2023
Introducción
La diarrea en terneros ha sido relacionada con patógenos infecciosos, representando un reto para los que se dedican a la producción de leche y carne a nivel global(1). Las bacterias infecciosas más importantes que causan la mortalidad asociada a la diarrea en terneros son E. coli y Salmonella(2), generando grandes pérdidas económicas si no se trata a tiempo la enfermedad con antimicrobianos apropiados y terapia de apoyo(2,3). Se han utilizado los antibióticos en animales para el tratamiento de enfermedades, prevención y control de infecciones comunes(4,5); sin embargo, el uso inadecuado y excesivo de antibióticos contribuye a la resistencia antimicrobiana amenazando la salud de animales y humanos(6). En relación a los animales que son destinados al sacrificio y finalmente al consumo de los humanos, estos actúan como reservorios de cepas resistentes a los antimicrobianos(7).
Por ejemplo, se ha reportado que, en las granjas lecheras de Egipto, se han aislado cepas de E. coli a partir de heces diarreicas de terneros que fueron resistentes a ampicilina(8). Las terneras eliminan frecuentemente microorganismos a través de sus heces, generando la diseminación de bacterias en el ambiente de la granja, lo que podría causar infecciones en los demás animales. Se han obtenido aislamientos de E. coli provenientes de heces de terneras lecheras que presentan resistencia a múltiples antibióticos del grupo de las fluoroquinolonas y se determinó el gen iucD como el más prevalente(9). Asimismo, en otros estudios se menciona sobre el operón de aerobactina (iucD), que produce cuatro tipos de sideróforos: enterobactina, salmoquelina, aerobactina y yersiniabactina. Los genes involucrados en la biosíntesis de sideróforos son encontrados en cepas uropatógenas (UPEC) y cepas comensales; sin embargo, salmoquelina, aerobactina y yersiniabactina son localizados en islas de patogenicidad asociados a UPEC, pero no en cepas comensales, sugiriendo que los sistemas de captación de hierro fueron adquiridos por transferencia horizontal de genes. La aerobactina es un sideróforo presente en la mayoría de las cepas UPEC, teniendo una gran estabilidad en la unión al Fe3+ y es uno de los encargados en el secuestro de hierro durante una infección del tracto urinario (ITU), la combinación de genes de adherencia/captación de hierro/toxicidad muestra la elevada virulencia y el potencial de daño que poseen las cepas para causar una ITU(9,10).
Por otro lado, la red francesa de monitoreo sobre resistencia a los antimicrobianos en animales enfermos indicó que cepas de E. coli portadoras de la mayoría de resistencias han sido aisladas de heces diarreicas de terneros neonatales(10), siendo la amoxicilina, tetraciclina y estreptomicina los principales antibióticos a los que se ha generado resistencia(7,11).
Es necesario reportar la resistencia que se viene generando a múltiples tipos de antibióticos para tener en cuenta su control y uso adecuado en bovinos, debido a que representa un peligro para la salud pública. Además, existen muy pocos estudios de resistencia a antibióticos en bovinos neonatos en el Perú, siendo este estudio un aporte para tener conocimiento de la situación actual. El objetivo de la investigación fue evaluar la resistencia de antibióticos de cepas de E. coli aisladas de heces diarreicas de terneros recién nacidos provenientes de cinco provincias de Cajamarca.
Material y métodos
Localización del estudio
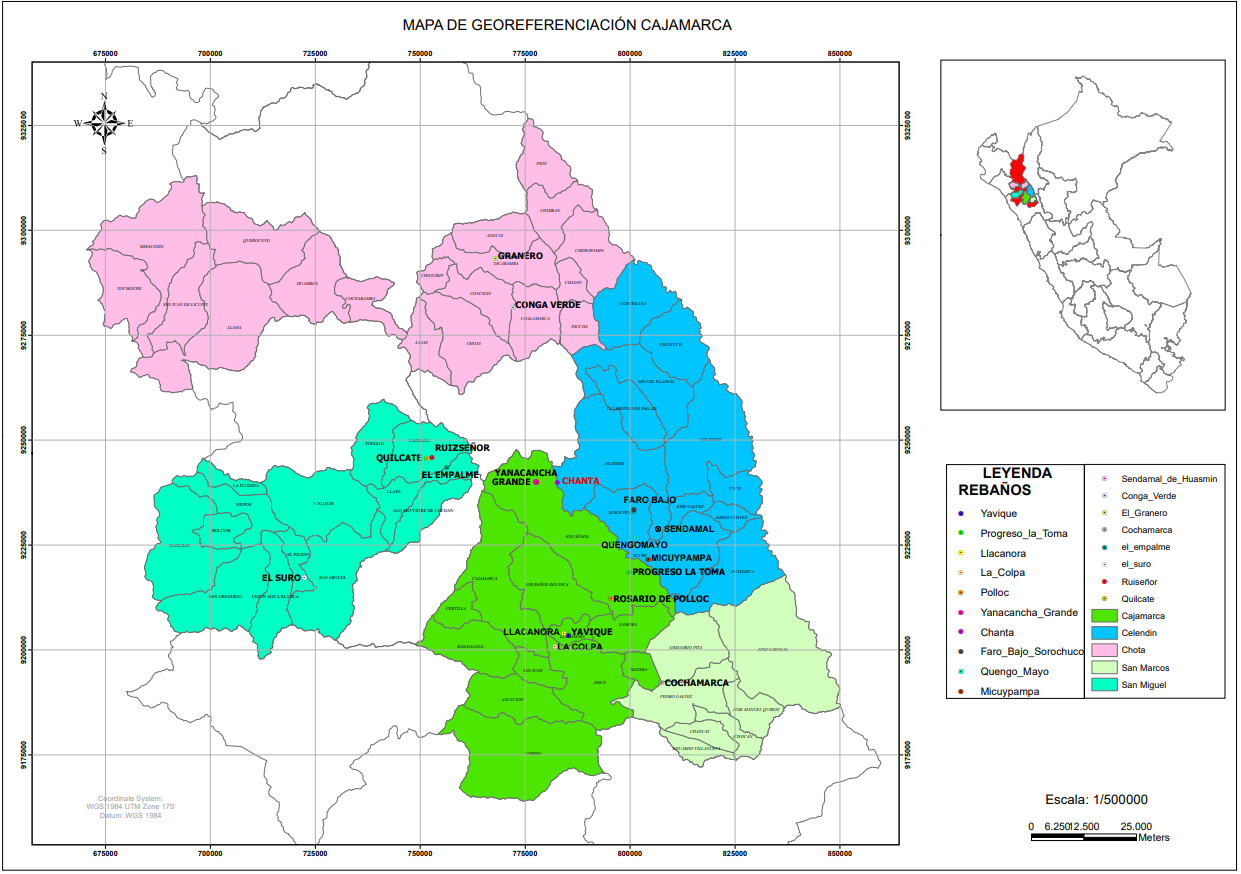
Se trabajó en 18 establos productores de leche bajo un sistema semi intensivo, los cuales están ubicados en la provincia de Chota, San Miguel, Celendín, San Marcos y Cajamarca, región de Cajamarca, Perú. Se logró recolectar un total de 52 muestras de terneros con diarrea hasta el primer mes de vida, de los cuales 35 terneros fueron machos y 17 terneros hembras, en época de lluvia (noviembre 2020 – mayo 2021) (Figura 1). Las muestras de heces (aproximadamente 3 g) se obtuvieron de manera directa del recto mediante el uso de bolsas estériles de polietileno de primer uso, estas muestras se identificaron y se llevaron en caja de tecnopor conteniendo hielo al laboratorio de Biotecnología en Sanidad Animal de la Estación Experimental Agraria Baños del Inca, donde se realizó el aislamiento de E. coli del total de las muestras.
Figura 1: Ubicación geográfica de los establos que participaron en el estudio, de las provincias de Chota, San Miguel, Celendín, San Marcos y Cajamarca, región de Cajamarca, Perú
Aislamiento e identificación de Escherichia coli
Con una asa bacteriológica estéril se sembraron 300 µl de heces en agar MacConkey II con sorbitol (Becton, Dickinson and Company® Loveton Circle Sparks, MD 21152, USA), posteriormente se incubó a 37 °C durante 24 h en estufa (Estufa universal Memmert UN-110/ Schwabach/Alemania), se aislaron colonias con morfología y desarrollo típico como son colonias de color rojo brillante las que se consideraron como E. coli.
Extracción de ácido desoxirribonucleico (ADN) de Escherichia coli
Tres colonias de E. coli seleccionadas que crecieron en el agar MacConkey II con sorbitol se cultivaron en medio líquido de crecimiento y multiplicación microbiana 2xYT medium (Sigma, REF. Y2377) a 37 °C por 18 h; el crecimiento bacteriano se determinó mediante concentración de valores predispuestos en el espectofotómetro (PCR MAX Lambda 64272, Bibby Scientific Ltd. Reino Unido), se realizó el cálculo de las Unidades Formadoras de Colonia (UFC) a 600 nm, determinando el crecimiento de bacterias viables en el medio de crecimiento. Para la obtención de la plantilla de ADN de E. coli se utilizó el kit de purificación Wizard® Genomic DNA (Promega, REF. A1120), con las indicaciones del fabricante, se almacenó el ADN en microtubos de polipropileno (Eppendorf™) de 1,500 µl en refrigeración, para lo cual se utilizó una refrigeradora Samsung, RT35K5930S8/PE, Samsung, México de 4 °C, utilizado para los distintos procesos de Reacción en Cadena de la Polimerasa (PCR).
Identificación molecular de Escherichia coli
La identificación molecular de E. coli se realizó mediante PCR empleando “cebadores” (F5’-TCAGCGCGAAGTCTTTATACC-3’, R5’-CGTCGGTAATCACCATTCCC-3’), para la amplificación del gen uidA (248 pb)(11,12,13). En la reacción de PCR se utilizó 1 µl (10 mM) de F y R de cada cebador, 7.7 µl de agua de grado molecular y 12.5 µl de G2 Green Master Mix (Promega, Madison, EE. UU), como templado se utilizó 2.8 µl de ADN a concentración de 50 µg/ml. El perfil térmico de la reacción de PCR fue: desnaturalización 94 °C/ 2 min, 25 ciclos desnaturalización 94 °C/30 seg, hibridación 55 oC/30 seg, extensión 72 °C/45 seg; extensión final 72 °C/2 min. Como control positivo se utilizó la cepa de referencia JM 109 de Escherichia coli (Promega, REF L2004). Los fragmentos de ADN amplificado (248 pb) para identificar cepas de E. coli se separaron por su peso molecular mediante electroforesis – agarosa 1%. Los fragmentos se analizaron mediante tinción en geles de agarosa con Sybr Green (Thermo Fisher) y se observaron en un transiluminador UV 302 nm Labnet U1001 Taiwán.
Prueba de susceptibilidad a los antimicrobianos
Para la evaluación de susceptibilidad/resistencia de las cepas de E. coli a los antibióticos se utilizó la metodología de Kirby-Bauer donde se tuvo como referencia parámetros establecidos para bacterias que se aislaron de animales por el Instituto de Estándares Clínicos y de Laboratorio (CLSI)(14) (Cuadro 1). Antes de la realización de la prueba de susceptibilidad se preparó agar Mueller Hinton (Merck KGaA 64271, Darmstact Germany) específico para determinar la sensibilidad de patógenos de importancia clínica según instrucciones del fabricante, y se esterilizó en autoclave (Autoclave digital automática AVDA050 Litros, Biogenics Lab , Perú); posteriormente se vertió en placas Petri de 35 mm de diámetro / 10 mm de alto y fueron sembrados dos o tres colonias aisladas con asa bacteriológica, se incubaron utilizando incubadora (Memmert CO2 ICO50 GmbH + Co. KG) en condiciones de aerobiosis por 18 h a 37 °C. La susceptibilidad a los antimicrobianos de todas las colonias aisladas se determinó frente a neomicina-N 30 μg, tetraciclina-TE 30 μg, sulfametoxazol-trimetropim-SXT 25 μg y enrofloxacina-ENR 5 μg (Discos - Thermo Scientific™); la sensibilidad de las cepas aisladas se clasificó como sensible, intermedio o resistente midiendo el halo de inhibición de acuerdo a los parámetros establecidos por el CLSI(14).
Cuadro 1: Interpretación de las pruebas de sensibilidad a antibióticos, por el método de difusión por discos (diámetro de la zona de inhibición)
Antibiótico | Concentración del disco (μg) | Sensible | Resistencia intermedia | Resistente |
Tetraciclina | 30 | ≥19 | 15-18 | ≤14 |
Sulfametropim | 25 | ≥16 | 11-15 | ≤10 |
Neomicina | 30 | ≥17 | 13-16 | ≤12 |
Enrofloxacina | 5 | ≥23 | 17-22 | ≤16 |
Análisis estadístico
Los resultados se analizaron mediante el software Graph Pad Prism 9.3.1 (Prism Software, Irvine, CA, USA). La normalidad de los datos fue determinada mediante “Kolmogorov-Smirnoff”. El análisis de varianza (ANOVA), seguido por el análisis de comparaciones múltiples de “Tukey” para evaluaciones entre antibióticos (parámetros relacionados a sensibilidad, resistencia). La información obtenida se consideró estadísticamente significativa a una P<0.05.
Resultados
Del total de muestras se seleccionaron un total de 52 con crecimiento positivo de E. coli en agar Mack Conkey II al Sorbitol. La identificación molecular de E. coli presente en heces de terneros con diarrea, se amplificó el gen uidA el cual codifica la enzima β–glucoronidasa(12,13,14). El procesamiento de los productos de PCR fue analizado mediante electroforesis en gel de agarosa al 1 %, método simple y eficaz para separar, identificar y purificar fragmentos de ADN con un tamaño molecular de 0.5 a 25 kb. Se observaron bandas electroforéticas del tamaño esperado: 248 pb positivas para la región amplificada del gen uidA. El mismo fue detectado en las 52 muestras analizadas, evidenciando la identificación de E. coli, dado que este gen es específico para la bacteria (Figura 2) de lo cual el 63.30 % (n=35) correspondían a terneros machos y el 32.69 % (n=17) procedían de terneros hembras.
Figura 2: Amplificación del gen uidA de muestras de heces de terneros mediante electroforesis en geles de agarosa (1%)
Carril 1 marcador de 100 pb. Muestras positivas en todos los carriles n=52.
Se analizó la presentación de susceptibilidad/resistencia a los fármacos de muestras de heces diarreicas de terneros donde se pudieron observar diferentes porcentajes de resistencia; gran parte de cepas de E. coli presentaron resistencia a la tetraciclina (96.15 %, 50/52) así mismo, mayor al 50 % de las muestras fueron resistentes a sulfametoxazol-trimetropim (51.92 %, 27/52), seguido con un porcentaje importante de resistencia a la neomicina (26.92 %, 14/52), además, se observó menor resistencia a enrofloxacina (9.61 %, 5/52) (Figura 3).
Figura 3: Porcentaje de cepas de E. coli con características de resistencia a cuatro fármacos
Se determinó también la presentación de la resistencia de antibióticos en cuanto a la variación de las cepas aisladas en cada ternero, la mayoría presentaban resistencia a uno (42.30 %, 22/52) y dos (30.76 %, 16/52) fármacos, el problema se agrava en un número importante de terneros presentándose resistencia múltiple en las cepas aisladas de E. coli observándose resistencia a tres (19.23 %, 10/52) y cuatro (5.76 %, 3/52) fármacos, siendo la resistencia a tetraciclina la más común en todos; además, la resistencia a enrofloxacina fue la que en menor proporción se presentó en las cepas aisladas (Figura 4).
Figura 4: Porcentaje de resistencia múltiple de fármacos utilizados en el control de diarrea en terneros
Se observó que la presentación de resistencia se mostró tanto en machos y hembras, observándose porcentajes de resistencia mayores en machos para tetraciclina (68 %) y neomicina (64.28 %) respectivamente; pero curiosamente en cuanto a las hembras pudo observarse que la resistencia a los fármacos sulfametropim (77.70 %), enrofloxacina (80 %) fue mayor con respecto a los machos (Figura 5).
Figura 5: Porcentaje de los fármacos más utilizado en el control de colibacilosis en terneros
Al análisis estadístico pudo encontrarse diferencia significativa (P<0.0001) en cuanto a la presentación de resistencia de las cepas de E. coli procedente de muestras de terneros con diarrea a la tetraciclina (Figura 6).
Figura 6: Diferencia estadística de resistencia de cepas de E. coli a tetraciclina
(P<0.0001). Los datos están expresados en valores absolutos y porcentaje (%).
Discusión
La E. coli se constituye como uno de los principales agentes bacterianos en producir infección urinaria en animales, septicemia y diarrea en animales de granja; el fenómeno de resistencia que expresan las cepas de E. coli a los fármacos usados en su control presentan fallo terapéutico, además, se están observando muchos casos de resistencia múltiple que aumenta a nivel mundial, convirtiéndose la diseminación de la resistencia en un problema de salud pública(15,16).
En esta investigación se pudo determinar diferentes características de resistencia a los antibióticos y con distintos porcentajes, lo cual ha permitido determinar que el fallo terapéutico a los antibióticos que expresa la bacteria en terrenos criados en los establos de ganado productores de leche en el región de Cajamarca – Perú está muy difundido, lográndose identificar que todas las cepas aisladas de E. coli muestran resistencia; así se pudo observar mayor porcentaje de resistencia a tetraciclina y con un importante porcentaje de resistencia múltiple con mayor prevalencia a dos fármacos, seguido a tres y cuatro antibióticos evaluados.
Los datos obtenidos de resistencia a los antibióticos en la región Cajamarca, permite mencionar que se da por la falta de registros de control sanitario en los rebaños, haciendo difícil realizar la trazabilidad de los fármacos utilizados y según versión de los propietarios algunos de estos animales cuando presentan esta signología de la enfermedad son tratados con antibióticos y otros no, pero todos presentaron resistencia al menos a un antibiótico analizado, lo que permite mencionar que la resistencia podría deberse al mal uso de antibióticos por parte de los criadores, siendo utilizados de manera frecuente(17) y se recomienda el monitoreo del ternero mediante reconstitución de electrolitos y en lo posible no administrar antibióticos por la presentación de resistencia a estos fármacos(18).
Otra posible causa de esta resistencia generalizada a los antibióticos podría ser que la mayoría de terneros tratados y principalmente en terneros no tratados, posiblemente este fenómeno se deba a que la leche y calostro provenientes de vacas que han sido tratadas con algún antibiótico facilita la presencia de residuos de antibióticos en la leche, aumentando la presión de selección de cepas de E. coli, con lo cual se seleccionan cepas resistentes, praxis muy instaurada en la ganadería regional de Cajamarca(19,20,21) además, se puede asumir que hay difusión de genes entre cepas comensales y patógenas de resistencia a los antibióticos entre animales y rebaños incrementando los niveles de resistencia(22,23,24).
El patrón de resistencia observado en cepas aisladas de E. coli tiene un orden de mayor a menor prevalencia la tetraciclina, sulfametropim, neomicina y enrofloxacina en menor proporción, resultados comunes que también fueron obtenidos por otros investigadores que reportan un patrón de resistencia a tetraciclinas, sulfonamidas, penicilinas y fluoroquinolonas(25), cefalotina, tetraciclina, trimetoprima-sulfadiazina, ampicilina(26), el patrón de multirresistencia más común fue ampicilina-kanamicina-estreptomicina-sulfametoxazol-tetraciclina(27).
El fenotipo de resistencia más frecuente de cepas de E. coli fue a la tetraciclina 96.15 % (50/52), esto posiblemente se debe a que cepas de E. coli porten fenotipos resistentes a tetraciclina por el uso inadecuado del fármaco por parte de los productores, que ha generado mayor presión de selección en cepas portadoras de genes que confieren resistencia a tetraciclina, contribuyendo mediante difusión de cepas la transferencia de genes de resistencia a los antimicrobianos(28); además, estas cepas se constituyen en posibles fuentes que diseminan la resistencia al medio ambiente cuando se esparce el estiércol en las zonas de pastoreo como el abono(29).
La presencia de resistencia de las cepas locales de E. coli a tetraciclina es generalizada con base en su utilización de amplio espectro en salud animal, como reservorio de bacterias gram negativas con genes de resistencia a tetraciclina como fuente de infección y con mayor prevalencia en E. coli causante de diarrea en terneros, problemática frecuente que también ha sido reportada en otras investigaciones con resultados similares(30,31,32).
Es muy importante determinar los genes que están involucrados en estos procesos de resistencia relacionados a la utilización de estos fármacos, como genes de adhesión, transportadores de hierro(33,34), como es el caso del gen iucD(9,35), además, los procesos de transferencia horizontal de genes, la presión de selección, que provoca el uso indiscriminado de antibióticos(36,37), todo lo mencionado es conocimiento necesario para evaluar los procesos de tratamiento y control de diarrea en terneros en la región de Cajamarca.
Conclusiones e implicaciones
Las cepas de E. coli causantes de diarrea neonatal en terneros en la región Cajamarca presenta una prevalencia de resistencia múltiple a los fármacos utilizados por los ganaderos en su control, observándose un perfil de resistencia a tetraciclina, sulfametropim, neomicina, y enrofloxacina (TSNE), como resultado a la mala utilización de fármacos que aumentan la presión de selección sobre cepas que fomentan la expresión de genes de virulencia y resistencia a los antibióticos convirtiéndose en focos de trasmisión de resistencia tanto para animales como en humanos por la posibilidad de transmisión horizontal entre microorganismos. Considerando que se debería realizar un diagnóstico definitivo, determinado el agente etiológico y la susceptibilidad a los antibióticos, luego aplicar de manera correcta el antibiótico seleccionado en dosis y frecuencia correcta. Además, en base a los resultados obtenidos es necesario determinar los genes de resistencia involucrados en resistencia múltiple a los antibióticos.
Literatura citada: