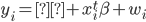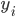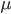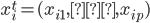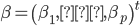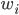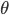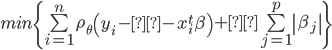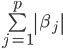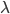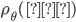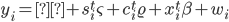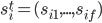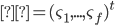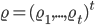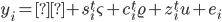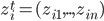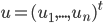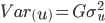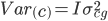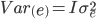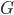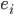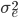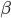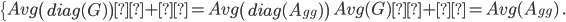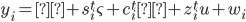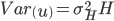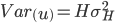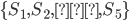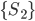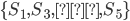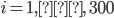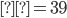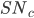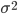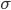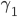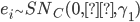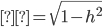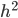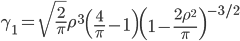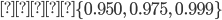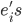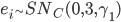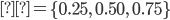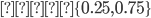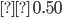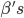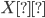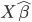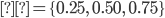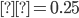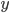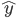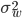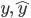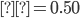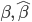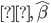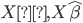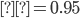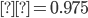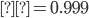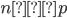https://doi.org/10.22319/rmcp.v14i1.6182
Artículo
Regresión cuantil para predicción de caracteres complejos en bovinos Suizo Europeo usando marcadores SNP y pedigrí
Jonathan Emanuel Valerio-Hernández a
Paulino Pérez-Rodríguez b*
Agustín Ruíz-Flores a
a Universidad Autónoma Chapingo. Posgrado en Producción Animal. Carretera Federal México-Texcoco Km 38.5, 56227, Texcoco, Estado de México, México.
b Colegio de Postgraduados. Socio Economía Estadística e Informática. Carretera Federal México-Texcoco Km 36.5, 56230, Texcoco, Estado de México.
*Autor para correspondencia: perpdgo@colpos.mx
Resumen:
Los modelos de predicción genómica generalmente suponen que los errores se distribuyen como variables aleatorias normales, independientes e idénticamente distribuidas con media cero e igual varianza. Esto no siempre se cumple, además puede haber fenotipos distantes de la media poblacional, los que usualmente se eliminan al realizar la predicción. La regresión cuantil (QR) afronta aspectos estadísticos como alta dimensionalidad, multicolinealidad y distribución fenotípica diferente de la normal. El objetivo de este trabajo fue comparar QR utilizando información de marcadores y pedigrí con los métodos alternativos tales como mejor predicción lineal insesgada genómica (GBLUP) y mejor predicción lineal insesgada genómica en un solo paso (ssGBLUP) para analizar los pesos al nacimiento (PN), destete (PD) y al año (PA) de bovinos Suizo Europeo y datos simulados con diferente grado de asimetría y proporción de datos atípicos. La capacidad predictiva de los modelos se evaluó mediante validación cruzada. El desempeño predictivo de QR tanto sólo con información de marcadores como con marcadores más pedigrí, con el conjunto de datos reales, fue mejor que las metodologías GBLUP y ssGBLUP para PD y PA. Para PN GBLUP y ssGBLUP fueron mejores, sin embargo, solo se utilizaron los cuantiles 0.25, 0.50 y 0.75, y la distribución de PN no fue asimétrica. En el experimento de datos simulados, las correlaciones entre efectos de marcador “verdadero” y efectos estimados, así como las correlaciones de señales “verdaderas” y estimadas fueron más altas cuando se usó QR comparado con GBLUP. Las ventajas de QR fueron más notorias con distribución asimétrica de los fenotipos y con mayor proporción de datos atípicos, como fue el caso del conjunto de datos simulados.
Palabras clave: Regresión cuantil, GBLUP, ssGBLUP.
Recibido: 30/03/2022
Aceptado: 04/08/2022
Introducción
La motivación principal del método de regresión cuantil (QR) reside en que la mayoría de los modelos para evaluación genética asumen normalidad, lo que no siempre se cumple. Otro problema es que en ocasiones registros fenotípicos muy alejados del promedio de la población se consideran como errores de registro o datos atípicos y por consiguiente se eliminan de los análisis, visto desde el punto de vista genómico se está perdiendo información valiosa de marcadores asociados a ciertas regiones de ADN con fuerte influencia en características de interés.
Con el método QR se obtienen resultados robustos y una visión amplia de las variables explicativas sobre las dependientes(1). Los datos generados a partir de experimentos ómicos frecuentemente son complejos y de gran dimensión, por consiguiente existe un desafío estadístico para extraer información biológica relevante del gran volumen de datos(2,3). El uso de un enfoque sólido como QR hace que la inferencia sea menos sesgada y esté menos sujeta a falsos positivos(2). En estudios recientes que utilizan QR, se describen aplicaciones diversas como estudios de asociación genética(4), genética de poblaciones(5), expresión génica(6,7) y selección genómica(8–10).
Uno de los primeros estudios donde se utilizó QR para predecir el mérito genético individual lo presentaron Nascimento et al(11), quienes utilizaron datos simulados encontrando ventajas al usar QR frente a metodologías convencionales. El mismo año(12) se publicaron resultados usando QR para ajustar curvas de crecimiento con datos de cerdos y marcadores moleculares; no solo ajustaron con éxito las curvas de crecimiento sino que identificaron marcadores de importancia asociados con la característica estudiada. Otro trabajo similar del mismo equipo de investigadores fue presentado por Nascimento et al(13), pero con datos de frijol. Recientemente Pérez-Rodríguez et al(10) extendieron el modelo de regresión cuantil para incluir información de pedigrí a través del uso de la matriz de relaciones genéticas aditivas, mejorando aún más la habilidad predictiva de los modelos y al mismo tiempo identificando las proporciones de las varianzas atribuidas a marcadores, relaciones entre individuos y el residual, lo cual permite obtener de manera precisa una partición de la varianza fenotípica.
El objetivo del presente estudio fue estudiar el poder predictivo del modelo de regresión cuantil utilizando datos simulados y datos reales (pesos al nacimiento, destete y al año) de bovinos Suizo Europeo y se consideraron los modelos: 1) QR con información de marcadores moleculares SNP (QRM), 2) QR incluyendo simultáneamente información de marcadores moleculares y genealógica derivada del pedigrí (QRH); 3) GBLUP el cual al igual que QRM solo incluyó información de marcadores moleculares, y 4) evaluación genómica en un solo paso (ssGBLUP) que incluyó información de marcadores y pedigrí.
Material y métodos
Genotipos
Se utilizó información de 300 animales (236 hembras, 64 machos) nacidos de 2001 a 2016 en ocho hatos ubicados en el Este, Centro y Oeste de México. Se recolectaron muestras de pelo para su genotipado por la compañía GeneSeek (Lincoln, https://www.neogen.com/, NE, EE. UU.), utilizando el panel GeneSeek® Genomic Profiler Bovine LDv.4, con 30,000 y 50,000 marcadores SNP, 150 animales con cada Chip. La genotipación se realizó en dos ocasiones distintas, inicialmente 150 individuos con el Chip de 30K y posteriormente otro grupo de 150 individuos con el Chip de 50K ya que el Chip de 30K no estuvo disponible en ese momento. Se utilizaron los SNP en común entre los chips de 30K y 50K (12,835 SNP). Se calcularon las proporciones de valores faltantes para cada marcador y para cada individuo. El promedio de valores faltantes por individuo fue 2.09 % con una desviación estándar de 7.50 %. La tasa de llamada promedio (proporción no faltante para cada marcador) fue 97.90 % con una desviación estándar del 4.66 %. Se eliminaron los marcadores con una tasa de llamada inferior al 95 %. Los genotipos se recodificaron como AA= 0, AB= 1 y BB= 2, de donde se obtuvo una matriz con 300 filas (individuos) y 12,835 columnas (marcadores) cuyas celdas toman valores en el conjunto 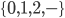 , donde “
, donde “ ” denota un valor faltante. Para los 12,835 marcadores en común de los dos chips, los valores faltantes se imputaron al azar, generando muestras de la distribución
” denota un valor faltante. Para los 12,835 marcadores en común de los dos chips, los valores faltantes se imputaron al azar, generando muestras de la distribución 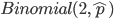 donde
donde 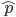 es la frecuencia del alelo mayor, calculado a partir de los genotipos de marcadores observados. Se eliminaron los marcadores monomórficos o aquellos con frecuencia de alelos menores (MAF) menor que 0.04. Después del control de calidad, 9,628 de los 12,835 SNP en común entre los dos chips estuvieron disponibles para análisis adicionales.
es la frecuencia del alelo mayor, calculado a partir de los genotipos de marcadores observados. Se eliminaron los marcadores monomórficos o aquellos con frecuencia de alelos menores (MAF) menor que 0.04. Después del control de calidad, 9,628 de los 12,835 SNP en común entre los dos chips estuvieron disponibles para análisis adicionales.
Fenotipos
La información fenotípica y de pedigrí de la población de ganado Suizo Europeo se obtuvo de la base de datos de la Asociación Mexicana de Criadores de Ganado Suizo de Registro. Los registros de pesos al nacer (PN), al destete (PD) y al año (PA) se utilizaron para el análisis. La edición de los fenotipos fue similar para PN, PD y PA, se descartaron los registros de animales no relacionados genéticamente con aquellos genotipados o con información faltante para hato, edad de la madre y manejo. Los grupos contemporáneos (GC) se definieron al eliminar los animales en GC de 2 individuos o con varianza igual a cero. Para PN los GC se definieron combinando los efectos del hato (8 hatos), año (1998 a 2016) y temporada de nacimiento; las temporadas de nacimiento se definieron considerando el calendario juliano, de 80 a 171 días, primavera; de 172 a 264 días, verano; de 265 a 354 días, otoño; de 355 a 366 días y de 1 a 79 días, invierno. Después de la edición de datos para PN se obtuvieron 330 registros. Para PD y PA los GC se definieron combinando los efectos del hato (6 hatos), año (de 1998 a 2016), temporada de nacimiento (igual que PN) y manejo. En el caso de PD los grupos de manejo se definieron de tres formas: terneros alimentados con leche de su madre; leche de su madre más alimento balanceado; y leche de su madre y nodriza más alimentación balanceada. Para PA los grupos de manejo se definieron de tres maneras: animales en pastoreo; animales en pastoreo con concentrado alimenticio; y animales estabulados con alimentación equilibrada. La edición de datos de PD y PA finalizó con 267 y 232 registros para análisis posteriores. En el Cuadro 1 se presenta un resumen del número de animales genotipados, y fenotipados para PN, PD y PA. La Figura 1 muestra las gráficas de violín para PN, PD y PA, la media muestral está representada por el punto rojo y la mediana muestral por la línea horizontal dentro del recuadro, de la gráfica es claro que las variables respuestas presentan una distribución asimétrica y los círculos con relleno sólido en la misma sugieren la presencia de datos atípicos.
Cuadro 1: Número de animales genotipados y fenotipados para el análisis de los pesos al nacer, al destete y al año de una población de ganado Suizo Europeo
Grupo | Peso al nacer | Peso al destete | Peso al año |
Genotipado | 300 | 300 | 300 |
Genotipado y fenotipado | 232 | 218 | 191 |
Fenotipados en QRM y GBLUP | 232 | 218 | 191 |
Fenotipados en QRH y ssGBLUP | 330 | 267 | 232 |
QRM=Regresión cuantil usando información de marcadores, QRH=Regresión cuantil usando información de marcadores y pedigrí, GBLUP=Mejor predictor lineal insesgado genómico, ssGBLUP=Evaluación genómica en un solo paso.
Figura 1: Gráficas de violín de peso al nacimiento (PN), al destete (PD) y al año (PA) en una población de bovinos Suizo Europeo
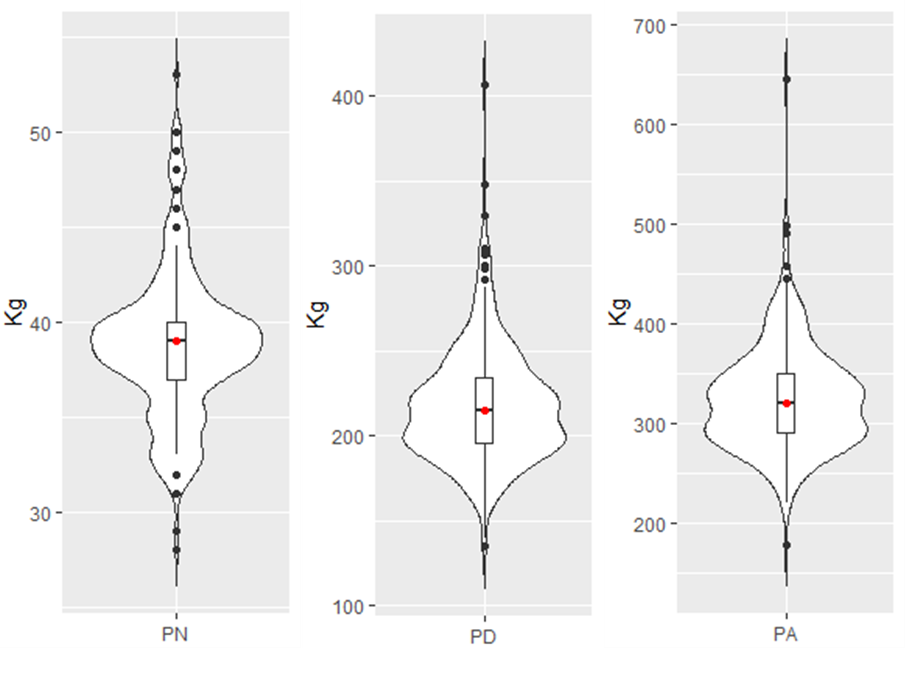
La media muestral está representada por el punto rojo y la mediana muestral por la línea horizontal dentro del recuadro
Modelos
Modelo de regresión cuantil con marcadores (QRM)
El modelo para regresión cuantil es:
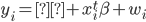 , ,
|
|
donde 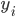 es el valor del fenotipo del i-ésimo animal;
es el valor del fenotipo del i-ésimo animal; 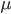 es un intercepto;
es un intercepto; 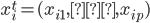 representa la i-ésima fila de la matriz de marcadores,
representa la i-ésima fila de la matriz de marcadores, 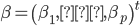 es el vector de coeficientes de regresión asociados con marcadores y
es el vector de coeficientes de regresión asociados con marcadores y 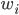 son variables aleatorias independientes tales que su cuantil
son variables aleatorias independientes tales que su cuantil 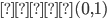 es cero. La estimación de los coeficientes de regresión para un cuantil de interés
es cero. La estimación de los coeficientes de regresión para un cuantil de interés 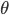 fijo se obtiene al resolver el problema de minimización siguiente:
fijo se obtiene al resolver el problema de minimización siguiente:
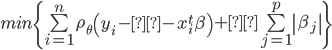 , ,
|
|
donde 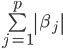 es la suma de los valores absolutos de los coeficientes de regresión;
es la suma de los valores absolutos de los coeficientes de regresión; 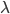 es el parámetro de penalización que controla la intensidad de la regularización; y
es el parámetro de penalización que controla la intensidad de la regularización; y 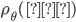 es la función definida como(1):
es la función definida como(1):
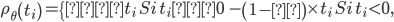
donde 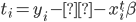 . Después de estimar los parámetros del modelo, los valores de cría estimados mediante marcadores (GEBV) se obtienen mediante la siguiente expresión:
. Después de estimar los parámetros del modelo, los valores de cría estimados mediante marcadores (GEBV) se obtienen mediante la siguiente expresión:
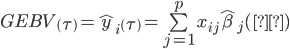 ,
,
donde 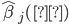 es el efecto del j-ésimo marcador, definido por la relación funcional obtenida para el cuantil de interés.
es el efecto del j-ésimo marcador, definido por la relación funcional obtenida para el cuantil de interés.
El modelo QR puede extenderse para incluir otros términos, en particular para las características de crecimiento, se utiliza el siguiente modelo:
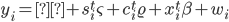 ,
,
donde 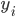 es el valor del fenotipo de la característica analizada (PN, PD o PA) del i-ésimo animal,
es el valor del fenotipo de la característica analizada (PN, PD o PA) del i-ésimo animal, 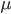 es un intercepto;
es un intercepto; 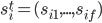 la i-ésima fila de la matriz de incidencia para los efectos fijos (sexo, edad de la madre, manejo),
la i-ésima fila de la matriz de incidencia para los efectos fijos (sexo, edad de la madre, manejo), 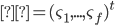 los coeficientes de regresión para los efectos fijos,
los coeficientes de regresión para los efectos fijos, 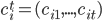 la i-ésima fila de la matriz de incidencia para efectos aleatorios de grupo contemporáneo (54, 43 y 37 para PN, PD y PA),
la i-ésima fila de la matriz de incidencia para efectos aleatorios de grupo contemporáneo (54, 43 y 37 para PN, PD y PA), 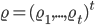 efectos aleatorios de grupo contemporáneo, el resto de los términos como se describieron anteriormente.
efectos aleatorios de grupo contemporáneo, el resto de los términos como se describieron anteriormente.
GBLUP
El modelo está dado por:
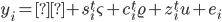 ,
,
donde 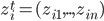 es la i-ésima fila de la matriz que conecta fenotipos con genotipos,
es la i-ésima fila de la matriz que conecta fenotipos con genotipos, 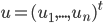 es el vector de efectos aleatorios para animales. Las varianzas genéticas aditiva, de grupo contemporáneo y residual se asumen
es el vector de efectos aleatorios para animales. Las varianzas genéticas aditiva, de grupo contemporáneo y residual se asumen 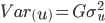 ,
, 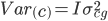 , y
, y 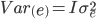 , respectivamente. La matriz de relaciones genómicas,
, respectivamente. La matriz de relaciones genómicas, 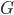 , se calcula como lo describen Lopez-Cruz et al(14) y Pérez-Rodríguez et al(15); brevemente, G = WW’/p, donde W es la matriz de marcadores estandarizada y centrada (cada marcador centrado restando la media de frecuencia de alelo y estandarizado, dividiendo por la desviación estándar de la muestra de la frecuencia de alelos), p es el número total de marcadores,
, se calcula como lo describen Lopez-Cruz et al(14) y Pérez-Rodríguez et al(15); brevemente, G = WW’/p, donde W es la matriz de marcadores estandarizada y centrada (cada marcador centrado restando la media de frecuencia de alelo y estandarizado, dividiendo por la desviación estándar de la muestra de la frecuencia de alelos), p es el número total de marcadores, 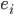 variables aleatorias normales e independientes con distribución normal con media 0 y varianza
variables aleatorias normales e independientes con distribución normal con media 0 y varianza 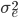 .
.
Modelo de regresión cuantil en un solo paso (QRH)
Este método se considera una extensión del modelo de cuantiles para una matriz de relaciones construida utilizando matrices de relaciones para animales genotipados y no genotipados y de los cuales se tiene disponible un pedigrí. La matriz que resulta se conoce en la literatura como matriz H(16,17), esta matriz está dada por:

donde, Agg es una submatriz de A para animales genotipados, Ga = βG + α; 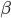 y
y  se obtienen al resolver el sistema de ecuaciones:
se obtienen al resolver el sistema de ecuaciones:
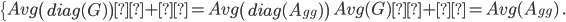
El modelo QRH está dado por:
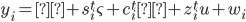 ,
,
donde 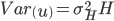 , el resto de los términos como se describieron previamente.
, el resto de los términos como se describieron previamente.
Modelo de regresión GBLUP en un solo paso (ssGBLUP)
El modelo ssGBLUP es equivalente al modelo GBLUP descrito anteriormente con la diferencia de que se reemplaza la matriz de relaciones genómicas G por la matriz de relaciones genéticas extendida H, se asume que 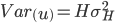 .
.
Validación cruzada
La capacidad predictiva de los modelos se evaluó mediante validación cruzada, la cual se realizó de la siguiente manera. El conjunto de datos se dividió en cinco grupos del mismo tamaño 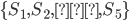 , 80 % de los datos se usaron para entrenamiento del modelo, el 20 % restante para validación. Por ejemplo,
, 80 % de los datos se usaron para entrenamiento del modelo, el 20 % restante para validación. Por ejemplo, 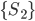 se usa como grupo de validación y el conjunto
se usa como grupo de validación y el conjunto 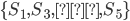 para entrenamiento del modelo. Los modelos se ajustaron utilizando el conjunto de entrenamiento, y el modelo ajustado se utilizó para obtener predicciones para el conjunto de validación. Este procedimiento se repitió cinco veces y se obtuvieron predicciones para cada grupo. Las correlaciones entre fenotipos observados y predichos se calcularon y promediaron para los conjuntos de prueba(18). Note que debido a que se trata de valores reales, no se conocen los valores de crías verdaderos, sino solamente se tienen los fenotipos observados, el modelo ajustado proporciona predicciones para valores de cría y las predicciones de otros efectos fijos y ambientales, con lo cual se obtiene una predicción del fenotipo mismo que se contrasta con el valor verdadero del fenotipo.
para entrenamiento del modelo. Los modelos se ajustaron utilizando el conjunto de entrenamiento, y el modelo ajustado se utilizó para obtener predicciones para el conjunto de validación. Este procedimiento se repitió cinco veces y se obtuvieron predicciones para cada grupo. Las correlaciones entre fenotipos observados y predichos se calcularon y promediaron para los conjuntos de prueba(18). Note que debido a que se trata de valores reales, no se conocen los valores de crías verdaderos, sino solamente se tienen los fenotipos observados, el modelo ajustado proporciona predicciones para valores de cría y las predicciones de otros efectos fijos y ambientales, con lo cual se obtiene una predicción del fenotipo mismo que se contrasta con el valor verdadero del fenotipo.
Simulación
Con la finalidad de evaluar el poder predictivo del modelo QR frente a GBLUP se realizó además una simulación de datos asimétricos con presencia de datos atípicos; la simulación del presente trabajo es análoga a la utilizada por Pérez-Rodríguez et al(10). La idea principal es resaltar que el modelo de regresión por cuantiles funciona de manera adecuada en la presencia de observaciones atípicas, varianzas no homogéneas y variables respuesta con respuestas con distribución asimétrica. En el contexto de selección, por ejemplo, no es poco usual contar con distribuciones asimétricas para los fenotipos debido al proceso mismo, ya que, si se selecciona para alguna característica Y, y si existe además de esto otra característica de interés O, entonces la distribución condicional de Y |O>o(19) es asimétrica. En el contexto de selección genómica es también usual encontrar subconjuntos de observaciones que difieren significativamente del resto y estas observaciones podrían considerarse como atípicas. Montesinos-López et al(20) propusieron un modelo con errores Laplace y mostraron que predice de manera adecuada aun en presencia de datos atípicos, el modelo propuesto es un caso especial del modelo de regresión cuantil que se estudia en el presente trabajo. Se consideraron los 9,628 SNP resultantes del control de calidad descrito anteriormente para 300 animales, la simulación de los datos se realizó considerando el modelo lineal:
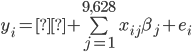 ,
,
donde 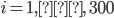 , con
, con 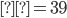 para PN, se supuso que los errores vienen de una distribución normal sesgada (
para PN, se supuso que los errores vienen de una distribución normal sesgada (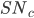 ) con media 0, varianza
) con media 0, varianza 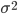 (parámetro de escala
(parámetro de escala 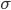 ) e índice de asimetría
) e índice de asimetría 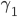 , es decir
, es decir 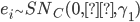 , con
, con 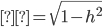 , se consideró
, se consideró 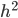 con un valor de 0.35,
con un valor de 0.35, 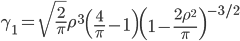 ,
, 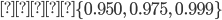 lo que lleva a diferentes grados de sesgo positivo. Solo se consideraron valores positivos de
lo que lleva a diferentes grados de sesgo positivo. Solo se consideraron valores positivos de 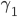 , ya que el sesgo negativo se obtiene simplemente cambiando el signo de las
, ya que el sesgo negativo se obtiene simplemente cambiando el signo de las 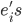 y por lo tanto las conclusiones obtenidas para el caso de sesgo positivo serán también válidas para el caso negativo(21,22). Se fijaron 50 marcadores con efecto diferente de cero simulándolos de una distribución normal con media 0 y varianza
y por lo tanto las conclusiones obtenidas para el caso de sesgo positivo serán también válidas para el caso negativo(21,22). Se fijaron 50 marcadores con efecto diferente de cero simulándolos de una distribución normal con media 0 y varianza 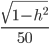 , el resto de los marcadores se establecieron en 0; las posiciones de los marcadores muestreados se tomaron al azar. Para introducir datos atípicos en los fenotipos, se generó cierta proporción de los residuos de
, el resto de los marcadores se establecieron en 0; las posiciones de los marcadores muestreados se tomaron al azar. Para introducir datos atípicos en los fenotipos, se generó cierta proporción de los residuos de 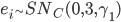 al azar, se consideraron dos proporciones, 5 y 10 %, por lo que se tomaron muestras de una mezcla de dos componentes de distribuciones normales sesgadas. Se generaron seis conjuntos de datos, tres diferentes coeficientes de asimetría 0.950, 0.975, 0.999 con sus dos alternativas de proporción de datos atípicos 5 % y 10 %. La distribución normal asimétrica se ha utilizado en predicción genómica(22) y también se ha sugerido su uso en selección canalizada(23). Una vez generados los datos se ajustó el modelo QRM con
al azar, se consideraron dos proporciones, 5 y 10 %, por lo que se tomaron muestras de una mezcla de dos componentes de distribuciones normales sesgadas. Se generaron seis conjuntos de datos, tres diferentes coeficientes de asimetría 0.950, 0.975, 0.999 con sus dos alternativas de proporción de datos atípicos 5 % y 10 %. La distribución normal asimétrica se ha utilizado en predicción genómica(22) y también se ha sugerido su uso en selección canalizada(23). Una vez generados los datos se ajustó el modelo QRM con 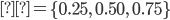 para compararlo con GBLUP. La selección de los cuantiles se realizó de acuerdo a Nascimento et al(11) quienes consideran estas tres posibilidades cuando la distribución de los fenotipos es asimétrica
para compararlo con GBLUP. La selección de los cuantiles se realizó de acuerdo a Nascimento et al(11) quienes consideran estas tres posibilidades cuando la distribución de los fenotipos es asimétrica 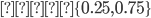 o bien cuando la distribución es simétrica
o bien cuando la distribución es simétrica 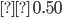 , pues nuestro interés fundamental en este trabajo se centró en la modelación de datos posiblemente asimétricos y con la presencia de valores atípicos. La selección de los parámetros también se realizó por conveniencia computacional ya que el ajuste del modelo se realiza mediante el uso de técnicas de cómputo intensivas basadas en cadenas de Markov Monte Carlo como se menciona en la sección de software y ajuste de los modelos. Para cada análisis se calculó la correlación entre los
, pues nuestro interés fundamental en este trabajo se centró en la modelación de datos posiblemente asimétricos y con la presencia de valores atípicos. La selección de los parámetros también se realizó por conveniencia computacional ya que el ajuste del modelo se realiza mediante el uso de técnicas de cómputo intensivas basadas en cadenas de Markov Monte Carlo como se menciona en la sección de software y ajuste de los modelos. Para cada análisis se calculó la correlación entre los 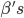 verdaderos y estimados, la correlación entre las señales verdaderas
verdaderos y estimados, la correlación entre las señales verdaderas 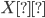 y estimadas
y estimadas 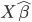 y el componente de varianza asociado con los residuos para cada modelo, el cual es una forma de evaluar la bondad de ajuste de los modelos. También se consideró el Criterio de Información de Desviación (DIC), éste se puede usar para seleccionar modelos candidatos; los modelos con menor DIC se prefieren contra modelos de mayor DIC(24).
y el componente de varianza asociado con los residuos para cada modelo, el cual es una forma de evaluar la bondad de ajuste de los modelos. También se consideró el Criterio de Información de Desviación (DIC), éste se puede usar para seleccionar modelos candidatos; los modelos con menor DIC se prefieren contra modelos de mayor DIC(24).
Software y ajuste de los modelos
Los modelos de regresión por cuantiles se ajustaron usando una estrategia computacional similar a la descrita por Pérez-Rodríguez et al(10). Las adaptaciones de los algoritmos para incluir efectos fijos y aleatorios no presentan gran dificultad computacional. Los códigos para el ajuste de los modelos se desarrollaron en los lenguajes de programación R(25) y C. Los códigos para el ajuste de los modelos fueron organizados de tal forma que puedan ser ejecutados fácilmente desde el software estadístico R y están disponibles solicitándolos al primer autor del presente estudio. En todos los casos se seleccionaron tres cuantiles, 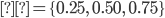 . Los modelos GBLUP y ssGBLUP se ajustaron con la librería de R BGLR(26).
. Los modelos GBLUP y ssGBLUP se ajustaron con la librería de R BGLR(26).
Resultados
Datos reales
Los Cuadros 2, 3 y 4 muestran los resultados del experimento realizado con datos de PN, PD y PA de una población de bovinos Suizo Europeo, evaluados bajo dos escenarios 1) solo con información de marcadores, y 2) información de marcadores y pedigrí. De manera general las correlaciones entre valores observados y predichos más altas se obtuvieron con QR, excepto para PN donde las correlaciones de GBLUP y ssGBLUP fueron más altas que las obtenidas con QRM y QRH, sin embargo, las correlaciones de QRM  y QRH
y QRH  fueron cercanas a las obtenidas con GBLUP y ssGBLUP (0.7902 vs 0.7924), (0.6981 vs 0.7055), respectivamente. Los valores de MSE más bajos se obtuvieron con QRM
fueron cercanas a las obtenidas con GBLUP y ssGBLUP (0.7902 vs 0.7924), (0.6981 vs 0.7055), respectivamente. Los valores de MSE más bajos se obtuvieron con QRM  y QRH
y QRH  en la característica de PD, mientras que en las características PN y PA los valores más bajos se obtuvieron con GBLUP y ssGBLUP. Los componentes de varianza asociados con el error obtenidos con QRM y QRH fueron menores que los obtenidos con GBLUP y ssGBLUP. Los valores de DIC más bajos de manera general se obtuvieron con QRM
en la característica de PD, mientras que en las características PN y PA los valores más bajos se obtuvieron con GBLUP y ssGBLUP. Los componentes de varianza asociados con el error obtenidos con QRM y QRH fueron menores que los obtenidos con GBLUP y ssGBLUP. Los valores de DIC más bajos de manera general se obtuvieron con QRM  y QRH
y QRH  , excepto para PN con el escenario de solo marcadores, donde el DIC más bajo se obtuvo con QRM
, excepto para PN con el escenario de solo marcadores, donde el DIC más bajo se obtuvo con QRM 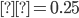 .
.
Cuadro 2: Promedios de la correlación Pearson y la desviación estándar (entre paréntesis) entre los valores fenotípicos observados (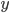 ) y valores fenotípicos predichos (
) y valores fenotípicos predichos (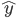 ), cuadrado medio del error, componentes de varianza asociados al error (
), cuadrado medio del error, componentes de varianza asociados al error (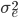 ,
, 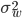 ) y criterio de información de desviación (DIC) para peso al nacimiento
) y criterio de información de desviación (DIC) para peso al nacimiento
Modelo | Cor(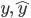 ) ) | MSE | 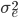 o o 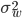
| DIC |
QRM 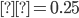 | 0.7521 | 3.9973 | 2.7260 | 513.5014 |
| (0.0753) | (1.6108) | (1.9762) | (531.5701) |
QRM 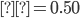 | 0.5619 | 7.3249 | 8.6297 | 970.7680 |
| (0.1501) | (0.4561) | (0.2660) | (6.9791) |
QRM  | 0.7902 | 3.6535 | 2.4268 | 716.4237 |
| (0.0766) | (0.0943) | (0.4829) | (35.7161) |
GBLUP | 0.7924 | 2.3269 | 3.0035 | 803.0675 |
| (0.0874) | (0.2063) | (0.5578) | (31.9814) |
QRH 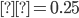 | 0.6713 | 3.5026 | 2.3645 | 872.3949 |
| (0.1329) | (1.2848) | (1.9670) | (432.0737) |
QRH 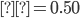 | 0.6816 | 2.9988 | 2.7372 | 659.1450 |
| (0.1253) | (0.7769) | (1.8239) | (1079.8674) |
QRH  | 0.6981 | 4.1405 | 2.8610 | 1077.2027 |
| (0.1140) | (0.6187) | (0.8666) | (60.6781) |
ssGBLUP | 0.7055 | 2.4463 | 3.2641 | 1189.4282 |
| (0.1191) | (0.2204) | (0.4244) | (26.5023) |
Cor(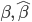 )= correlación entre fenotipos observados y predichos, MSE=cuadrados medios del error,
)= correlación entre fenotipos observados y predichos, MSE=cuadrados medios del error, 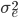 o
o 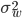 =componentes de varianza asociados al error, DIC=criterio de información de desviación.
=componentes de varianza asociados al error, DIC=criterio de información de desviación.
Cuadro 3: Promedios de la correlación Pearson y la desviación estándar (entre paréntesis) entre los valores fenotípicos observados (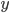 ) y valores fenotípicos predichos (
) y valores fenotípicos predichos (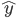 ), cuadrado medio del error, componentes de varianza asociados al error (
), cuadrado medio del error, componentes de varianza asociados al error (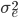 ,
, 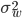 ) y criterio de información de desviación (DIC) para peso al destete
) y criterio de información de desviación (DIC) para peso al destete
Modelo | Cor(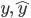 ) ) | MSE | 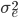 o o 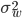
| DIC |
QRM 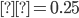 | 0.5661 | 476.5293 | 419.4138 | 1550.5339 |
| (0.2212) | (17.4612) | (23.3216) | (13.9644) |
QRM 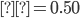 | 0.5695 | 357.7328 | 396.8138 | 1576.8871 |
| (0.2307) | (8.9681) | (47.7433) | (21.5826) |
QRM  | 0.5493 | 175.1298 | 67.9660 | 737.2216 |
| (0.2196) | (47.6181) | (82.0807) | (1150.7340) |
GBLUP | 0.5677 | 294.5807 | 376.7794 | 1583.2355 |
| (0.2377) | (36.6279) | (24.1379) | (16.2187) |
QRH 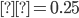 | 0.4816 | 644.1278 | 551.5150 | 1962.1296 |
| (0.0672) | (50.8464) | (64.8091) | (20.9916) |
QRH 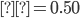 | 0.4797 | 366.5940 | 356.9005 | 1537.7760 |
| (0.0274) | (56.8604) | (238.5303) | (903.3492) |
QRH  | 0.3918 | 216.1753 | 5.9471 | -706.1573 |
| (0.0544) | (53.2417) | (11.7834) | (2034.7757) |
ssGBLUP | 0.4712 | 303.0404 | 421.8316 | 1982.3314 |
| (0.0502) | (37.6933) | (55.2774) | (21.9229) |
Cor(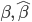 )= correlación entre fenotipos observados y predichos, MSE=cuadrados medios del error,
)= correlación entre fenotipos observados y predichos, MSE=cuadrados medios del error, 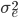 o
o 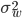 =componentes de varianza asociados al error, DIC=criterio de información de desviación.
=componentes de varianza asociados al error, DIC=criterio de información de desviación.
Cuadro 4: Promedios de la correlación Pearson y la desviación estándar (entre paréntesis) entre los valores fenotípicos observados (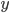 ) y valores fenotípicos predichos (
) y valores fenotípicos predichos (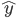 ), cuadrado medio del error, componentes de varianza asociados al error (
), cuadrado medio del error, componentes de varianza asociados al error (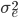 ,
, 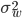 ) y criterio de información de desviación (DIC) para peso al año
) y criterio de información de desviación (DIC) para peso al año
Modelo | Cor(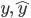 ) ) | MSE | 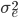 o o 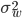
| DIC |
QRM 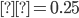 | 0.5421 | 1037.6529 | 953.6807 | 1487.1104 |
| (0.1350) | (175.2648) | (261.8652) | (35.8873) |
QRM 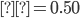 | 0.5341 | 868.3651 | 964.4477 | 1524.0511 |
| (0.1355) | (34.0429) | (113.1832) | (12.4648) |
QRM  | 0.5115 | 938.8244 | 700.7849 | 1284.0829 |
| (0.1290) | (241.2205) | (465.2109) | (402.9787) |
GBLUP | 0.5330 | 725.7579 | 924.8388 | 1526.7596 |
| (0.1389) | (71.3999) | (90.0089) | (11.6346) |
QRH 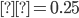 | 0.5306 | 1277.9493 | 1172.2877 | 1850.7122 |
| (0.1411) | (44.0948) | (108.7991) | (17.2025) |
QRH 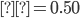 | 0.5098 | 894.4148 | 1061.3157 | 1883.6773 |
| (0.1700) | (35.3996) | (129.4702) | (15.4422) |
QRH  | 0.5027 | 915.1871 | 666.8830 | 1706.4933 |
| (0.1748) | (162.7629) | (413.5046) | (209.8455) |
ssGBLUP | 0.4712 | 778.6416 | 1071.3096 | 1891.9029 |
| (0.0502) | (84.9871) | (128.2878) | (17.5592) |
Cor(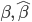 )= correlación entre fenotipos observados y predichos, MSE=cuadrados medios del error,
)= correlación entre fenotipos observados y predichos, MSE=cuadrados medios del error, 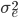 o
o 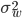 =componentes de varianza asociados al error, DIC=criterio de información de desviación.
=componentes de varianza asociados al error, DIC=criterio de información de desviación.
Datos simulados
Los resultados del ejercicio de simulación donde se compara QR con GBLUP bajo diferentes grados de asimetría y proporciones de datos atípicos se muestran en el Cuadro 5. En la columna 2 se registran las correlaciones entre los efectos de marcador “verdaderos” y los efectos de marcador estimados, las correlaciones obtenidas con QR fueron más altas que las obtenidas con GBLUP. La columna 3 muestra las correlaciones entre las “señales verdaderas” y las estimadas, las correlaciones más altas se obtuvieron con QR. La columna 4 registra la estimación de los componentes de varianza asociados al error y la columna 5 los valores de DIC, los valores más bajos en ambas columnas se obtuvieron con QR  .
.
Cuadro 5: Promedios de la correlación Pearson y desviación estándar (entre paréntesis) entre efectos de marcador “verdaderos y estimados, señales “verdaderas” y estimadas, componentes de varianza asociados al error y valores de DIC para datos simulados con diferentes grados de asimetría y proporción de valores atípicos
Modelo | Cor(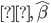 ) ) | Cor(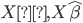 ) ) | 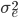 o o 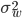
| DIC |
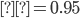 , 5% datos atípicos , 5% datos atípicos
|
QR 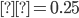 | 0.0784 | 0.4963 | 0.6821 | 620.5455 |
| (0.0034) | (0.0336) | (0.1806) | (49.3305) |
QR 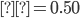 | 0.0766 | 0.4643 | 0.6644 | 665.8219 |
| (0.0042) | (0.0493) | (0.0703) | (16.3032) |
QR  | 0.0606 | 0.4269 | 0.1438 | 290.6870 |
| (0.0132) | (0.0386) | (0.1421) | (148.9695) |
GBLUP | 0.0722 | 0.4910 | 0.7375 | 691.6503 |
| (0.0064) | (0.0398) | (0.0723) | (19.9391) |
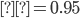 , 10% datos atípicos , 10% datos atípicos
|
QR 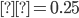 | 0.0614 | 0.4369 | 0.4683 | 407.6496 |
| (0.0183) | (0.0329) | (0.4030) | (330.6304) |
QR 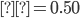 | 0.0728 | 0.4579 | 0.7947 | 706.7931 |
| (0.0045) | (0.0420) | (0.1063) | (20.5797) |
QR  | 0.0574 | 0.4061 | 0.4482 | 381.4644 |
| (0.0092) | (0.0399) | (0.3225) | (474.7138) |
GBLUP | 0.0654 | 0.4556 | 0.8717 | 731.9104 |
| (0.0057) | (0.0314) | (0.0890) | (21.8563) |
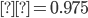 , 5% datos atípicos , 5% datos atípicos
|
QR 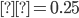 | 0.0773 | 0.4835 | 0.5578 | 582.4254 |
| (0.0087) | (0.0562) | (0.2523) | (83.0548) |
QR 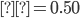 | 0.0771 | 0.4689 | 0.6369 | 662.0337 |
| (0.0074) | (0.0515) | (0.0868) | (23.8018) |
QR  | 0.0598 | 0.4169 | 0.2398 | 219.1691 |
| (0.0128) | (0.0450) | (0.2033) | (444.5060) |
GBLUP | 0.0703 | 0.4804 | 0.7316 | 692.6392 |
| (0.0056) | (0.0333) | (0.0831) | (24.0645) |
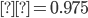 , 10% datos atípicos , 10% datos atípicos
|
QR 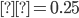 | 0.0731 | 0.4386 | 0.8739 | 677.0858 |
| (0.0081) | (0.0789) | (0.1077) | (23.5472) |
QR 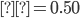 | 0.0734 | 0.4529 | 0.8154 | 711.2935 |
| (0.0078) | (0.0615) | (0.0845) | (14.9809) |
QR  | 0.0541 | 0.3945 | 0.3628 | 385.6030 |
| (0.0056) | (0.0583) | (0.2572) | (393.1935) |
GBLUP | 0.0640 | 0.4491 | 0.8913 | 736.7880 |
| (0.0077) | (0.0517) | (0.0654) | (14.8343) |
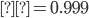 , 5% datos atípicos , 5% datos atípicos
|
QR 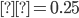 | 0.0615 | 0.5286 | 0.1535 | 205.6973 |
| (0.0144) | (0.0271) | (0.1657) | 277.5807 |
QR 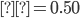 | 0.0741 | 0.5514 | 0.4860 | 614.2282 |
| (0.0037) | (0.0167) | (0.0663) | 15.7647 |
QR  | 0.0467 | 0.4855 | 0.0166 | -271.4761 |
| (0.0112) | (0.0150) | (0.0192) | 288.4509 |
GBLUP | 0.0737 | 0.5428 | 0.5305 | 625.9703 |
| (0.0030) | (0.0121) | (0.0353) | 11.3632 |
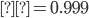 , 10% datos atípicos , 10% datos atípicos
|
QR 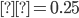 | 0.0768 | 0.4807 | 0.7817 | 650.8593 |
| (0.0080) | (0.0687) | (0.0888) | 22.8417 |
QR 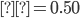 | 0.0696 | 0.4630 | 0.6154 | 511.5287 |
| (0.0148) | (0.0600) | (0.3369) | 412.6645 |
QR  | 0.0507 | 0.3967 | 0.0204 | -160.1660 |
| (0.0031) | (0.0505) | (0.0127) | 213.0462 |
GBLUP | 0.0659 | 0.4649 | 0.7876 | 709.7240 |
| (0.0065) | (0.0418) | (0.0528) | 14.8566 |
Cor(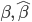 )= correlación entre efectos de marcador “verdaderos” y estimados, Cor(
)= correlación entre efectos de marcador “verdaderos” y estimados, Cor(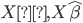 )= correlación entre señales “verdaderas” y estimadas,
)= correlación entre señales “verdaderas” y estimadas, 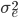 o
o 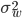 =componentes de varianza asociados al error, DIC=criterio de información de desviación.
=componentes de varianza asociados al error, DIC=criterio de información de desviación.
Discusión
En este estudio se compararon las metodologías de análisis QR con GBLUP y ssGBLUP. Esta comparación se hizo con fenotipos simulados con diferentes grados de asimetría y proporciones de datos atípicos y datos reales de pesos al nacimiento, al destete y al año.
Datos reales
Las correlaciones de fenotipos observados y predichos obtenidos de la validación cruzada con datos reales fueron más altas al usar QRM y QRH en las características de PD y PA. Para PN las correlaciones más altas se obtuvieron con GBLUP y ssGBLUP; sin embargo, en este estudio solo se probaron tres cuantiles 0.25, 0.50 y 0.75, hay evidencia en otros estudios donde QR es mejor que GBLUP como en el caso del trabajo de Nascimento et al(4), quienes compararon QR con modelos como BLASSO, BayesB y RR-BLUP. Estos autores encontraron una ganancia en la capacidad predictiva de QR frente a RR-BLUP de 15.15 %, cabe señalar que matemáticamente RR-es equivalente a GBLUP, además de que los conjuntos de datos usados en este experimento presentaron asimetría.
Los valores del cuadrado medio del error (MSE) miden el promedio de los errores al cuadrado, es decir, la diferencia entre el estimador y lo que se estima, por lo que se prefieren valores bajos; los promedios de MSE de QRM y QRH fueron menores que los obtenidos con GBLUP y ssGBLUP únicamente para PD. El estimador de la varianza residual es un indicativo de que tan bien o mal se ajusta el modelo a los datos observados, se prefieren valores bajos; los componentes de varianza del error menores se obtuvieron con QRM y QRH para las tres características analizadas. Finalmente, el valor de DIC se usa para seleccionar modelos candidatos y al igual que MSE y los componentes de varianza del error se prefieren valores bajos. Los valores de DIC más bajos se obtuvieron con QRM  y QRH
y QRH  , excepto en el escenario de solo marcadores y PN, donde el DIC más bajo se obtuvo con QRM
, excepto en el escenario de solo marcadores y PN, donde el DIC más bajo se obtuvo con QRM 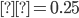 . El cuadrado medio del error, la varianza residual y el DIC son valores que ayuda a escoger el modelo de mejor ajuste. Al examinar estos valores de manera conjunta se observa que QRM y QRH son mejores en algunos de ellos mientras que en otros no, es decir, QR presenta un desempeño igual o superior a GBLUP y ssGBLUP; aunque debe resaltarse que solo se probaron tres cuantiles y que QR presenta ventajas al usarse en datos asimétricos y datos atípicos, para este caso solo había datos atípicos y las distribuciones no fueron asimétricas. Mendes et al(27) compararon QR con el método bayesiano del LASSO (BLASSO), estos autores reportaron un aumento de 6.7 y 20.0 % en la precisión y consideraron los cuantiles 0.15 y 0.45 en la evaluación del rendimiento de la canal y el grosor del tocino, respectivamente, sin embargo, las características evaluadas en su estudio fueron asimétricas.
. El cuadrado medio del error, la varianza residual y el DIC son valores que ayuda a escoger el modelo de mejor ajuste. Al examinar estos valores de manera conjunta se observa que QRM y QRH son mejores en algunos de ellos mientras que en otros no, es decir, QR presenta un desempeño igual o superior a GBLUP y ssGBLUP; aunque debe resaltarse que solo se probaron tres cuantiles y que QR presenta ventajas al usarse en datos asimétricos y datos atípicos, para este caso solo había datos atípicos y las distribuciones no fueron asimétricas. Mendes et al(27) compararon QR con el método bayesiano del LASSO (BLASSO), estos autores reportaron un aumento de 6.7 y 20.0 % en la precisión y consideraron los cuantiles 0.15 y 0.45 en la evaluación del rendimiento de la canal y el grosor del tocino, respectivamente, sin embargo, las características evaluadas en su estudio fueron asimétricas.
En el análisis de datos reales, una limitación del presente estudio es el tamaño de muestra, lo cual puede impactar la variabilidad de los parámetros estimados con los modelos y en consecuencia la variabilidad de las predicciones, sin embargo, todos los modelos se ajustaron utilizando la misma información y por tanto la comparación de la capacidad predictiva de los modelos se considera razonable, lo ideal sería contar con tamaños de muestra grandes, pero por cuestión de limitaciones económicas esto no siempre es posible. Por otro lado, actualmente es muy común utilizar modelos de predicción en el que el número de registros fenotípicos es más pequeño que el número de predictores (SNPS), es decir 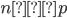 , aun bajo este contexto numerosos estudios han mostrado que los métodos bayesianos proveen herramientas sofisticadas que permiten derivar predicciones razonables siempre y cuando los parámetros de regularización sean seleccionados de manera adecuada por ejemplo utilizando métodos de validación cruzada(28–30).
, aun bajo este contexto numerosos estudios han mostrado que los métodos bayesianos proveen herramientas sofisticadas que permiten derivar predicciones razonables siempre y cuando los parámetros de regularización sean seleccionados de manera adecuada por ejemplo utilizando métodos de validación cruzada(28–30).
Datos simulados
En el experimento de datos simulados las correlaciones entre efectos de marcador “verdadero” y efectos estimados, así como las correlaciones de señales “verdaderas” y estimadas fueron más altas cuando se usó QR comparado con GBLUP. Estos resultados son similares a los obtenidos por otros investigadores(10), quienes simularon datos con tres diferentes coeficientes de asimetría 0.75, 0.95, 0.999 con 5 % y 10 % de datos atípicos y encontraron que las correlaciones obtenidas con QR fueron más altas que las obtenidas con la regresión de cresta bayesiana (BRR), además, este patrón fue más evidente con una mayor asimetría y proporción de datos atípicos. En este estudio se realizaron simulaciones con coeficientes de asimetría de 0.950, 0.975, 0.999 y los cuantiles con los que se obtuvieron correlaciones más altas variaron entre 0.25 y 0.50; la ventaja de QR es que se puede probar diferentes cuantiles obteniendo mejores resultados dependiendo del cuantil utilizado, esta ventaja en la capacidad de predicción de efectos de marcadores y señales ha sido aprovechada por otros investigadores(4), quienes no encontraron ninguna asociación de rasgo usando el modelo tradicional GWAS de SNP único, pero, cuando usaron QR con cuantiles extremos como 0.1 el modelo fue capaz de encontrar hasta 7 SNP asociados con las características estudiadas.
Los coeficientes de varianza del error indican qué tan bien se ajusta el modelo propuesto a los datos estudiados, entre más pequeño sea este valor, el ajuste es mejor, el DIC es otro valor que se usa para comparar modelos candidatos. Los modelos con un DIC más pequeño se prefieren a los modelos con un DIC mayor(24). Los estimadores de varianza residual y los valores de DIC más bajos se obtuvieron con QR  , quizá esto se deba a que en la simulación se usaron coeficientes de asimetría altos 0.950, 0.975, 0.999, por consiguiente, se espera que un cuantil que se ajuste mejor sea el más alto, en este caso 0.75.
, quizá esto se deba a que en la simulación se usaron coeficientes de asimetría altos 0.950, 0.975, 0.999, por consiguiente, se espera que un cuantil que se ajuste mejor sea el más alto, en este caso 0.75.
QR tuvo un desempeño igual o mejor que GBLUP y ssGBLUP para predecir características de crecimiento PN, PD y PA, las ventajas de este método son más notorias cuando los datos están más sesgados y presentan mayor proporción de datos atípicos como en el caso del experimento de simulación.
Conclusiones e implicaciones
El desempeño predictivo de QR tanto sólo con información de marcadores como con marcadores más pedigrí, con el conjunto de datos reales, fue mejor que las metodologías GBLUP y ssGBLUP para PD y PA. Para PN GBLUP y ssGBLUP fueron mejores; sin embargo, solo se utilizaron los cuantiles 0.25, 0.50 y 0.75, y la distribución de PN no fue asimétrica. En el experimento de datos simulados, las correlaciones entre efectos de marcador “verdadero” y efectos estimados, así como las correlaciones de señales “verdaderas” y estimadas fueron más altas cuando se usó QR comparado con GBLUP. Las ventajas de QR fueron más notorias con distribución asimétrica de los fenotipos y con mayor proporción de datos atípicos, como fue el caso del conjunto de datos simulados.
Agradecimientos
Al Consejo Nacional de Ciencia y Tecnología, México, por el apoyo financiero para el primer autor durante sus estudios de doctorado. Los autores también agradecen a la Asociación Mexicana de Criadores de Ganado Suizo de Registro por permitir el uso de sus bases de datos, a los criadores cooperantes por su amable cooperación en este estudio.
Conflictos de interés
Los autores declaran que no existen conflictos de interés.
- Koenker R, Bassett G. Regression quantiles. Econometrica 1978;46(1):33. https://doi.org/10.2307/1913643.
- Briollais L, Durrieu G. Application of quantile regression to recent genetic and -omic studies. Hum Genet 2014;133(8):951–966. https://doi.org/10.1007/s00439-014-1440-6.
- Wang L, Wu Y, Li R. Quantile regression for analyzing heterogeneity in ultra-high dimension. J Am Stat Assoc 2012;107(497):214-222. https://doi.org/10.1080/01621459.2012.656014.
- Nascimento M, Nascimento ACC, Silva FF, Barili LD, Do Vale NM, Carneiro JE, Cruz CD, Carneiro PCS, Serão NVL. Quantile regression for genome-wide association study of flowering time-related traits in common bean. PLoS One 2018;13(1):1-14. https://doi.org/10.1371/journal.pone.0190303.
- Fisher E, Schweiger R, Rosset S. Efficient construction of test inversion confidence intervals using quantile regression. J Comput Graph Stat 2016;29:140-148, http://arxiv.org/abs/1612.02300.
- Logan JAR, Petrill SA, Hart SA, Schatschneider C, Thompson LA, Deater-Deckard K, de Thorne LS, Bartlett C. Heritability across the distribution: An application of quantile regression. Behav Genet 2012;42(2):256–267. https://doi.org/10.1007/s10519-011-9497-7.
- Vinciotti V, Yu K. M-quantile regression analysis of temporal gene expression data. Stat Appl Genet Mol Biol 2009;8(1). https://doi.org/10.2202/1544-6115.1452.
- Gianola D, Cecchinato A, Naya H, Schön CC. Prediction of complex traits: Robust alternatives to best linear unbiased prediction. Front Genet 2018;9:195. https://doi.org/10.3389/fgene.2018.00195.
- Oliveira GF, Nascimento ACC, Nascimento M, Sant’Anna IdeC, Romero JV, Azevedo CF, Bhering LL, Moura ETC. Quantile regression in genomic selection for oligogenic traits in autogamous plants: A simulation study. PLoS One 2021;16(1):1-12. https://doi.org/10.1371/journal.pone.0243666.
- Pérez-Rodríguez P, Montesinos-López OA, Montesinos-López A, Crossa J. Bayesian regularized quantile regression: A robust alternative for genome-based prediction of skewed data. Crop J 2020;8(5):713-722. https://doi.org/10.1016/j.cj.2020.04.009.
- Nascimento M, e Silva FF, de Resende MDV, Cruz CD, Nascimento ACC, Viana JMS, Azebedo CF, Barroso LMA. Regularized quantile regression applied to genome-enabled prediction of quantitative traits. Genet Mol Res 2017;16(1). https://doi.org/10.4238/GMR16019538.
- Barroso LMA, Nascimento M, Nascimento ACC, Silva FF, Serão NVL, Cruz, CD, et al. Regularized quantile regression for SNP marker estimation of pig growth curves, J. Anim Sci Biotechnol 2017;8:59. https://doi.org/10.1186/s40104-017-0187-z.
- Nascimento AC, Nascimento M, Azevedo C, Silva F, Barili L, Vale N, et al. Quantile regression applied to genome-enabled prediction of traits related to flowering time in the common bean. Agronomy 2019;9(12):1-11. https://doi.org/10.3390/agronomy9120796.
- López-Cruz M, Crossa J, Bonnett D, Dreisigacker S, Poland J, Jannink JL, Singh RP, Autrique E, de los Campos G. Increased prediction accuracy in wheat breeding trials using a Marker × Environment interaction genomic selection model. G3 Genes Genom Genet 2015;5(4):569–582. https://doi.org/10.1534/g3.114.016097.
- Pérez-Rodríguez P, Crossa J, Rutkoski J, Poland J, Singh R, Legarra A, et al. Single-step genomic and Pedigree Genotype × Environment interaction models for predicting wheat lines in international environments. Plant Genome 2017;10(2). https://doi.org/10.3835/plantgenome2016.09.0089.
- Christensen O, Lund M. Genomic relationship matrix when some animals are not genotyped. Genet Sel Evol 2010;42(2):1-8. http://www.gsejournal.org/content/42/1/2.
- Aguilar I, Misztal I, Johnson DL, Legarra A, Tsuruta S, Lawlor TJ. Hot topic: A unified approach to utilize phenotypic, full pedigree, and genomic information for genetic evaluation of Holstein final score. J Dairy Sci 2010;93(2):743-752. https://doi.org/10.3168/jds.2009-2730.
- Crossa J, Pérez P, de los Campos G, Mahuku G, Dreisigacker S, Magorokosho C. Genomic selection and prediction in plant breeding. J Crop Improv 2011;25(3):239-261. https://doi.org/10.1080/15427528.2011.558767.
- Arnold BC, Beaver RJ. Hidden truncation models. Shankhya. Indian J Stat 2000;62(1):23–35. http://www.jstor.org/stable/25051286. Accessed Jul 6, 2022.
- Montesinos-López A, Montesinos-López OA, Villa-Diharce ER, Gianola D, Crossa J. A robust Bayesian genome-based median regression model. Theor Appl Genet 2019;132(5):1587-1606. https://doi.org/10.1007/s00122-019-03303-6.
- Pérez-Rodríguez P, Villaseñor-Alva JA. On testing the skew normal hypothesis. J Stat Plan Inference 2010;140(11):3148-3159. https://doi.org/10.1016/j.jspi.2010.04.013.
- Pérez-Rodríguez P, Acosta-Pech R, Pérez-Elizalde S, Cruz CV, Espinosa JS, Crossa J. A Bayesian genomic regression model with skew normal random errors. G3 Genes|Genom|Genet 2018;8(5):1771–1785. https://doi.org/10.1534/g3.117.300406.
- Domínguez-Viveros J. Parámetros genéticos en la varianza residual de variables de comportamiento en toros de lidia. Arch Zoot 2020;69(267):354–358. https://doi.org/10.21071/az.v69i267.5354.
- Spiegelhalter DJ, Best NG, Carlin BP, van der Linde A. Bayesian measures of model complexity and fit. J R Stat Soc: Series B Stat Methodol 2002;64(4):583–639. https://doi.org/10.1111/1467-9868.00353.
- R Core Team. R: A language and environment for statistical computing. R Foundation for statistical computing 2021. Vienna, Austria. https://www.R-project.org/.
- Pérez P, de los Campos G. Genome-wide regression and prediction with the BGLR statistical package. Genetics 2014;198(2):483-495. https://doi.org/10.1534/genetics.114.164442.
- Mendes dos Santos P, Nascimento ACC, Nascimento M, Fonseca e Silva F, Azevedo CF, Mota RR, et al. Use of regularized quantile regression to predict the genetic merit of pigs for asymmetric carcass traits. Pesqui Agropecu Bras 2018;53(9):1011–1017. https://doi.org/10.1590/S0100-204X2018000900004.
- Gianola D. Priors in whole-genome regression: The Bayesian alphabet returns. Genetics 2013;194(3):573-596. https://doi.org/10.1534/genetics.113.151753.
- de los Campos G, Hickey JM, Pong-Wong R, Daetwyler HD, Calus MPL. Whole-genome regression and prediction methods applied to plant and animal breeding. Genetics 2013;193(2):327-345. https://doi.org/10.1534/genetics.112.143313.
- Ferragina A, de los Campos G, Vazquez AI, Cecchinato A, Bittante G. Bayesian regression models outperform partial least squares methods for predicting milk components and technological properties using infrared spectral data. J Dairy Sci 2015;98(11):8133-8151. https://doi.org/10.3168/jds.2014-9143.
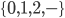 , donde “
, donde “ ” denota un valor faltante. Para los 12,835 marcadores en común de los dos chips, los valores faltantes se imputaron al azar, generando muestras de la distribución
” denota un valor faltante. Para los 12,835 marcadores en común de los dos chips, los valores faltantes se imputaron al azar, generando muestras de la distribución 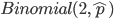 donde
donde 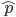 es la frecuencia del alelo mayor, calculado a partir de los genotipos de marcadores observados. Se eliminaron los marcadores monomórficos o aquellos con frecuencia de alelos menores (MAF) menor que 0.04. Después del control de calidad, 9,628 de los 12,835 SNP en común entre los dos chips estuvieron disponibles para análisis adicionales.
es la frecuencia del alelo mayor, calculado a partir de los genotipos de marcadores observados. Se eliminaron los marcadores monomórficos o aquellos con frecuencia de alelos menores (MAF) menor que 0.04. Después del control de calidad, 9,628 de los 12,835 SNP en común entre los dos chips estuvieron disponibles para análisis adicionales.