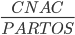 (PARTOS: número de partos totales en la VR).
(PARTOS: número de partos totales en la VR). https://doi.org/10.22319/rmcp.v14i1.6073
Artículo
Efecto de la consanguinidad y selección sobre los componentes de un índice productivo, en ratones bajo apareamiento estrecho
Dulce Janet Hernández López a
Raúl Ulloa Arvizu a
Carlos Gustavo Vázquez Peláez a
Graciela Guadalupe Tapia Pérez a*
a Universidad Nacional Autónoma de México, Departamento de Genética y Bioestadística de la Facultad de Medicina Veterinaria y Zootecnia. Ciudad de México, México.
*Autor de correspondencia: tapiadoctora@gmail.com
Resumen:
Con objeto de examinar la influencia de la depresión endogámica en algunas características productivas del ratón de laboratorio, se reanalizaron 871 registros provenientes de 20 generaciones en una línea con cruza consanguínea estrecha con selección a un índice productivo (CNHS) comparando con una línea sin selección, con cruzamiento endogámico (n= 135). Se calcularon los coeficientes de endogamia (F) para cada generación. En todos los componentes del índice (vida reproductiva, estros postparto fértiles y tamaño de camada), se compararon las dos líneas, en las 15 generaciones disponibles de la no seleccionada, por método de mínimos cuadrados, agrupando cada cinco generaciones. La seleccionada, se analizó en las 20 generaciones, para diferencias intergeneracionales con el mismo método. La depresión endogámica se estimó en todas las generaciones con una regresión lineal de consanguinidad (expresada en 10 %) en todos los componentes. Se observó diferencia significativa (P<0.01) entre líneas en las variables analizadas. Los estros post parto fértiles de la línea seleccionada se mantuvieron constantes, hubo un decremento de 0.331 en la no seleccionada (P<0.01). El índice productivo se mantuvo estable (aumentó 0.071) en la seleccionada, en la no seleccionada disminuyó (0.39) hasta desaparecer (G15). La depresión endogámica impactó en la vida reproductiva de ambas, decreció 4.741 días en la seleccionada vs. 7.718 días en la no seleccionada (P<0.01). En la no seleccionada afectó en mortalidad al destete y ciclo estral, la selección al índice contrarrestó ese impacto, probablemente debido a la selección de genes que favorecen el desarrollo gonadal de los ratones.
Palabras clave: Ratones, Selección, Vida reproductiva, Número de estros, Depresión endogámica.
Recibido: 05/10/2021
Aceptado: 16/08/2022
Introducción
En la actualidad se han desarrollado un gran número de líneas de ratones genéticamente diferentes, que tienen propósitos de investigación particulares. Las líneas consanguíneas fueron el prototipo de las líneas genéticamente estandarizadas, que permitieron elaborar experimentos eliminando la variabilidad de origen genético. Aunque la genómica, proporciona a los laboratorios las herramientas necesarias para producir ratones con las características que demanda la investigació, cuando se ha fijado una característica, se necesita de un riguroso proceso de selección y apareamiento dirigido para mantener la viabilidad de la línea, que generalmente conlleva a depresión endogámica(1,2).
La base genética de este fenómeno está relacionada con tres hipótesis a saber, dominancia parcial (mayor expresión de alelos recesivos deletéreos, sobredominancia (superioridad de heterocigotos sobre ambos tipos de homocigotos) y epistasis (mayor probabilidad de combinaciones genéticas favorables para heterocigotos)(3).
Los criadores de animales domésticos de razas puras utilizan la endogamia para fijar rasgos genéticos deseables dentro de una población o para intentar eliminar los rasgos deletéreos, la depresión por endogamia puede afectar los ingresos económicos de los criadores(4). Los estudios en ratones al ofrecer un mayor número de generaciones en menor tiempo ayudan a comprender la depresión endogámica, en poblaciones donde se busca seleccionar alguna característica.
En el ratón, se observó una reducción de 7.2 % en el tamaño de camada por cada 10 % de incremento en consanguinidad, bajo apareamiento consecutivo de hermanos completos sin selección(2) y, con el mismo incremento de consanguinidad en cruzas entre medios hermanos sin selección, el decremento fue de 6.22% en el tamaño de camada(5).
La depresión fue menos severa en líneas bajo selección dirigida que en líneas sin selección(6), esto fue observado cuando se seleccionó para tamaño de camada en ratones, encontrando que la reducción en la aptitud reproductiva fue significativamente menor en las líneas consanguíneas bajo selección, comparada con la de las líneas consanguíneas sin selección; lo anterior se explica porque gracias a la selección, hay un aumento de los genes relacionados con la mejor aptitud reproductiva, que contrarresta la depresión endogámica causante de la reducción de dicha aptitud(7). Se ha visto que el comportamiento de una línea consanguínea seleccionada para tamaño de camada es similar al de una no consanguínea, seleccionada para la misma característica. En un estudio, la consanguinidad permitió superar el límite de selección para tamaño de camada grande, cuando en la línea seleccionada se accedió a un cruzamiento consanguíneo(8).
El número de crías destetadas por hembra por semana (CDHS), es un índice productivo que se mide durante y al término de la vida reproductiva en cada pareja de ratones. Se utiliza en las colonias fundadoras de algunas compañías de animales de laboratorio(9,10). A pesar de que se recomienda seleccionar a los ratones provenientes de familias con más alto CDHS para mantener líneas de laboratorio, mediante cruzamiento consanguíneo estrecho con características fijadas(11), en la literatura hay poca información del efecto de esa selección en ratones endogámicos acerca de las variables incluidas en él.
Por lo que el objetivo de este estudio fue evaluar el efecto de la endogamia sobre los componentes de un índice productivo, en el modelo animal de ratón de laboratorio, durante 20 generaciones de selección con cruza consanguínea estrecha, así como evaluar si la selección puede afectarse en su avance, por el efecto de la depresión endogámica, en las características que lo constituyen.
Material y métodos
El presente trabajo es un estudio retrospectivo, transversal, comparativo y observacional. Se reanalizaron 871 registros de un bioterio, que fueron tomados durante cinco años en ratones con selección continua y cruza consanguínea estrecha (hermano con hermana) donde hubo cinco líneas seleccionadas a un índice productivo: (CDHS) en 20 generaciones (n= 871). Los datos, recabados entre 1989-1994, se habían analizado con el objetivo de la obtención de heredabilidad realizada, para el índice productivo, una descripción detallada se puede ver en Tapia-Pérez(12).
Para este estudio se añadió una línea de la misma cepa contemporánea, del propio bioterio (n= 135), con cruza consanguínea estrecha sin selección hasta la generación 15; después de esta generación las parejas dejaron de ser fértiles.
Los animales se alojaron en jaulas de policarbonato tipo caja de zapatos, la cual ofrece un área de 375 cm, con tapa tipo Cambridge de acero inoxidable y filtro de poliéster rígido tipo Kraft; se proporcionó alimento ad libitum, agua potable filtrada por ósmosis reversible acidificada a un pH de 2.5. El aire se filtraba, y se mantuvo una temperatura de 18 a 26 °C. La identificación de los animales fue individual, primero, por medio de muescas en las orejas, y los registros por medio de tarjetas en cada jaula. Estas tarjetas después se resumieron en carpetas de registro llamadas AER (Análisis de Eficiencia Reproductiva), a partir de las cuales se calculó el índice CDHS cada tres generaciones, o cuando se detectaron líneas divergentes (esto podría darse en la cuarta generación en cada línea seleccionada), para obtener y seleccionar la sublínea con el promedio más alto, que además contara con crías de tercer parto, con cuando menos dos hembras y dos machos para cría. Cabe hacer notar que aquellas parejas estériles (no se registró gestación), infértiles (se registró gestación, pero no parto) o que canibalizaron a sus crías tuvieron un valor CDHS de cero, que fue incluido para obtener el promedio dado que se consideran resultado de la depresión endogámica. En cuanto a la línea no seleccionada, el manejo fue similar; esta línea fue la única que continuó activa de las cinco que comenzaron al mismo tiempo de las seleccionadas, las otras cuatro se perdieron en la segunda generación. El manejo reproductivo en ambas líneas fue bajo un método monogámico intensivo, es decir, se colocaron en la misma jaula un macho con una hembra y permanecieron juntos toda su vida reproductiva (165 ± 3.6 días). Los apareamientos se iniciaron cuando los animales alcanzaron la madurez sexual (8-10 semanas).
Las parejas seleccionadas para reproducción se formaron aleatoriamente, una hembra y un macho hermanos completos, provenientes del tercer parto de sus padres, tanto en la generación de selección (3 o 4) como en las anteriores. En cada generación se mantuvo un promedio de ocho parejas por línea.
Descripción de variables
Las variables que se analizaron fueron:
VR: vida reproductiva, medida como los días totales en reproducción.
EPPF: número total de estros post parto fértiles en la VR. Se considera un estro post parto fértil cuando la hembra tiene un parto en el primer estro, dentro de 35 o menos días del anterior (ya que se asume un período de gestación de 21 días, con una implantación dentro de 5 días, si ésta se lleva a cabo cuando la madre aún está lactando una camada previa, se puede dar en 14 días máximo, así: 21 + 14 =35)(9).
CNAC: número total de crías nacidas en la VR.
CDEST: número total de crías destetadas en la VR.
CNPP: crías nacidas por parto = 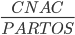 (PARTOS: número de partos totales en la VR).
(PARTOS: número de partos totales en la VR).
CDPP: crías destetadas por parto = 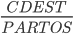 .
.
CDHS: (Índice de eficiencia productiva) es el número de crías destetadas por hembra por semana = 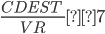 .
.
Porcentaje de mortalidad al destete = 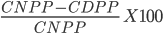 .
.
Análisis estadístico
Para el análisis estadístico, la línea seleccionada corresponde al conjunto de las cinco líneas con selección a CDHS dividida en: línea seleccionada durante 15 generaciones (S15G) n= 733 , la misma línea seleccionada en las 20 generaciones (S20G) n= 871 y línea no seleccionada, que permaneció hasta la generación 15 (NS15G) n=135, ya que en ésta quedaron cinco parejas, de las cuales solo una llegó al tercer parto y las crías no eran suficientes para hacer las cruzas.
Se agrupó la información de cinco generaciones en cada línea, para todas las variables, de manera que, los datos analizados en cada nivel corresponden a los de cinco generaciones sucesivas. Se agruparon de esta forma para observar el resultado de la selección en la quinta generación, debido a que, como se explicó, se seleccionaba mínimo cada tres generaciones o cuando se detectaban líneas divergentes, que podría darse hasta en cuatro generaciones. El nivel 1 contiene la suma de las generaciones de 1 a 5; el nivel 2, de 6 a 10 y el nivel 3 de 11 a 15 de S15G y NS15G, mientras que el nivel 4 únicamente corresponde a S20G, en las generaciones de 16 a 20.
Se realizaron pruebas de normalidad de las variables mencionadas por el método de Kolmogorov-Smirnov.
El modelo lineal general utilizado para comparar S15G y NS15G fue (Modelo 1):
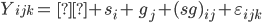
Donde
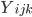 es la suma de cinco generaciones sucesivas de las parejas, para cada variable cuantitativa;
es la suma de cinco generaciones sucesivas de las parejas, para cada variable cuantitativa;
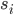 es el efecto del i-ésimo grupo de selección (i=1,2);
es el efecto del i-ésimo grupo de selección (i=1,2);
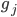 es el efecto de la generación agrupada cada cinco generaciones (j=1,2,3);
es el efecto de la generación agrupada cada cinco generaciones (j=1,2,3);
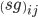 el efecto de la interacción entre el grupo de selección y la generación agrupada;
el efecto de la interacción entre el grupo de selección y la generación agrupada; 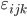 el error aleatorio (
el error aleatorio (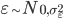 ).
).
El modelo de análisis para S20G (Modelo 2), sólo incluyó el efecto de la generación agrupada gi, (i=1,2,3,4):
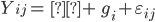
Donde
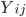 es la suma de 5 generaciones sucesivas de las parejas, para cada variable cuantitativa, en la iésima generación;
es la suma de 5 generaciones sucesivas de las parejas, para cada variable cuantitativa, en la iésima generación;
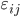 el error aleatorio (
el error aleatorio (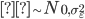 ).
).
Ambos modelos se analizaron por el método de cuadrados mínimos. El coeficiente de consanguinidad se calculó para cada animal, con el Programa Computacional Pedigree Viewer©, desarrollado por Brian Kinghorn(13), que utiliza el método desarrollado por Wright (1922)(14).
La depresión por endogamia media (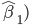 y su error estándar (e.e.), de CDHS se estimaron por el método de cuadrados mínimos con los coeficientes de consanguinidad (Fi) de cada generación, en unidades de 10 % para las líneas S15 y NS15 (i=1,2,3, …,15) y S20 (i=1,2,3, ..., 20), con el siguiente modelo de regresión lineal simple (Modelo 3).
y su error estándar (e.e.), de CDHS se estimaron por el método de cuadrados mínimos con los coeficientes de consanguinidad (Fi) de cada generación, en unidades de 10 % para las líneas S15 y NS15 (i=1,2,3, …,15) y S20 (i=1,2,3, ..., 20), con el siguiente modelo de regresión lineal simple (Modelo 3).
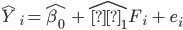
Donde
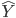 i Es el promedio de cada componente del índice en la generación i-ésima;
i Es el promedio de cada componente del índice en la generación i-ésima;
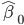 es la estimación del intercepto;
es la estimación del intercepto;
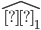 es la depresión por endogamia media, Fi es el coeficiente de consanguinidad (10%);
es la depresión por endogamia media, Fi es el coeficiente de consanguinidad (10%);
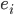 es el error aleatorio (
es el error aleatorio (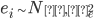 ).
).
Dado que el aumento de la consanguinidad se consideró en unidades de 10 %, las depresiones endogámicas se relacionaron con esta medida, que se eligió para poder comparar los resultados de este estudio con otros artículos de ratones donde se calcula de esa forma.
Los modelos se analizaron con el paquete estadístico, IBM SPSS Versión 22(15), los porcentajes de mortalidad al destete, de S15G y NS15G, en cada una de las tres generaciones agrupadas, se analizaron mediante la prueba de Ji-cuadrada, con el programa MedCalc ® en línea(16). Se consideró el valor P≤0.05 como significativo y P≤0.01 como altamente significativo.
Resultados
Modelos lineales
Modelo 1
Se observó un efecto de grupo de selección (si) altamente significativo (P<0.01) en todas las variables. En cuanto a la interacción (sg)ij, en las variables EPPF, CNAC, CDEST y CDHS fue altamente significativo (P<0.01), en VR y CDPP el efecto de la interacción resultó significativo (P<0.05), lo que no sucedió en la variable CNPP (P>0.05) (Cuadro 1).
Cuadro 1: Medias mínimo cuadráticas (M) y errores estándar (EE) de la interacción (sg)ij, en cinco generaciones agrupadas, para S15G y NS15G
LÍNEA | GA | F(%) | EST | EPPF | VR | CNAC | CDEST | CNPP | CDPP | CDHS |
S15GA | 1 a 5 | 50 | M | 1.74a | 165.2a | 20.35a | 17.77a | 4.77a | 4.16a | 0.66a |
(n=733) | EE | 1.58 | 3.52 | 8.16 | 7.65 | 1.59 | 0.54 | 0.029 | ||
6 a 10 | 82.6 | M | 2.18a | 129.5b | 19.01a | 16.09a | 4.62a | 3.92a | 0.598a | |
EE | 1.62 | 3.44 | 10.33 | 10.29 | 2. 06 | 2.27 | 0.029 | |||
11 a 15 | 93.9 | M | 2.1a | 146.2a | 21.06a | 19.31a | 5.01a | 4.60a | 0.687a | |
EE | 1.5 | 3.32 | 9.78 | 9.61 | 1.92 | 2.00 | 0.028 | |||
NS15GB | 1 a 5 | 50 | M | 2.65a | 146.7a | 17.16a | 13.63a | 3.76a | 2.96a | 0.59a |
(n=135) | EE | 1.63 | 6.64 | 7.52 | 7.164 | 1.26 | 1.49 | 0.055 | ||
6 a 10 | 82.6 | M | 1.32b | 99.67b | 7.76b | 4.64b | 3.04a | 1.45b | 0.12b | |
EE | 1.41 | 7.74 | 4.37 | 4.85 | 1.45 | 1.70 | 0.064 | |||
11 a 15 | 93.9 | M | 0.3c | 99.75b | 7.85b | 6.15b | 3.87a | 2.88a | 0.20b | |
EE | 0.66 | 8.36 | 5.26 | 5.59 | 1.57 | 2.10 | 0.069 | |||
P(SG) | <0.01 | 0.047 | <0.01 | 0.01 | 0.482 | 0.048 | <0.01 |
GA= generaciones agrupadas. F= consanguinidad obtenida en 5 generaciones. EST= estadístico. EPPF= número de estros post parto fértiles. VR= vida reproductiva de la pareja en días. CNAC= número total de crías nacidas en la VR. CDEST= número total de crías destetadas en la VR. CNPP= crías nacidas por parto. CDPP= crías destetadas por parto. CDHS= número de crías destetadas por hembra por semana.
A,B literales diferentes denotan diferencias altamente significativas entre los grupos de selección si (P<0.01).
P(SG) es la significancia calculada por el modelo,
.abc literales distintas denotan diferencias significativas (P<0.05) intergeneracionales, dentro de línea.
Las medias de los EPPF de S15G subieron 0.44 estros y se mantuvieron (P>0.05), mientras que las medias de NS15G decrecieron 2.4 estros promedio, de 1 a 5 hasta las generaciones 11 a 15 (P<0.05) (Cuadro 1 y Figura 1). La vida reproductiva (VR), disminuyó en S15G, en las generaciones 6 a 10 casi 36 días (P<0.05), después se recuperó, aunque no al nivel de las primeras cinco generaciones, mientras que en NS15G se mantuvo 47 días más baja que en las primeras cinco generaciones (P<0.05). Tanto el número de crías nacidas y destetadas, en el total de vida reproductiva de las parejas, los valores más bajos se observaron en las generaciones 6 a 10 en ambas líneas; sin embargo, sólo NS15G mostró diferencias significativas (P<0.05) con una disminución de 9.4 y 9 crías respectivamente. Tanto CNPP como CDPP, se obtuvieron como promedio de todos los partos en la vida reproductiva de la hembra, agrupados cada cinco generaciones y fueron más bajos en NS15G, el pico más bajo se observó entre las generaciones 6 a 10 en ambas, pero sólo fue significativo en NS15G en las CDPP con una disminución de 0.7 crías destetadas (P<0.05) (Cuadro1).
Figura 1: Medias y desviaciones estándar de número de estros post parto fértiles (EPPF) con la consanguinidad agrupada cada cinco generaciones (1 a 5, 6 a 10 y 11 a 15)
S15G y NS15G, son las líneas con selección y sin selección para CDHS en las 15 generaciones de la última.
El índice (CDHS), se mantuvo estable a través de todas las generaciones acumuladas (P>0.05) en S15G, mientras que NS15G tiene una caída abrupta de las generaciones 1-5 a 6-10 (-0.47 crías) (P<0.05), con una leve recuperación en las siguientes cinco generaciones agrupadas (0.08 crías) (P<0.05); después se pierde debido a una gran mortalidad (Cuadro 1 y Figura 2).
Figura 2: Medias y desviaciones estándar del número de crías destetadas por hembra por semana (índice de eficiencia productiva) (CDHS) con la consanguinidad agrupada cada 5 generaciones (1 a 5, 6 a 10 y 11 a 15)
S15G y NS15G, son las líneas con selección y sin selección para CDHS en las 15 generaciones de la última.
Modelo 2
Cuando se analizó S20G, se observó el pico más bajo en las generaciones 6 a 10 (P<0.05), en casi todos los componentes, mientras que EPPF y el índice CDHS no mostraron cambios significativos intergeneracionales (P>0.05) (Cuadro 2).
Cuadro 2: Medias mínimo cuadráticas (M) y errores estándar (EE) de cinco generaciones agrupadas, para S20G
GA | F(%) | EST | EPPF | VR | CNAC | CDEST | CNPP | CDPP | CDHS |
1 a 5 | 50 | M | 1.59a | 165.2a | 18.64a | 16.29a | 4.77a | 4.17a | 0.616a |
EE | 0.12 | 3.58 | 0.81 | 0.76 | 0.14 | 0.14 | 0.031 | ||
6 a 10 | 82.6 | M | 1.79a | 130.0b | 15.26b | 12.92b | 4.63a | 3.92b | 0.598a |
EE | 0.12 | 3.49 | 0.78 | 0.74 | 0.14 | 0.15 | 0.030 | ||
11 a 15 | 93.9 | M | 1.78a | 146.7c | 17.64ab | 16.17a | 5.01a | 4.59a | 0.687a |
EE | 0.11 | 3.38 | 0.76 | 0.71 | 0.13 | 0.14 | 0.029 | ||
16 a 20 | 97.4 | M | 1.80a | 133.9bc | 17.26ab | 15.17ab | 5.18b | 4.49ab | 0.675a |
EE | 0.11 | 3.39 | 0.76 | 0.72 | 0.14 | 0.14 | 0.029 |
GA= generaciones agrupadas. F= consanguinidad agrupada en 5 generaciones. EST= estadístico. EPPF= número de estros post parto fértiles. VR= vida reproductiva de la pareja en días. CNAC= número total de crías nacidas. CDEST= número total de crías destetadas en la VR. CNPP= Crías nacidas por parto. CDPP= crías destetadas por parto. CDHS= número de crías destetadas por hembra por semana.
abc Literales distintas denotan diferencias significativas (P<0.05) intergeneracionales-
Modelo 3. Depresión endogámica
Hubo un efecto altamente significativo (P<0.01) de depresión endogámica en S15G, en la VR, CNPP y CDPP, mientras que CNAC, CDEST, EPPF y CDHS no fue significativo (P>0.05); en NS15G, las características EPPF, VR, CNAC, CDEST y CDHS mostraron un efecto altamente significativo (P<0.01) de la depresión endogámica, sin embargo, en CNPP y CDPP, fue no significativo (P>0.05) (Cuadro 3).
Cuadro 3: Coeficientes de regresión no estandarizados medios (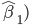 y su error estándar (EE), de los componentes de CNHS y del índice mismo, en NS15G, S15G y S20G, sobre el coeficiente de consanguinidad
y su error estándar (EE), de los componentes de CNHS y del índice mismo, en NS15G, S15G y S20G, sobre el coeficiente de consanguinidad
Línea | Estadístico | EPPF | VR | CNAC | CDES | CNPP | CDPP | CDHS |
S15G (n=733) |
EE | 0.052 0.030 | 4.741 1.276 | -0.454 0.256 | -0.332 0.256 | -0.466 0.084 | -0.578 0.086 | -0.001 0.007 |
NS15G (n=135) |
EE | 0.331 0.066 | 7.718 2.507 | -1.705 0.341 | -1.325 0.326 | 0.028 0.087 | -0.010 0.109 | -0.062 0.015 |
S20G (n= 871) |
EE | 0.047 0.026 | -4.76 1.09 | -0.361 0.213 | -0.268 0.210 | 0.02 0.036 | 0.026 0.036 | -0.001 0.007 |
S15G= línea con 15 generaciones de selección. NS15= línea sin selección durante 15 generaciones. S20G= misma línea con selección durante 20 generaciones. EPPF: Número de estros post parto fértiles. VR= vida reproductiva en días. CNAC= crías nacidas en el total de la vida reproductiva. CDES= crías destetadas en el total de la vida reproductiva. CNPP= crías nacidas por parto. CDPP= crías destetadas por parto. CDHS= número de crías destetadas por hembra por semana.
Los coeficientes de regresión en negritas fueron altamente significativos (P<0.01).
La vida reproductiva disminuyó en ambas líneas por cada 10 % de consanguinidad, NS15G 7.718 días vs 4.741 en S15G (P<0.01). En CNAC y CDEST solo hubo efecto de depresión endogámica en NS15G (-1.705 y -1.325 crías respectivamente) (P<0.01). CNPP y CDPP se obtuvieron como un promedio de los partos en la VR de cada pareja, en estas variables la depresión endogámica afectó solo a S15G (-0.466 y -0.578 crías respectivamente) (P<0.01) mientras que NS15G hubo una aparente estabilidad (P>0.05), debido a que el número de partos promedio en las generaciones acumuladas fue disminuyendo (4.2, 1.7 y 1.1), en las generaciones acumuladas 1 a 5, 6 a 10 y 11 a 15 respectivamente, mientras que en la seleccionada se mantuvieron casi sin cambios (3.9, 3.8 y 3.6) . El índice CDHS no mostró depresión endogámica en S15G (P>0.05) lo contrario ocurrió en NS15G (P<0.01). Por otro lado, S20G mostró depresión endogámica altamente significativa de –4.76 días en la VR por cada 10 % de aumento en la consanguinidad (P<0.01) (Cuadro 3).
Mortalidad al destete
El porcentaje de mortalidad por parto al destete, con respecto a CNPP tuvo un comportamiento similar en ambas líneas, con un pico máximo en las generaciones 6 a 10, sin embargo, la línea no seleccionada mantuvo una mortalidad más alta que la seleccionada (52.30 %) teniendo una diferencia con la seleccionada del 36.8 (P<0.01) (Figura 3). La sobrevivencia por parto (100 – % mortalidad por parto) entonces, decae en las generaciones de las 6 a la 10 en la línea no seleccionada 31 % (78.47 - 47.7 %) y la seleccionada 2.37 % (87.22 - 84.85 %) manteniéndose más alta en la línea con selección.
Figura 3: Mortalidad por parto al destete, en porcentaje respecto a las CNPP, con la consanguinidad agrupada en 5 generaciones
S15G y NS15G, son las líneas con selección y sin selección para CDHS en las 15 generaciones de la última.
Discusión
En la literatura se encuentran pocos trabajos de selección sobre fertilidad a largo plazo y su efecto en la depresión endogámica en ratones; en un estudio de selección de tamaño de camada al primer parto que comenzó en 1972, evitando los cruzamientos entre hermanos completos, medios hermanos o primos, después de 124 generaciones de selección se encontró una consanguinidad de 0.64 en una de sus líneas, lo que llevó a ésta a una mayor depresión endogámica (-0.39), con un menor número de crías vivas en el primer parto, por cada 10 % de consanguinidad(17). Los resultados del presente trabajo coinciden con aquél, cuando se obtuvo el promedio de las crías nacidas vivas por parto (CNPP) en la línea con selección, el efecto de la depresión endogámica fue de -0.466, un poco más alto que aquél, debido a que en ese trabajo sólo se midió el primer parto. El número de crías nacidas por parto, en el grupo NS15G, mostró un efecto no significativo de la consanguinidad (P>0.05) (Cuadro 3), lo que pareciera contrario a lo esperado; la explicación es que el número de partos en la vida reproductiva de las parejas fue disminuyendo conforme aumentaban las generaciones y al promediar el tamaño de camada al parto y al destete (de toda la VR de cada pareja) con estos, parece que se hubiera mantenido constante (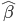 1= 0.028). Otra diferencia es que la consanguinidad en dicho estudio es menor, aunque el número de generaciones de selección es de 124, durante ese tiempo (1972 a 2007), hubo varios cambios en la dirección de la selección en ese trabajo, pero se trató de evitar las cruzas endogámicas(17). No se encontró en la literatura, algún trabajo con selección a tamaño de camada de toda la vida reproductiva con cruza endogámica, sin embargo, en un estudio con selección a un índice combinando tamaño de camada con peso al nacimiento, pero con cruzamientos abiertos (no endogámicos) durante 150 generaciones, se obtuvieron tamaños de camada en la vida reproductiva de una pareja, de 17.6 crías nacidas y 20.2 crías en promedio(18), muy similares al promedio de CNAC al inicio de este estudio. La disminución de este promedio en las generaciones siguientes en el presente trabajo, se debe muy probablemente a la depresión endogámica, que en la línea no seleccionada fue de -1.705 crías por cada 10 % de consanguinidad (P<0.01). Cabe hacer notar que en las características CNAC y CDEST, originalmente utilizadas para obtener el CDHS (en toda la VR de cada pareja), hubo un efecto altamente significativo (P<0.01) de depresión endogámica en NS15G lo que no ocurre en la S15G (Cuadros 1,2,3). En aquel estudio, se observó un aumento en los niveles de testosterona en los machos, mientras que, en las hembras, hubo una elevación de progesterona en una de sus líneas; éstas mostraron un mayor número de ovocitos por ciclo, pero una mayor pérdida de embriones, y una disminución de la vida reproductiva, en comparación con la línea sin selección. Estos resultados coinciden con el presente trabajo, ya que también se obtuvo una disminución en la vida reproductiva en S1G y S20G (Cuadros 1,2).
1= 0.028). Otra diferencia es que la consanguinidad en dicho estudio es menor, aunque el número de generaciones de selección es de 124, durante ese tiempo (1972 a 2007), hubo varios cambios en la dirección de la selección en ese trabajo, pero se trató de evitar las cruzas endogámicas(17). No se encontró en la literatura, algún trabajo con selección a tamaño de camada de toda la vida reproductiva con cruza endogámica, sin embargo, en un estudio con selección a un índice combinando tamaño de camada con peso al nacimiento, pero con cruzamientos abiertos (no endogámicos) durante 150 generaciones, se obtuvieron tamaños de camada en la vida reproductiva de una pareja, de 17.6 crías nacidas y 20.2 crías en promedio(18), muy similares al promedio de CNAC al inicio de este estudio. La disminución de este promedio en las generaciones siguientes en el presente trabajo, se debe muy probablemente a la depresión endogámica, que en la línea no seleccionada fue de -1.705 crías por cada 10 % de consanguinidad (P<0.01). Cabe hacer notar que en las características CNAC y CDEST, originalmente utilizadas para obtener el CDHS (en toda la VR de cada pareja), hubo un efecto altamente significativo (P<0.01) de depresión endogámica en NS15G lo que no ocurre en la S15G (Cuadros 1,2,3). En aquel estudio, se observó un aumento en los niveles de testosterona en los machos, mientras que, en las hembras, hubo una elevación de progesterona en una de sus líneas; éstas mostraron un mayor número de ovocitos por ciclo, pero una mayor pérdida de embriones, y una disminución de la vida reproductiva, en comparación con la línea sin selección. Estos resultados coinciden con el presente trabajo, ya que también se obtuvo una disminución en la vida reproductiva en S1G y S20G (Cuadros 1,2).
En el presente estudio se reveló, que el número de EPPF tiene una disminución constante a lo largo de las generaciones estudiadas en NS15G, con un decremento de 0.331 estros post parto fértiles por cada 10 % de aumento en la consanguinidad (P<0.01), en comparación con S15G que se mantiene casi constante (P>0.05) (Cuadro 3); este comportamiento se observa también en CDHS (el índice productivo) con una disminución marcada en NS15G de las generaciones 1 - 5 a 6 - 10, con una ligera recuperación en las últimas cinco; la depresión endogámica fue de -0.062 (P<0.01) crías destetadas por hembra por semana por cada 10 % de aumento en consanguinidad, vs -0.001 (P>0.05) en S15G. Un estudio mostró que una deleción de Kiss1r en las neuronas del eje GnRH interrumpe la señal de kisspeptina, la proteína que induce la secreción de GnRH (hormona liberadora de gonadotropina); esto resulta en infertilidad debida a hipogonadismo, probablemente la consanguinidad tuvo un efecto negativo en este mecanismo, a través de la interrupción de esta señal(19) en los ratones S15G; en el presente trabajo no se midieron los efectos en los machos. Algo muy parecido se encontró en cerdos bajo selección para prolificidad, cuando ésta se hizo con índices de familias, ya que se vio un aumento en la depresión endogámica de tres veces más a la esperada sin selección y una disminución de la respuesta a la selección(20).
La VR, se vio afectada por la consanguinidad tanto en S15G, con una disminución de 4.741 días, en NS15G disminuyó casi tres días más (7.718 días) (P<0.01 en ambas) (Cuadro 3), en un estudio donde hubo selección para mayor tiempo de vida en ratones, se encontró que el aumento estuvo relacionado con mutaciones, que aumentaban los niveles de la hormona del crecimiento en el eje GH/IGF-1(21), que podría estar relacionada con una disminución en el tiempo de vida de los ratones, como sucedió en las líneas seleccionadas para un índice involucrando tamaño de camada y peso al nacer, sin consanguinidad(18) en el presente trabajo disminuyó en ambas líneas ya que el objetivo de selección fue distinto.
La mortalidad postparto es más alta en NS15G, desde las primeras cinco generaciones de cruzamiento consanguíneo estrecho, comparada con S15G, resultado similar al obtenido por Sallah(5), tanto en la línea seleccionada, como la no seleccionada, con apareamiento entre medios hermanos, donde, la mayor mortalidad se dio en las generaciones intermedias. Charlesworth(22) explica esto bajo dos hipótesis: 1) la de dominancia, donde la endogamia aumenta la frecuencia de los individuos que expresan los efectos de mutaciones deletéreas o 2) sobredominancia, donde los homocigóticos tendrían una menor aptitud por falta de alelos, con ventaja heterocigótica que mantendrían equilibrando la selección a frecuencias intermedias en los heterocigóticos.
Como resultado de lo anterior, el número de crías al destete por parto en NS15G, cae significativamente en las generaciones 6 a 10 (1.51 crías), con una recuperación en las siguientes generaciones acumuladas, mientras que, en el número de crías al nacimiento por parto, se mantiene en todas las generaciones (P<0.05) (Cuadro 1) (Figura 3). Un estudio reciente(23) reveló que, en camadas con menos de cuatro crías al nacimiento, en ratones sin selección, existe una mayor mortalidad al destete, y en el presente estudio la línea NS15G presentó menos de cuatro crías al nacer como promedio en las primeras cinco generaciones.
El resultado en estas características era presumible, ya que por un lado se puede esperar una alta depresión endogámica cuando se realiza cruza consanguínea estrecha (hermano, hermana) y, por otro lado, debido a la baja heredabilidad (0.024 a 0.063), del índice productivo(12), sólo se puede esperar un progreso de reproducción limitado; un resultado similar se obtuvo en el tamaño de camada con una consanguinidad de 0.61 en ocho generaciones de selección con consanguinidad en cruzas entre medios hermanos (5).
Estos resultados, conllevan a reflexionar si en un programa de selección en animales domésticos, aun evitando cruzamientos entre hermanos, generaciones más adelante se pudieran llegar a dar entre parientes, y esto induzca una endogamia, con los efectos contraproducentes que aquí se vieron.
En un programa de mejoramiento de ganado Holstein, se encontró que con 1 % de incremento en consanguinidad, la producción de leche en 305 días disminuyó 36.3 kg en promedio, en vacas de 4 a 5 años, y 2.42 kg de grasa(24). Recientemente, se evaluó la implementación de selección genómica, en la pérdida de diversidad genética en ganado Holstein y Jersey en Norte América, debida a la consanguinidad; sus resultados mostraron un incremento de endogamia de 1.19 a 2.06 % por generación, en un período de 10 años en ganado Holstein, y advirtieron sobre la necesidad de implementar medidas para evitar la endogamia en este tipo de programas(25). En la población Holstein de México, se encontró que, con niveles menores al 5 % de consanguinidad, no se detectó en grasa ni en proteína de la leche, ningún efecto, sin embargo, cuando la consanguinidad aumentó a más de 5 %, se encontró una disminución en la producción de leche de 260 kg por lactación, además de una pérdida en la producción de grasa de 11 kg y de 10 kg en proteína con respecto al promedio de los grupos con menos de 5 %(26).
Conclusiones e implicaciones
Como muestran los resultados de NS15G para un índice productivo (número de crías por hembra por semana), la depresión por consanguinidad afectó a sus distintos componentes, especialmente en las características reproductivas, que se pudieron regular en gran medida mediante una selección simultánea de éstas, probablemente debido a un mantenimiento de genes que favorecieron el desarrollo gonadal en las hembras y machos. El trabajo es relevante porque no se había analizado la selección de un índice productivo en ratones en sus diferentes componentes, de una forma integral, además de mostrar que, en un programa de selección con consanguinidad simultánea, se favorece la fijación de alelos deseables en el sostenimiento de los ciclos reproductivos y la sobrevivencia de las crías.
Literatura citada: