https://doi.org/10.22319/rmcp.v13i3.6067
Artículo
Detección del virus de la diarrea viral bovina en artiodáctilos silvestres en cautiverio en México
Jocelyn Medina-Gudiño a
Ninnet Gómez-Romero a
José Ramírez-Lezama b
Luis Padilla-Noriega c
Emilio Venegas-Cureño d
Francisco Javier Basurto-Alcántara a*
a Universidad Nacional Autónoma de México. Facultad de Medicina Veterinaria y Zootecnia, Departamento de Microbiología e Inmunología. Ciudad de México, México.
b Universidad Nacional Autónoma de México. Facultad de Medicina Veterinaria y Zootecnia, Departamento de Patología, Ciudad de México, México.
c Universidad Nacional Autónoma de México. Facultad de Medicina. Departamento de Microbiología y Parasitología. Ciudad de México, México.
d Servicio Nacional de Sanidad, Inocuidad y Calidad Agroalimentaria. Centro Nacional de Servicios de Diagnóstico en Salud Animal, Departamento de Validación de Técnicas, Estado de México, México.
*Autor de correspondencia: basurto@unam.mx
Resumen:
El virus de la diarrea viral bovina pertenece al género Pestivirus de la familia Flaviviridae. Los pestivirus infectan a un extenso rango de artiodáctilos, silvestres y domésticos, en los cuales ocasionan una gran variedad de desórdenes respiratorios, gastrointestinales y reproductivos que derivan en pérdidas relevantes para la industria pecuaria. El uso compartido de fuentes de agua y alimento entre los ambientes naturales y pecuarios incrementa el contacto directo e indirecto entre animales domésticos y silvestres, lo que aumenta el riesgo de transmisión interespecie de pestivirus. Por este motivo, la vigilancia de enfermedades causadas por pestivirus debería considerar la prevalencia de estos patógenos en animales silvestres. Actualmente se desconoce la diversidad genética de pestivirus en poblaciones silvestres en México. Este grupo de trabajo recolectó muestras de suero de 371 artiodáctilos silvestres en cautiverio en cuatro regiones de cuatro estados de México que incluyen a Veracruz, Querétaro, el Estado de México y la Ciudad de México. Dos muestras de suero de búfalas de agua y una muestra de suero de una gama fueron positivas al virus de la diarrea viral bovina mediante reacción en cadena de la polimerasa con transcripción reversa. El análisis filogenético de las secuencias amplificadas las agrupó dentro del subgenotipo 1b del virus de la diarrea viral bovina. Además, se logró el aislamiento de un virus citopático a partir de la muestra de suero de la gama. Este estudio constituye el primer reporte del virus de la diarrea viral bovina en artiodáctilos silvestres en México.
Palabras clave: Pestivirus, VDVB 1b, Genotipificación Aislamiento, Fauna silvestre.
Recibido: 20/09/2021
Aceptado: 21/12/2021
Introducción
El virus de la diarrea viral bovina (VDVB) pertenece al género Pestivirus, ubicado dentro de la familia Flaviviridae. Este género incluye al virus de la diarrea viral bovina 1 (VDVB1) y al virus de la diarrea viral bovina 2 (VDBV2), entre otros virus de importancia veterinaria. De acuerdo con el Comité Internacional de Taxonomía de los Virus (ICTV), el VDB1 y el VDVB2 se encuentran actualmente clasificados en las especies Pestivirus A y Pestivirus B, respectivamente(1). El VDVB1 tiene 21 subgenotipos (1a-1u), mientras que el VDVB2 tiene 4 subgenotipos (2a-2d)(2). Adicionalmente, las cepas del VDVB se clasifican como biotipos citopáticos (CP) o no citopáticos (NCP), dependiendo de su efecto en el cultivo celular(3). Los biotipos CP causan vacuolización y muerte celular, mientras que los biotipos NCP no provocan alteraciones visibles en el cultivo celular(4,5).
El VDVB infecta a un amplio rango de animales pertenecientes al orden Artiodactyla. Por varias décadas, el VDVB ha sido principalmente reconocido como un agente patógeno para los bovinos domésticos, en los que puede causar desde infecciones subclínicas hasta infecciones caracterizadas por inapetencia, fiebre transitoria, diarrea, alteraciones respiratorias y alteraciones reproductivas como abortos, momificaciones, defectos congénitos, mortinatos o el nacimiento de animales inmunotolerantes persistentemente infectados (PI)(6,7). En la actualidad, al VDBV también se le reconoce como agente causal de alteraciones reproductivas, respiratorias, inmunológicas y neurológicas en artiodáctilos silvestres(8-14).
El VDVB puede transmitirse verticalmente por infección transplacentaria, durante la monta o mediante el uso de semen o embriones infectados(15). Mientras que la transmisión horizontal ocurre mediante el contacto directo o indirecto con secreciones orales y nasales de animales infectados(12). Factores como el uso común de suelo y las migraciones animales pueden promover la diseminación de pestivirus entre animales domésticos y silvestres(9), debido a que las fuentes naturales de agua y comida representan puntos comunes de interacción en los que los virus pueden diseminarse hacia una gran variedad de huéspedes(12).
Las infecciones por el VDVB generan pérdidas económicas significativas para la industria pecuaria, atribuidas a la pérdida de la producción láctea, disminución en el rendimiento reproductivo, retraso en el crecimiento, defectos congénitos, predisposición a enfermedades concomitantes y aumento en la mortalidad de animales jóvenes(16). En países como Inglaterra y Nueva Zelanda, se estima que las pérdidas ascienden a 46 millones de dólares/año y 44.5 millones de dólares/año, respectivamente(17,18). Mientras que en Dinamarca ocurren pérdidas aproximadas de 20 millones de dólares por cada millón de partos(19).
En México, la información de la diversidad genética de los pestivirus circulantes se encuentra limitada a un estudio realizado en ganado bovino, en el cual las variantes genéticas identificadas incluyen a los subgenotipos 1a, 1b, 1c y 2a del VDVB(20).
Al considerar el impacto económico que las infecciones por el VDVB puede ocasionar en la industria pecuaria(17-19), que la transmisión del VDVB entre artiodáctilos domésticos y silvestres es posible(9,12,21) y que se ha detectado la presencia de anticuerpos contra el VDVB en venados cola blanca en México, el objetivo de este estudio fue detectar e identificar a los genotipos del VDVB presentes en animales silvestres en México para determinar su potencial prevalencia en las poblaciones evaluadas.
Material y métodos
Muestras de suero
Se recolectaron 371 muestras de suero de artiodáctilos silvestres en cautiverio con edades de 2 a 3 años. Las especies muestreadas incluyeron búfalos de agua (Bubalus bubalis), venados gamos (Dama dama), venados cola blanca (Odocoileus virginianus), antílopes eland (Taurotragus oryx) y jabalíes (Sus scrofa) provenientes de Veracruz, Querétaro, el Estado de México y la Ciudad de México.
Se recolectaron entre 3 ml y 6 ml de sangre a partir de la vena yugular en tubos VacutainerTM. Una vez formado el coágulo, las muestras se centrifugaron por 20 min a 2,000 rpm en una centrífuga clínica. El suero se transfirió a tubos de microcentrífuga y se almacenó a -70 °C hasta su uso.
Extracción de ARN
El ARN total se extrajo de las muestras de suero con TRIzolTM LS Reagent, de acuerdo con las recomendaciones del fabricante. Brevemente, se mezclaron 400 µl de suero con 900 µl de TRIzolTM LS Reagent al invertir seis veces el tubo de microcentrífuga. La mezcla se incubó durante 5 min a 4 °C. Se añadieron 240 µl de cloroformo, se homogeneizó y posteriormente se incubó por 5 minutos a 4 °C. La mezcla se centrifugó por 15 min a 13,000 g y 4 °C. Se transfirieron 200 µl del sobrenadante a un tubo libre de nucleasas, se añadieron 600 µl de isopropanol y se homogeneizaron. La mezcla se incubó a -20 °C por una hora y posteriormente se centrifugó por 15 min a 13,000 xg y 4 °C. Se descartó el sobrenadante, se lavó el botón con 1 ml de etanol al 75% y se centrifugó por 5 min a 13,000 xg y 4 °C. Se descartó el sobrenadante y se secó el botón por 5 min a temperatura ambiente. Los botones se suspendieron en 20 µl de agua libre de nucleasas y se almacenaron hasta su uso a -70 °C. También se extrajo el ARN total de células MDBK libres del VDVB y de la cepa de referencia NADL para incluirlos respectivamente como control negativo y positivo en la RT-PCR.
Transcripción reversa (RT)
La retrotranscripción se realizó de acuerdo con el protocolo descrito por el fabricante de la M-MLV Reverse Transcriptase (Thermo-Fisher) en un volumen total de 20 µl/reacción. Brevemente, se utilizaron: 500 ng de ARN (1-10 µl), 1 µl de iniciadores aleatorios (Invitrogen) (0.2 µg/µl), 1 µl de dNTP Mix (10 mM c/u) y agua libre de nucleasas (c.b.p. 12 µl). Se homogeneizó mediante pipeteo y se incubó durante 5 min a 65 °C. Posteriormente se añadieron 4 µl de Buffer (5 X), 2 µl de DTT (0.1 M) y 1 µl de ribonuclease inhibitor (40 U/µl). Se homogeneizó mediante pipeteo y se incubó durante 2 min a 37 °C. A continuación, se agregó 1 µl de M-MLV Reverse Transcriptase (200 U/ µl) y se incubó por 50 min a 37 °C para sintetizar el ADN complementario. La reacción se detuvo al calentar la mezcla por 15 min a 70 °C.
Reacción en cadena de la polimerasa (PCR) y secuenciación
Para la PCR de la región 5’ UTR de los pestivirus se utilizaron los iniciadores 5UTRfwd/STAR-Trev, 324/326 y HCV90/HCV368, los cuales amplifican respectivamente a fragmentos de 292 pb, 288 pb y 278 pb(22-24). Cada reacción se preparó en un volumen total de 25 µl con: 2 µl de cDNA, 1 µl de iniciador sentido (10 µM), 1 µl de iniciador antisentido (10 µM), 1 µl de dNTP Mix (10 mM c/u), 0.2 µl de Taq polimersa (5 U/µl), 5 µl de buffer (10 X) y 14.8 µl de agua libre de nucleasas.
La PCR se realizó en un termociclador Select Cycler II (Select BioProducts) con los parámetros descritos a continuación. Desnaturalización inicial durante 4 min a 94 °C, 30 ciclos de amplificación con: desnaturalización por 30 seg a 94 °C; alineamiento por 30 seg a 56.2 °C (iniciadores 5UTRfwd/STAR-Trev), 55 °C (iniciadores 324/326) y 50 °C (iniciadores HCV90/HCV368); extensión por 30 seg a 72 °C. Extensión final durante 10 min a 72 °C.
Los productos de la RT-PCR se separaron mediante electroforesis en gel de agarosa al 1%, de acuerdo con la metodología descrita(15,25). Los geles de agarosa se tiñeron con GelRed® de acuerdo con el protocolo del fabricante. Los productos de la RT-PCR se visualizaron mediante un transiluminador de luz ultravioleta y se purificaron con el QIAquick® Gel Extraction Kit (Qiagen GmbH) de acuerdo con el protocolo del fabricante. Los amplicones se secuenciaron por duplicado en ambas direcciones con el BigDyeTM Terminator Sequencing Kit en el ABI PRISM® 3130xl Genetic Analyzer del Instituto de Biotecnología de la UNAM.
Análisis filogenético
La reconstrucción filogenética de las secuencias de la región 5’ UTR de los virus detectados se realizó con el programa MEGA 10, mediante el método de máxima verosimilitud y el modelo de sustitución Kimura 2 con 1,000 bootstraps(26-28).
Aislamiento viral
El aislamiento viral se realizó mediante el uso de técnicas estándar para cultivo celular(29). Brevemente, se utilizaron cajas para cultivo celular de 25 cm2 con un 70 % de confluencia de células MDBK (libres del VDVB) en MEM suplementado con suero equino al 5%. A partir de las muestras de suero identificadas como positivas mediante RT-PCR, se depositaron 500 μl de suero en las cajas para cultivo celular. La identificación de los cultivos de células MDBK infectados se realizó mediante la observación de los efectos citopáticos (vacuolización y lisis) a las 48 h de incubación, RT-PCR y secuenciación.
El título del aislamiento se determinó mediante el método de Reed-Muench(30). Brevemente, se realizaron diluciones del virus aislado mediante el uso de microplacas de 96 pozos. Las diluciones logarítmicas decimales se realizaron por triplicado desde 10-1 hasta 10-8. En cada uno de los pozos empleados en la titulación se colocaron 100 μl del sobrenadante del cultivo con el virus aislado y 900 μl de MEM suplementado con suero equino al 5%. Las microplacas se incubaron por 48 h a 37 ºC.
Resultados
Se detectó el ARN del VDVB mediante RT-PCR con los iniciadores 324/326 en la muestra de suero de una gama de dos años identificada como 7916 (Ciudad de México). Mientras que los iniciadores 5UTRfwd/STAR-Trev y 324/326 permitieron la detección del ARN del VDVB en las muestras de suero de dos búfalas de agua de seis meses identificadas como BUMA 6 y BUMA 15 (Mil Aguas, Veracruz).
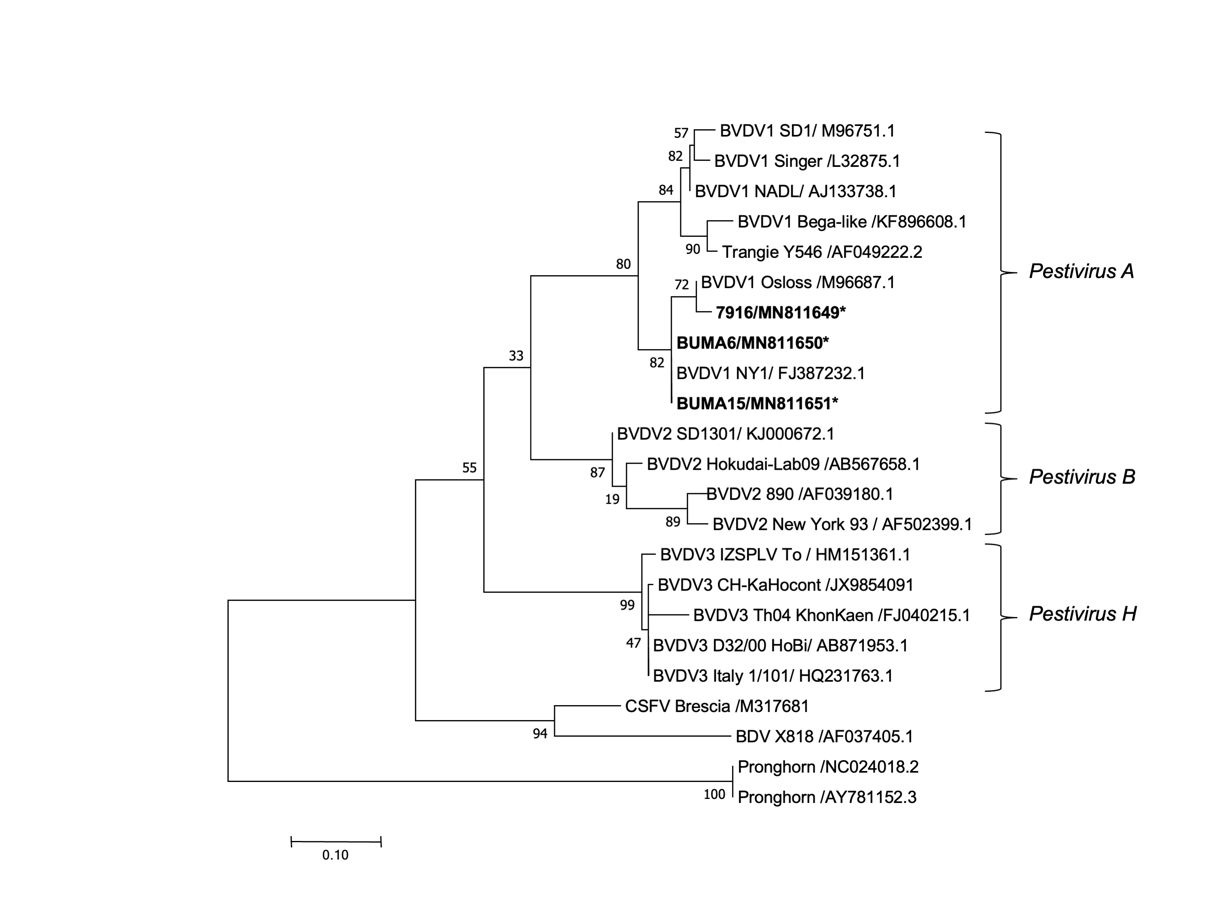
Se realizó el análisis filogenético de los amplicones de la región 5’ UTR para determinar las especies y subgenotipos del VDVB detectado en las muestras. Las secuencias de las 3 muestras positivas se agruparon en el subgenotipo 1b del VDVB (Figura 1).
Figura 1: Análisis filogenético de pestivirus basado en un fragmento de la región 5´ UTR
La reconstrucción del árbol filogenético enraizado de la región 5´ UTR se elaboró con el método de máxima verosimilitud en Mega 10, con el modelo de sustitución Kimura 2 y 1,000 bootstrap. Las secuencias de virus de referencia se encuentran identificadas por su número de acceso del GenBank, mientras que las secuencias virales obtenidas en este estudio se identifican en negritas y con un asterisco.
El análisis filogenético de la región 5’ UTR indicó que las secuencias provenientes de las muestras identificadas como BUMA 6 y BUMA 15 (número de acceso GenBank: MN811650 y MN811651) presentan un 99.8 % de identidad con la cepa de referencia NY del VDVB, perteneciente al subgenotipo 1b. Mientras que la secuencia proveniente de la muestra identificada como 7916 (número de acceso GenBank: MN811649) tiene un 99.8 % de identidad con la cepa de referencia Osloss del VDVB, también perteneciente al subgenotipo 1b.
Se obtuvo el aislamiento de un virus con biotipo CP a partir de la muestra de suero proveniente de la gama 7916 mediante el uso de células MDBK libres del VDVB. El título del VDVB 1b aislado fue de 106 DICC50/ml. No se logró el aislamiento viral a partir de las muestras de suero provenientes de las búfalas de agua BUMA 6 y BUMA 15.
Discusión
El subgenotipo 1b del VDVB se considera el más distribuido a nivel mundial en el ganado bovino, seguido por los subgenotipos 1a y 1c(2). El subgenotipo 1b ha sido detectado en artiodáctilos domésticos en diversos países (incluido México) de cinco continentes(2,20). Mientras que, en animales silvestres, el subgenotipo 1b ha sido detectado en países como Canadá (bisonte)(31), China (yak)(32), Alemania (bongo)(33) y Estados Unidos (alpaca y venado cola blanca)(34-35).
La información actualmente disponible sobre la prevalencia de diarrea viral bovina en animales silvestres en México corresponde a un único estudio realizado por Cantu et al. en 2008. Los autores obtuvieron muestras de suero de venados cola blanca procedentes de tres estados del noreste de México (Coahuila, Nuevo León y Tamaulipas) y determinaron una seroprevalencia del 63.5 % en las muestras analizadas(36). Sin embargo, el estudio se limitó a la detección de anticuerpos y no se realizó la identificación directa del VDVB ni su caracterización genética, por lo que el presente estudio representa el primer reporte de aislamiento y detección directa del VDVB en animales silvestres en México, así como el primer reporte de la prevalencia del VDVB en animales silvestres del sureste del país.
Aunque los iniciadores utilizados pueden permitir la detección de diversas especies de pestivirus (Pestivirus A-E, G-H)(22-24), en este trabajo solo se detectó el ARN del subgenotipo 1b del VDVB1 (Pestivirus A), el cual se encontró en aproximadamente el 1% de las muestras de artiodáctilos silvestres analizadas.
La circulación del subgenotipo 1b del VDVB1 en animales domésticos y silvestres en México, sugiere la posibilidad de diseminación del virus entre ambos tipos de poblaciones animales. Por lo que la información generada en este trabajo podría ser de utilidad en el diseño de programas eficaces para el control de la enfermedad que incluyan el monitoreo de artiodáctilos domésticos y silvestres, así como el uso de vacunas que confieran protección específica contra los subgenotipos actualmente circulantes del VDVB en México.
Las infecciones con los biotipos CP o NCP del VDVB tienen diferentes implicaciones en la severidad de la enfermedad(37,38). El biotipo NCP es comúnmente detectado en muestras asociadas con alteraciones respiratorias, mientras que el biotipo CP suele detectarse en muestras de animales con alteraciones reproductivas, entéricas o sistémicas(38). Las infecciones con el biotipo CP pueden causar trastornos en el desarrollo embrionario, como momificaciones, hidrocefalia, displasia retinal, artrogrifosis y abortos(39). Por otra parte, las infecciones intrauterinas con el biotipo NCP que ocurren entre los días 42 y 125 de la gestación, pueden ocasionar infecciones persistentes en el feto y por consiguiente, el nacimiento de animales persistentemente infectados que son inmunotolerantes al VDVB, permanecen seronegativos y diseminan al virus a lo largo de toda su vida(6,40). Además, la infección simultánea con biotipos CP y NCP en artiodáctilos PI ocasiona una presentación letal, conocida como enfermedad de las mucosas. Por lo que la presencia de biotipos CP y NCP en una misma explotación o localidad es un evento relevante que debe considerarse porque incrementa la posibilidad de que ocurran coinfecciones con ambos biotipos(41).
Los toros PI pueden diseminar entre 104 y 107 DICC50/ml del VDVB en semen a lo largo de su vida(42), título similar al del VDVB aislado en este estudio a partir de una gama clínicamente sana (106 DICC50/ml). Este título sugiere que el animal podría haber cursado con un estado temprano de viremia durante una infección aguda, aunque aún subclínica, lo que permitiría la detección del virus en la muestra de suero. El título es similar a lo indicado en estudios previos, donde se reportan 105.8 DICC50/ml en suero y desde 105.8 DICC50/ml hasta 106.3 DICC50/ml en secreciones nasales de venados cola blanca(12).
En contraste con los hallazgos previamente descritos en otros estudios, el aislamiento del VDVB CP aquí detallado se obtuvo de una gama libre de signos clínicos. Se desconoce si el título viral del aislamiento con el biotipo CP y la ausencia de signos clínicos observados en esta gama en particular, podrían estar relacionados con una infección subclínica recurrente. Cabe destacar que el VDVB aislado a partir del suero de la gama fue capaz de replicarse en células de origen bovino (MDBK) in vitro, lo que sugiere que este aislamiento podría ser capaz de infectar a ambas especies, aunque se requiere realizar estudios adicionales para evaluar su capacidad de transmisión y virulencia en ambas especies.
En conclusión, este estudio es el primer informe de detección y aislamiento del VDVB (Pestivirus A, subgenotipo 1b, biotipo CP) en búfalos y venados gamo en México. No se detectaron otras especies de pestivirus en el análisis de las muestras. Se requieren más estudios para obtener una mejor caracterización de los pestivirus circulantes en poblaciones de animales silvestres en México porque pueden representan una fuente de infección para las especies domésticas y viceversa. No obstante, la información generada en este estudio contribuye a un mejor entendimiento de la diversidad genética y epidemiológica de pestivirus en las poblaciones evaluadas.
Agradecimientos
El financiamiento para este estudio fue proporcionado por el Programa de Apoyo a Proyectos de Investigación e Innovación Tecnológica IN217919 “Identificación y caracterización genética de las cepas del virus de la diarrea viral bovina circulantes en poblaciones ganaderas de México”. Facultad de Medicina Veterinaria y Zootecnia. Universidad Nacional Autónoma de México (FMVZ-UNAM).
Conflicto de intereses
Los autores declaran que no existe conflicto de intereses.
Aprobación ética
La recolección de las muestras fue aprobada por el Comité Interno para el Cuidado y Uso de los Animales. Facultad de Medicina Veterinaria y Zootecnia. Universidad Nacional Autónoma de México (FMVZ-UNAM).
Literatura citada: