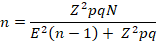
https://doi.org/10.22319/rmcp.v13i3.5873
Artículo
Detección del virus de la lengua azul en ovinos por RT- PCR en tiempo real en diferentes sistemas de producción en San Martín, Perú
Alicia María López Flores a*
Roni David Cruz Vasquez a
Víctor Humberto Puicón Niño de Guzmán a
Alicia Bartra Reátegui a
Orlando Ríos Ramírez a
Fredy Fabián Domínguez a
a Universidad Nacional de San Martin. Laboratorio de Sanidad Animal de la Escuela Profesional de Medicina Veterinaria, San Martin, Perú.
* Autor de correspondencia: alicialopezflores@unsm.edu.pe
Resumen:
El presente estudio tuvo como objetivo determinar la prevalencia del Virus de la Lengua Azul (VLA) en ovinos, por la técnica reacción en cadena de la polimerasa con Transcripción Reversa (RT- PCR) en tiempo real. Se evaluaron 366 ovinos procedentes de las diez provincias de la región del Perú. La metodología empleada fue la toma de muestras sanguínea de la vena yugular del ovino, seguidamente se realizó el proceso de extracción y purificación de ARN con el kit QIAmp®, luego la transcripción reversa para obtener el ADNc, y finalmente realizar la RT- PCR en tiempo real, para lo cual se utilizó el kit SuperScript III platinium One- step qRT–PCR, siendo los iniciadores y sondas dirigidos al segmento 10 del gen NS3 del VLA. Los resultados de la prueba de RT-PCR en tiempo real revelaron dos ovinos positivos con valor de ciclo umbral (Ct) de 35.21 y 35.57, siendo la prevalencia de 0.54 % de ovinos positivos a VLA en sistema de crianza extensiva, con las condiciones del ambiente que favorecen el desarrollo del vector Culicoide. Se concluye que mediante la técnica RT-PCR en tiempo real, se confirma la presencia del VLA en esta región del Perú, lo que hace necesario futuros estudios para determinar la detección de otros serotipos potenciales de VLA en la Amazonia peruana, con la finalidad de mejorar las estrategias de control de la enfermedad.
Palabras clave: ARN, Gen, Diagnóstico molecular, Rumiantes, Amazonia.
Recibido: 19/11/2020
Aceptado: 08/11/2021
Introducción
El virus de la lengua azul (VLA), genero Orbivirus(1), sin envoltura, el genoma posee 10 segmentos de doble cadena de ARN, siete proteínas estructurales y cinco no estructurales(2). La VP7 es una proteína de la cápside vírica(3). La VP2 de la superficie del virión(4). Además, los serotipos 9, 13, 18(5) y 27(6), siendo el serotipo 28 en identificarse últimamente(7).
La lengua azul es una enfermedad, no contagiosa y transmitido a través de la picadura de insectos Culicoides(8). De los 1,357 especies de Culicoides en el mundo(9), solo unos 30 han sido reportados como vectores(10). Los vectores al alimentarse de un animal con VLA, estarán infectados toda su vida(11), pueden infectar a los animales con variaciones en los signos clínicos(12).
En ovinos, se manifiesta descarga nasal seromucosa, edema facial, fiebre trombo hemorrágico, coronitis y ulceración en labios y paladar duro(13); la viremia suele detectarse entre 3 y 5 días después de la infección alcanzando puntos máximos febriles en el día 7(14). Sin embargo, estos signos clínicos (p. ej., fiebre en ovinos y bovinos, salivación en bovinos, edema facial en ovinos) se observaron en rebaños infectados y no infectados, estos signos no son un indicativo de la enfermedad(15). Así mismo, las lesiones macroscópicas son edema pulmonar, consolidación pulmonar cráneo - ventral, lengua tumefacta y cianótica; y en mucosa ruminal lesiones hemorrágicas(16). El bovino, caprino, bubalinos y otros rumiantes silvestres, actúan como reservorios del virus, siendo animales que podrían no manifestar signos clínicos(17,18,19), sin embargo, existen reportes de mortalidad en bovinos con el serotipo 8 del VLA(20); otros signos clínicos como la amaurosis que es la imposibilidad de pararse y la ausencia de reflejo de succión en terneros infectados(21). Por lo tanto, los diferentes serotipos del VLA en rumiantes pequeños y grandes, indica su expansión enzoótica(22). Así mismo, la transmisión del virus, huésped-vector son complejos, con una variedad de impulsores ecológicos(23).
La epidemiologia del VLA incluida en la lista del Código Sanitario para los Animales Terrestres de la OIE(24). Ha generado brotes en Israel en el 2006, generando pérdidas de $ 2.5 millones(25), así mismo, un brote en Europa Occidental(26). De este modo, ha repercutido en un costo anual de $ 3,000 millones, se convierte en una de las enfermedades de importancia económica(27).
En la actualidad, esta enfermedad es a nivel mundial, excepto la Antártida(28). Reportado en Brasil en ovinos(16); en Sudán, en trastornos reproductivos en bovinos(29); en Japón(30) e India, en una transmisión transplacentaria del VLA-1, en la etapa media de gestación en ovinos(31). En Australia, el VLA, se originaron a partir del Sudeste Asiático(32). Mientras en Reino Unido, los animales silvestres en zoológicos son susceptibles a arbovirus, actuando como hospederos nativos de los Culicoides(33).
En Perú, el VLA ha sido reportado en ovinos, camélidos y en animales silvestres de áreas tropicales(34,35,36). Los estudios preliminares, se realizaron en 1984 y 1987, encontrándose 88, 41 y 56 % de ovinos seropositivos a VLA provenientes de tres regiones (norte, centro y sur)(37) y 21 % de alpacas seroreactores de la región sur(38). Estudios en Tayassu tajacu en Madre de Dios, se reporta un 7.5 % de muestras con anticuerpos específicos al VLA y el 29.2 % tuvieron anticuerpos al serogrupo VLA(34). Recientemente, Navarro et al(35) confirmaron la presencia del virus en ovinos de crianza extensiva, además identificaron los Culicoides vectores de la enfermedad.
No obstante, la región San Martín en su clima tropical, es un ambiente óptimo para el vector y facilita sus vías de infección en hospedadores domésticos y silvestres; mencionado por Felippe-Bauer(39) quienes reportaron especies de Culicoides.
En relación al diagnóstico, la prueba de seroneutralización, es específica, pero su desventaja es la baja sensibilidad y costo(40). No obstante, el ELISA competitivo es sugerido en llamas, rumiantes silvestres(41), ovinos y caprinos(42). En la actualidad, las pruebas recomendadas por la OIE para VLA son la RT- PCR, el ensayo de inmunodifusión en gel de agar y el ELISA competitivo(43,44).
Siendo la RT- PCR en tiempo real para la detección rápida del VLA dirigido al Seg-1 / VP1(45,46), Seg-2/VP2 y Seg-10/NS3(47), es el método ampliamente utilizado(48), en la detección de todos los serotipos de VLA(49), basada en sonda de fluorescencia TaqMan(50,51,52,53), para detectar VLA en muestras de ovinos infectados(54). Por lo tanto, el objetivo del estudio fue detectar al VLA en ovinos mediante RT- PCR tiempo real, considerándose la influencia de las características ambientales y presencia de la enfermedad.
Material y métodos
Lugar de estudio
La investigación se realizó en la región San Martín, Perú. Se recolectaron muestras sanguíneas en ovinos en condición de salud estable, en las 10 provincias de la región San Martín (Cuadro 1), en el periodo de agosto a diciembre de 2018.
Cuadro 1: Cantidad de ovinos muestreados por provincia idóneas para el desarrollo del vector
Provincias | N° Muestras |
Moyobamba | 9 |
Rioja | 11 |
El Dorado | 35 |
Lamas | 41 |
Bellavista | 74 |
Picota | 41 |
Mariscal Cáceres | 15 |
Huallaga | 24 |
Tocache | 30 |
San Martin | 86 |
Total | 366 |
Recolección de muestra
Se recolectaron 366 muestras de sangre de la vena yugular de los ovinos seleccionados al azar, provenientes de 42 granjas. Para la recolección se emplearon agujas y tubos vacutainer al vacío con anticoagulante EDTA. Las muestras se transportaron en un contenedor refrigerado con gel refrigerante para su procesamiento en el laboratorio de Biología y Genética Molecular de la Escuela Profesional de Agronomía - Facultad Ciencias Agrarias, Universidad Nacional San Martin.
Según el Censo del 2012(55), la población es de 7,656 ovinos en todo el departamento de San Martin. Basado en esta población se calculó la muestra:
Donde: n es el tamaño de la muestra; Z= es el nivel de confianza 95%= 1.96; p= es la probabilidad de éxito 50%/100= 0.5; q= es la probabilidad de fracaso 50%/100 = 0.5; E= es el nivel de error 5%/100 = 0.05; N= es el tamaño de la población= 7656
n = (1.96)2 (0.5) (0.5) (7656)
(0.05)2(7656-1) + (1.96)2(0.5) (0.5)
n=366
Fórmula de la subpoblación muestral.
Nh= subpoblación o grupo; N= población total ; ,-.n= muestra total; nh=muestra de los grupos.
Extracción de ARN
Para el procesamiento de la muestra sanguínea se llevó a centrifugar a 800 xg durante 10 min, seguidamente se extrajo el plasma y se adicionó PBS estéril, se invirtió el tubo varias veces para mezclar, volver a centrifugar a 800 xg durante 10 min para separar los glóbulos rojos del PBS. Para la extracción de ARN se utilizó el kit QIAmp®, marca Qiagen. El paso de extracción del ARN se realizó según los procedimientos especificados del fabricante. El producto final obtenido es ARN en Buffer, para finalmente seguir con la RT- PCR en tiempo real que tiene por diana el segmento 10 del VLA (Gen NS3).
Transcripción reversa - Reacción en cadena de la polimerasa en tiempo real
La RT- PCR en tiempo real es una prueba de elección para el diagnóstico. El método aquí descrito es recomendado por la OIE y Hofmann et al(56), para detectar el segmento 10 del Gen NS3 del VLA. Para la obtención del ADNc mediante transcripción reversa y PCR en tiempo real se utilizó el kit SuperScript III platinium One- step qRT –PCR, marca Invitrogen, siendo las secuencias de los iniciadores para la detección del segmento 10 del gen NS3 del VLA. Las soluciones primarias del iniciador se diluyeron a una concentración de 20 pmol/μl, las secuencias nucleotídicas de los iniciadores: VLA_IVI_F 5’-TGG-AYA-AAG-CRA-TGT-CAA-A-3’, VLA_IVI_R 5’-ACR-TCA-TCA-CGA-AAC-GCT-TC-3’(57). La solución de la sonda para el gen NS3 del VLA, se diluyó hasta una concentración de 5 pmol/μl, la secuencia de la sonda: VLA_IVI_P 5’FAM-ARG-CTG-CAT-TCG-CAT-CGT-ACG- C-3’ BHQ1.
Se adicionaron 0.5 μl de cada iniciador primario a concentración de 20 pmol/μl a cada pocillo, la placa se debe mantener sobre hielo. Seguidamente se adiciona 2 μl de muestras de ARN, tanto de la muestra diana como de los controles positivos y negativos, a los pocillos correspondientes de la placa siguiendo la distribución. La temperatura de desnaturalización fue 95 °C durante 5 min, y se mantuvieron sobre hielo otros 3 min. Se preparó un volumen de la mezcla primaria(57) de la RT- PCR, siguiendo las instrucciones del fabricante. La sonda se incluyó en la mezcla primaria para obtener una concentración final de 0.2 pmol/μl por muestra. Se distribuyeron 22 μl de mezcla primaria en cada pocillo de la placa situado en el termociclador de tiempo real programado para una transcripción reversa y amplificación, detección mediante fluorescencia del ADNc.
Condiciones de temperatura
La reacción fue llevada en light cycler 480 System Roche Applied Biosystems. Siguiendo el siguiente perfil térmico: Transcripción reversa 48 °C por 30 min, inactivación de la transcriptasa reversa o desnaturalización inicial 95 °C por 2 min, seguido, por 50 ciclos, amplificación 95 °C por 15 seg, 56 °C por 30 seg, 72 °C por 30 seg(57).
Resultados y discusión
Análisis RT- PCR en tiempo real
Los resultados de RT-PCR en tiempo real indicó que el 0.54 % (2/366) de las muestras fueron positivos al VLA. Para el primer individuo con código (E8), positivo a VLA presentó un valor de ciclo umbral (Ct) de alrededor de 35.21, con un valor de temperatura de disociación™ de 84 °C para VLA. Para la muestra con código (G12) – C+ VLA 1/10, presentó un Ct de 28.22 y 31.63 para la muestra con código (H12) – C+VLA 1/100. No se observó amplificación en el control negativo. Se evidencio la alta carga viral y la amplificación de un producto específico (Figura 1).
Figura 1: Resultados de la prueba de RT-PCR en tiempo real para el primer individuo positivo (E8), en la curva de amplificación, se observa con valor de Ct de 35.21. Software Opticon Monitor v.3.0
Para el segundo individuo (E6), positivo a VLA presentó un valor de Ct de alrededor de 35.57, con un valor de temperatura de disociación™ de 84 °C. Para la muestra (G12)- C+VLA 1/10, presentó un Ct de 28.81 y 32.72 para la muestra (H12)- C+VLA 1/100. No se observó amplificación en el control negativo. Se evidenció la alta carga viral y la amplificación de un producto específico (Figura 2).
Figura 2. Resultados de la prueba de RT-PCR en tiempo real para el segundo individuo positivo (E6), en la curva de amplificación, se observan un valor Ct de 35.57. Software Opticon Monitor v.3.0.
Una revisión panorámica en América del Sur, mediante estudios serológicos para la detección de anticuerpos efectuados en bovinos, caprinos, ovinos y búfalos indica variadas prevalencias mínimas y máximas en los siguientes países: en Argentina (0–95 %)(58), Brasil (1.22-89.69 %)(59,60), Chile (0-19.6 %)(61,62), Colombia (51.8-56 %)(63.64), Ecuador (10 %)(65), Guyana(0-56 %)(66), Suriname (82-91 %)(66) y Venezuela (74.8-94.7 %)(67); en el Perú, los estudios preliminares de 1984 y 1987 reportaron una seroprevalencia de 87.5, 41 y 55.5 % al VLA en ovejas del norte, centro y sur de la sierra del país(37). En la presente investigación, se detectó el genoma viral del VLA, siendo la prevalencia de 0.54 % en sueros de ovinos mediante la RT- PCR en tiempo real, que confirma la presencia epidemiológica de este virus en esta región. Las zonas donde se detectaron los animales positivos se encuentran muy cerca de afluentes de agua como el rio Sisa y la quebrada de Ishangayacu. Siendo las sensibilidades ambientales como componente clave de la capacidad del vector(68).
Asimismo, es posible que la infección por el VLA en los ovinos muestreados haya sido subclínica, ya que los animales estuvieron en condiciones normales. Esto se explica según Maclachlan et al(69), porque el desarrollo de los signos clínicos de la enfermedad depende si la infección es endémica o no; como consecuencia, los animales presentan anticuerpos, pero rara vez presentan signos clínicos. Esto es posiblemente porque en el ovino el periodo de viremia raramente persiste por más de 14 días, a diferencia del bovino cuya viremia puede ser de hasta 90 a 120 días.
Tal como lo menciona Navarro(36), el Perú es uno de los países predisponentes a presentar la enfermedad de VLA, ya que posee diversos ecosistemas propicios para el desarrollo de Culicoides. Se considera necesario la determinación de la seroprevalencia del VLA en zonas ganaderas, identificar los diversos serotipos, levantar un mapa de la ubicación de las diversas especies de Culicoides spp., en las diferentes áreas geográficas y altitudes, y determinar las zonas endémicas.
Entorno de los ovinos positivos a VLA
Se realizó una evaluación de los alrededores de las granjas para observar el hábitat del vector Culicoide. Encontrándose que los animales positivos a VLA están cerca de afluentes de agua, lo que contribuye con el desarrollo del vector, (Cuadro 2). Por otra parte, los ovinos positivos fueron de la raza Pelibuey, sin embargo, no es determinante la predisposición por la raza. Además, la crianza extensiva de los ovinos permite su crianza junto a los vacunos, gallinas, perros y caballos. En las encuestas realizadas los propietarios de las granjas extensivas, intensivas y semi-intensiva no desparasitan a sus animales, ni fumigan por la presencia de moscas, mosquitos o zancudos.
Cuadro 2: Entorno de los ovinos positivos al VLA
Provincia | Distrito | Ubicación de la granja | Sistema de explotación | Entorno de la granja | Raza | Condición |
Bellavista | San Pablo | Sector Angélica | extensivo | Margen izquierda del rio Sisa | Pelibuey | 3 |
Bellavista | San Pablo | Caserío de San Ignacio | extensivo | Margen izquierda de la quebrada Ishangayacu | Pelibuey | 2.5 |
Estudios epidemiológicos, indican que el VLA existe en una extensa zona en el mundo entre los 40° latitud norte y 35° latitud sur, con ecosistemas tropicales, subtropicales y templados(70), coincidiendo estas características con la región San Martín.
Los primeros estudios de la presencia del vector transmisor del VLA en nuestra región se dieron con Felippe-Bauer et al(39), quienes identificaron cinco especies de Culicoides vectores del virus. De igual manera, Navarro et al(35), de los 7,930 mosquitos capturados, el 94.8 % fueron identificados como Culicoides insignis, además se confirma la presencia del VLA en ovinos en la región Pucallpa(36).
Por otra parte, el vector Culicoide suele desarrollarse en zonas donde existen ciertos tipos de conductores como el uso de la tierra, el comercio, la crianza de animales y la presencia de animales silvestres como reservorio del VLA; este último se refuerza con el trabajo que realizaron Rivera et al(34), quienes encontraron un 7.5 % de huanganas (Tayassu pecari) positivos al VLA en la región Madre de Dios.
El presente trabajo de investigación es el primero que se desarrolló en toda la región San Martin, donde los resultados mediante RT- PCR en tiempo real dan la presencia VLA. Felippe-Bauer et al(39) mencionan haber encontrado el vector Culicoide del virus. Por lo tanto, se requiere detectar otros serotipos existentes de VLA en animales domésticos y silvestres de la región que son susceptibles o reservorios de la enfermedad.
Sistemas de explotación
En el sistema de explotación los productores optan en su mayoría por un sistema extensivo de crianza de ovinos en la Región San Martín (Cuadro 3).
Cuadro 3: Sistemas de producción
Provincias | Sistemas de explotación | ||
Intensivo | Extensivo | Semi Intensivo | |
Moyobamba | 0 | 9 | 0 |
Rioja | 0 | 11 | 0 |
El Dorado | 0 | 35 | 0 |
Lamas | 0 | 27 | 14 |
Bellavista | 0 | 74 | 0 |
Picota | 0 | 29 | 12 |
Mariscal Cáceres | 0 | 15 | 0 |
Huallaga | 0 | 19 | 5 |
Tocache | 0 | 20 | 10 |
San Martin | 33 | 15 | 38 |
|
| Total | 366 |
Conclusiones e implicaciones
Se concluye que el VLA tiene una baja prevalencia en ovinos en la Región San Martin del Perú, sin embargo, es necesario estudios futuros para determinar la morbilidad y la detección de otros serotipos potenciales de VLA en el país, para dilucidar mejor el manejo de vectores y las estrategias de control de la enfermedad.
Agradecimientos
El presente trabajo de investigación recibió el apoyo financiero del Instituto de investigación y Desarrollo de la UNSM–T, concurso de proyectos de tesis de pregrado, periodo 2018, con Resolución N° 611 -2018 – UNSM/CU – R/ NLU.
Literatura citada: