https://doi.org/10.22319/rmcp.v11i3.5516
Artículo
Linfonodos y carne molida de res como reservorios de Salmonella spp. de importancia en salud pública
Tania Palós Gutiérrez a
Maria Salud Rubio Lozano a*
Enrique Jesús Delgado Suárez b
Naisy Rosi Guzmán a
Orbelin Soberanis Ramos b
Cindy Fabiola Hernández Pérez c
Rubén Danilo Méndez Medina d
a Universidad Nacional Autónoma de México (UNAM). Facultad de Medicina Veterinaria y Zootecnia (FMVZ), Centro de Enseñanza Práctica, Investigación en Producción y Salud Animal, Avenida Universidad 3000, Ciudad Universitaria, Ciudad de México, México.
b UNAM, FMVZ. Departamento de Medicina Preventiva y Salud Pública. Ciudad de México. México.
c Servicio Nacional de Sanidad, Inocuidad y Calidad Agroalimentaria. Departamento de Secuenciación y Bioinformática del Centro Nacional de Referencia de Plaguicidas y Contaminantes. Ciudad de México, México.
d UNAM, FMVZ. Departamento de Patología. Ciudad de México. México.
* Autor de correspondencia:msalud65@gmail.com
Resumen:
El objetivo fue estimar la frecuencia de contaminación, diversidad de serotipos y tipificación por multilocus (MLST) de Salmonella enterica (SE) en linfonodos y carne molida de bovino. Para ello, se analizaron 1,545 muestras provenientes de 400 canales bovinas. El muestreo incluyó linfonodos superficiales (LNS) y profundos, carne molida magra y con grasa, obtenidas en estación cálida (abril-julio) y fría (septiembre-diciembre) durante 2017 y 2018. Los aislamientos puros se sometieron a secuenciación completa del genoma. Con estos datos, se realizó la predicción in silico de serotipos y del perfil de MLST. En total, se obtuvieron 78 aislamientos de SE (5 % del total de muestras analizadas). La frecuencia de contaminación se asoció con el tipo de muestra ( 2=23.7, P<0.0001) y con la época del año (
2=23.7, P<0.0001) y con la época del año ( 2=20.3, P<0.0001), siendo mayor en LNS (9.7 %) y en estación cálida (7.0 %). Los serotipos predominantes fueron Anatum y Reading (n=23 cada uno), Typhimurium (n= 11) y London (n= 9). El perfil de MLST de cepas de los serotipos Typhimurium (ST 19 y 34) y Kentucky (ST 198) se ha reportado previamente en aislamientos involucrados en casos clínicos. Se concluye que los linfonodos y la carne molida de res son reservorios de SE de importancia en salud pública, especialmente durante los meses cálidos del año. Por tanto, es necesario establecer medidas encaminadas a prevenir la diseminación, a lo largo de la cadena productiva, de las cepas asociadas con animales aparentemente sanos.
2=20.3, P<0.0001), siendo mayor en LNS (9.7 %) y en estación cálida (7.0 %). Los serotipos predominantes fueron Anatum y Reading (n=23 cada uno), Typhimurium (n= 11) y London (n= 9). El perfil de MLST de cepas de los serotipos Typhimurium (ST 19 y 34) y Kentucky (ST 198) se ha reportado previamente en aislamientos involucrados en casos clínicos. Se concluye que los linfonodos y la carne molida de res son reservorios de SE de importancia en salud pública, especialmente durante los meses cálidos del año. Por tanto, es necesario establecer medidas encaminadas a prevenir la diseminación, a lo largo de la cadena productiva, de las cepas asociadas con animales aparentemente sanos.
Palabras clave: Salmonella, Bovinos, Linfonodos, Carne molida, Serotipos, MLST.
Recibido: 21/09/2019
Aceptado:25/11/2019
Introducción
La salmonelosis transmitida por alimentos es una preocupación de salud pública a escala global(1). La carne de diferentes especies, entre ellas la de bovino, funciona como reservorio de su principal agente causal: Salmonella enterica subsp. enterica, de aquí en adelante referida como Salmonella(2). Particularmente, la carne de res molida se ha vinculado con brotes recientes de salmonelosis en América del Norte(3), por lo cual se considera uno de los principales vehículos de exposición humana a Salmonella. En México se han reportado porcentajes de muestras positivas de entre 16 y 68 % en carne molida en puntos de venta(4,5), por lo cual la investigación en esta área resulta relevante desde el punto de vista de salud pública.
Evidencia experimental reciente documenta el aislamiento de Salmonella a partir de nódulos linfáticos de bovinos aparentemente sanos, en frecuencias variables de entre <10 y hasta >90 %(6,7). Asimismo, se ha demostrado que los linfonodos superficiales presentan mayores tasas de contaminación que los profundos; mientras que el número de animales con linfonodos contaminados es mucho mayor en ganado de engorda comercial que en los de desecho(7,8). No obstante, los resultados suelen variar ostensiblemente según la zona geográfica y la época del año, un fenómeno determinado por mecanismos aun sin descifrar.
En trabajos con cepas de Salmonella obtenidas de animales de desecho, la tipificación de aislamientos, por electroforesis en gel de campos pulsados, demostró clonalidad entre cepas de linfonodos y las presentes en carne molida producida a partir de estos animales(9). Sin embargo, este tipo de estudios no se ha realizado en animales de engorda comercial.
A pesar de las altas tasas de positividad a Salmonella reportadas en muestras bovinas en México(4-6), la contribución de los linfonodos a este fenómeno prácticamente no ha sido abordada. Por tanto, el objetivo del presente estudio es estimar la frecuencia de contaminación y la diversidad de serotipos de Salmonella presentes en nódulos linfáticos, así como en carne y grasa asociada con estos últimos, en diferentes épocas del año.
Material y métodos
Diseño del estudio y determinación del tamaño de muestra
El tamaño de muestra se calculó mediante la fórmula estadística para estimar una proporción de una población, cuando no se conoce el número de elementos en esa población(10):
 ;
;
Donde: n= tamaño de muestra; Zα2= Valor de Z en una distribución normal Zα= 1.96 cuando α= 0.05; p= proporción de la población con la característica estudiada (si se desconoce se emplea 0.5, como en este caso); q= proporción de la población sin la característica estudiada(1-p); d= error o precisión deseada, fijado en 10% (0.1).
Bajo estos supuestos, se obtuvo un tamaño de muestra de 96, el cual se redondeó a 100. El muestreo se replicó dos veces por año, durante dos años consecutivos, con dos etapas en cada año. Las muestras tomadas entre abril y julio se designaron como muestras de estación “cálida” y las recolectadas entre septiembre y diciembre como muestras de estación “fría”.
Las canales provenían de bovinos machos enteros, cruzas de Bos indicus, con una edad promedio de 24 a 36 meses, procesadas en un rastro Tipo Inspección Federal del estado de Veracruz y trasportadas en refrigeración (<4 ºC), durante aproximadamente 8 h, hasta un punto de venta de la Ciudad de México. Una vez allí, las canales se mantuvieron en refrigeración durante dos días hasta la recolección de las muestras (72 a 96 h post mortem). El punto de venta se visitó los lunes y martes de cada semana, hasta completar entre 5 y 10 canales por semana, según la cantidad de canales disponibles.
Toma de muestras
De cada canal se extrajeron linfonodos superficiales (LNS, cervical superficial y subilíaco) y profundos (LNP, axilar propio y celíaco). La selección de linfonodos se hizo tomando en cuenta la probabilidad de que fueran incluidos en el proceso de molienda, por su ubicación anatómica. Además de los linfonodos, se tomaron aproximadamente 200 g de carne magra (CM, 50 % del diezmillo y 50 % del sirloin, por ser los cortes más usados para producir carne molida) y de carne con grasa (CG), de las zonas circundantes de los LNS y LNP (aproximadamente 50 % de cada una). Previo a los análisis, las porciones individuales de cada tipo de muestra se combinaron para formar una sola muestra compuesta. En algunas ocasiones, ciertas partes de la canal estaban comprometidas para la venta y no estuvieron disponibles para el muestreo. Por ello, no fue posible obtener todos los tipos de muestra del 100 % de las canales. Por consiguiente, la unidad de muestreo se definió como la muestra compuesta de LNS, LNP, CM y CG. En total, se recolectaron 1,545 muestras de todas las fuentes en los dos años de duración del estudio (Cuadro 1).
Cuadro 1: Distribución de las 1,545 muestras de carne y de linfonodos analizadas por estación y año entre el 3 de abril de 2017 y el 14 de diciembre de 2018
Tipo de muestra | Estación cálida |
|
| Estación fría |
|
2017 | 2018 | Total |
| 2017 | 2018 | Total |
LNS | 168 | 98 | 266 |
| 33 | 102 | 135 |
LNP | 166 | 98 | 264 |
| 33 | 102 | 135 |
CM | 130 | 98 | 228 |
| 33 | 102 | 135 |
CG | 149 | 98 | 247 |
| 33 | 102 | 135 |
Total |
|
| 1,005 |
|
|
| 540 |
Estación cálida: abril-julio, estación fría: septiembre-diciembre.
LNS= linfonodos superficiales, LNP= linfonodos profundos, CM= carne magra, CG= carne con grasa.
Las porciones individuales de cada tipo de muestra se colocaron en bolsas plásticas estériles, previamente identificadas y se mantuvieron en hieleras con geles refrigerantes (aproximadamente a 4º C), durante su traslado al laboratorio (duración de dos horas como máximo).
Análisis microbiológicos
La preparación de las muestras de linfonodos se realizó según métodos descritos previamente(11), con algunas modificaciones. Los linfonodos se pesaron y, posteriormente, se sumergieron en agua hirviendo durante 5 seg, con el fin de esterilizar su superficie. A continuación, se les agregó la mitad del agua peptonada tamponada (APT) necesaria para alcanzar una dilución aproximada de 1:10 (8 g de LNP en 80 ml de APT y 25 g de LNS en 225 ml de APT) y se molieron durante 3 seg en una licuadora Oster previamente esterilizada. Las muestras molidas se vaciaron en una bolsa Stomacher®, previamente identificada, y con el resto del APT se arrastró el remanente contenido en el vaso de la licuadora, asegurando la transferencia de toda la muestra a la bolsa Stomacher®, realizando posteriormente la homogenización de la mezcla durante 1 min.
Para el análisis de las muestras de carne magra (CM), se tomaron 25 g y se molieron en una licuadora Oster estéril durante 30 seg. Posteriormente, el contenido se colocó en una bolsa de Stomacher® previamente identificada y se agregaron 225 ml de APT, homogenizando la mezcla durante 1 min. Por último, las muestras de carne con grasa (CG) se prepararon moliendo de manera conjunta en una licuadora Oster estéril, durante 30 seg, aproximadamente 1/3 de grasa y 2/3 de carne de las zonas circundantes de LNS y LNP (50 % proveniente de cada tipo de linfonodo). Después de la molienda se pesaron asépticamente 25 g y se siguió el mismo procedimiento descrito para la carne magra.
Los homogenatos se dejaron reposar durante 2 h a temperatura ambiente, antes de seguir los procedimientos de pre-enriquecimiento, enriquecimiento selectivo, aislamiento y confirmación bioquímica de Salmonella spp., establecidos en la norma oficial mexicana vigente(12). También se realizó la confirmación molecular de los aislamientos presuntamente positivos a Salmonella spp., mediante PCR, utilizando el gen invA (284 pb), según métodos descritos previamente(13). El ADN se extrajo con el kit “Ez-10 Spin Column Bacterial Genomic DNA Miniprep Kit” (BioBasic, Inc., Canadá), siguiendo las instrucciones del proveedor, a partir de las cepas puras, previamente refrescadas en caldo tripticasa soya (MCD Lab®, PRONADISA-CONDA®, España) por 18-24 h. Se utilizaron los cebadores sentido (CGCCATGGTATGGATTTGTC) y contrasentido (GTGGTAAAGCTCATCAAGCG), en reacciones de PCR con un volumen total de 10 μl, empleando los reactivos del MyTaqTM Mix (Bioline, U. K.) con las siguientes concentraciones finales: 5 μl de MyTaqTM Mix, 0.2 μl de cada dNTP y 2.1 μl de agua libre de nucleasas. Las condiciones de termociclado fueron: 94 ºC/3 min de desnaturalización inicial; posteriormente, 35 ciclos de desnaturalización, alineación y extensión (95ºC/45 seg, 62ºC/30 seg, 72ºC/45 seg, respectivamente) y 72ºC/2 min de extensión final. Los productos de amplificación por PCR se procesaron por electroforesis en gel de agarosa (SeaKem® LE Agarose, Lonza, EEUU) al 2%. Los geles se corrieron en buffer tris/borato/EDTA (TBE 1x), a 80 V por 50 min, utilizando SYBR Safe DNA Gel Stain (Invitrogen, USA) para revelar los fragmentos de ADN. La visualización y digitalización de imágenes se realizó en un fotodocumentador Gel Logic 2200 (Kodak, USA) con el software Care Stream® (Carestream Health, Inc., USA). En cada corrida se incluyó una cepa de S. enterica subsp. enterica ser. Typhimurium, del propio laboratorio, previamente confirmada por métodos bioquímicos, PCR y secuenciación completa del genoma. Los aislamientos confirmados se conservaron de dos modos. En el primero, se elaboraron inóculos de 1 ml tomando colonias frescas y mezclándolas en caldo infusión-cerebro-corazón (Merck, Germany) con 10% de glicerol y se mantuvieron a – 70 °C en un ultracongelador. Por otra parte, se realizó un respaldo de los aislamientos en agar soya tripticaseina (TSA, PRONADISA-CONDA®, España), almacenado a temperatura ambiente.
Predicción de serotipos y tipificación por multi-locus (MLST)
El serotipo de las cepas obtenidas se predijo a partir de los datos de secuenciación completa del genoma (lecturas crudas). El ADN genómico se extrajo a partir de colonias refrescadas en caldo TSA con agitación a 37 ºC durante aproximadamente 18 h. Transcurrido este tiempo, se tomó 1 ml de caldo TSA y se centrifugó a 5,000 xg durante 10 min para obtener un pellet de células. Posteriormente, se utilizó el kit de extracción “High Pure PCR Template Preparation Kit” (Roche Molecular Systems, Inc., Switzerland), según instrucciones del fabricante, para obtener el ADN genómico.
La secuenciación se realizó en un equipo Illumina NextSeq (Illumina, USA), utilizando el kit Nextera XT versión 3 (Illumina, USA) para preparar la biblioteca de ADN, con un inserto de 150 pb y una profundidad estimada mínima de 30X. Las lecturas crudas así obtenidas se utilizaron para predecir el serotipo, mediante análisis in silico, con ayuda del programa SeqSero(14). Por último, se realizó la tipificación por multilocus (MLST), basada en siete genes esenciales (aroC, dnaN, hemD, hisD, purE, sucA)(15), en el servidor del Centro de Epidemiología Genómica(16). Como la tipificación por MLST se ha empleado por décadas y existe una base de datos de acceso público(17), es posible estimar la importancia epidemiológica de los aislamientos, mediante la comparación con las ST reportadas previamente en muestras clínicas de humanos y de animales. Además, el perfil de alelos se utilizó para generar un árbol filogenético de mínima expansión, con ayuda del programa GrapeTree(18), para analizar la diversidad de ST en la muestra bajo estudio.
Análisis estadístico
Se utilizó la prueba de Ji-cuadrada para determinar si existía asociación entre el tipo de muestra, la época del año y el serotipo de Salmonella con la frecuencia de contaminación. En caso de observar asociación significativa, se utilizó la razón de probabilidades para estimar los factores con mayor influencia en la tasa de contaminación de las diferentes matrices estudiadas. Los datos se analizaron con el programa Statgraphics Centurion XV (StatPoint, Technologies, USA).
Resultados
En general, se observó una frecuencia de contaminación con Salmonella spp. del 5.0 %, con 78 aislamientos obtenidos a partir de las 1,545 muestras analizadas en los dos años (Figura 1). Se evidenció una fuerte asociación entre el tipo de muestra y la positividad al patógeno ( 2=23.7, P<0.0001), con una mayor probabilidad de encontrar muestras positivas en LNS que en las demás fuentes (razón de probabilidades 3.2, intervalo de confianza al 95% 2.0-5.0, P<0.0001).
2=23.7, P<0.0001), con una mayor probabilidad de encontrar muestras positivas en LNS que en las demás fuentes (razón de probabilidades 3.2, intervalo de confianza al 95% 2.0-5.0, P<0.0001).
Figura 1: Frecuencia de contaminación con Salmonella spp. en muestras de carne magra (n=363), carne con grasa (n=382), linfonodos profundos (n=399) y linfonodos superficiales (n=401) de bovino, recolectadas entre abril de 2017 y diciembre de 2018
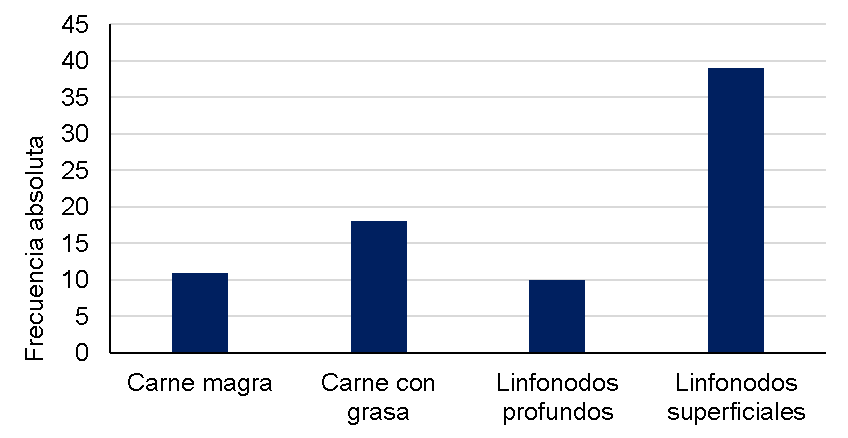
También se observó una asociación significativa entre la frecuencia de contaminación y la época del año ( 2=20.3, P<0.0001). La probabilidad de encontrar muestras positivas en la estación cálida fue mucho más alta que en la fría (razón de probabilidades 4.7, intervalo de confianza al 95% 2.2-9.8, P<0.0001) (Figura 2).
2=20.3, P<0.0001). La probabilidad de encontrar muestras positivas en la estación cálida fue mucho más alta que en la fría (razón de probabilidades 4.7, intervalo de confianza al 95% 2.2-9.8, P<0.0001) (Figura 2).
Figura 2: Frecuencia de contaminación con Salmonella spp. en muestras de carne magra (n=363), carne con grasa (n=382), linfonodos profundos (n=399) y linfonodos superficiales (n=401) de bovino, recolectadas entre abril de 2017 y diciembre de 2018

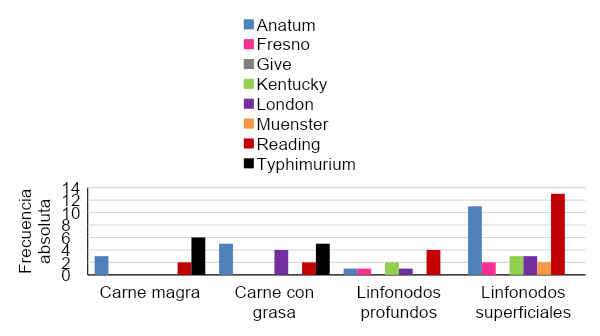
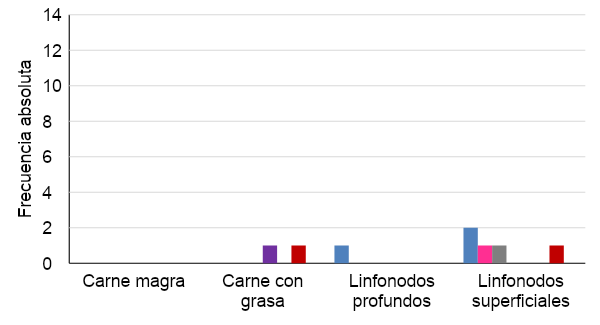
El serotipo de Salmonella también estuvo asociado con el tipo de muestra ( 2=43.8, P=0.0025). Salmonella typhimurium sólo se detectó en muestras de carne de la estación cálida. Por el contrario, los serotipos Muenster (n=2) y Kentucky (n=5) sólo se presentaron en linfonodos, también de la estación cálida (Figura 3). Asimismo, la única cepa del serotipo Give se aisló a partir de LNS en la estación fría. Aunque la diversidad de serotipos fue mayor en ambos tipos de linfonodos que en carne magra o con grasa, en general, las cepas de los serotipos Reading y Anatum (n=23 de cada una), Typhimurium (n=11) y London (n=9), fueron las predominantes.
2=43.8, P=0.0025). Salmonella typhimurium sólo se detectó en muestras de carne de la estación cálida. Por el contrario, los serotipos Muenster (n=2) y Kentucky (n=5) sólo se presentaron en linfonodos, también de la estación cálida (Figura 3). Asimismo, la única cepa del serotipo Give se aisló a partir de LNS en la estación fría. Aunque la diversidad de serotipos fue mayor en ambos tipos de linfonodos que en carne magra o con grasa, en general, las cepas de los serotipos Reading y Anatum (n=23 de cada una), Typhimurium (n=11) y London (n=9), fueron las predominantes.
Figura 3: Número de aislamientos de S. enterica subsp. enterica por serotipo y fuente de aislamiento en las estaciones cálida (a) y fría (b)


La tipificación por MLST mostró que los aislamientos de cada serotipo correspondieron a una misma ST (Figura 4). La excepción fue Salmonella typhimurium, la cual presentó dos ST (19 y 34). Sin embargo, ambos ST sólo se diferenciaron en el alelo dnaN. Por tanto, satisfacen el criterio para ser considerados como complejo clonal, al coincidir en seis de los siete alelos incluidos en el esquema de tipificación por MLST(15).
Figura 4: Árbol de mínima expansión generado a partir del perfil de MLST de 78 aislamientos de S. enterica subsp. enterica.
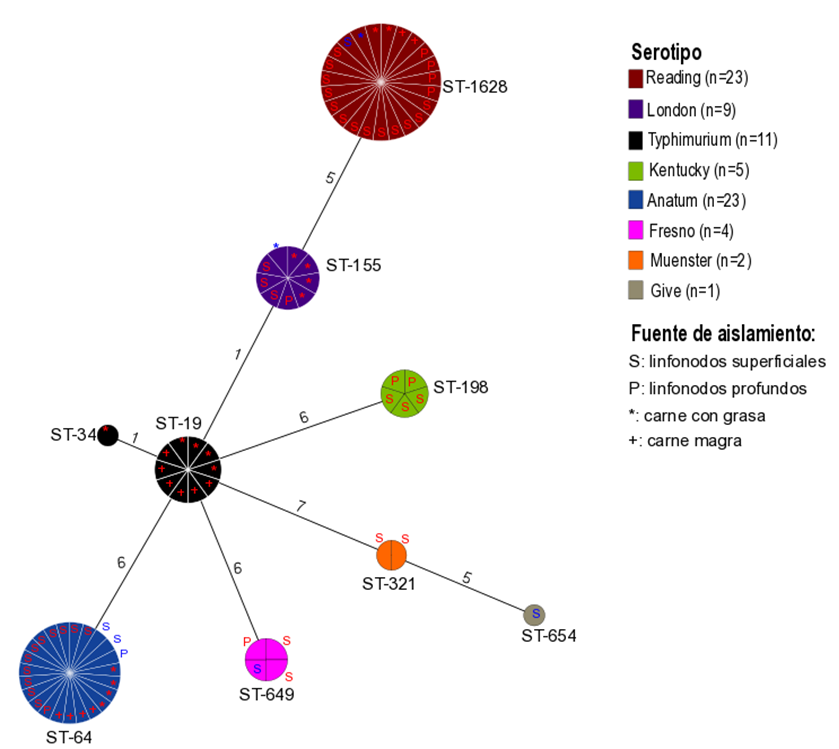
Cada círculo corresponde a un ST y las divisiones dentro de un mismo círculo corresponden a un aislamiento. Los números en las ramas del árbol indican la cantidad de alelos con secuencias distintas entre ST. Los serotipos se indican mediante código de colores y la fuente de aislamiento se señala al interior o adyacente a cada círculo (en texto rojo, si provienen de estación cálida; o en azul, si son de estación fría).
Discusión
Las frecuencias de positividad al patógeno aquí observadas (2.5 a 9.7 %), son inferiores a las reportadas en otros trabajos con carne molida (16-68 %)(4,5) y con linfonodos (50-94 %)(6,19,20). No obstante, la variabilidad de este fenómeno entre zonas geográficas y épocas del año está bien documentada(20,21). En general, el estudio confirma la importancia de los bovinos aparentemente sanos como reservorio de diversos serotipos de Salmonella de importancia epidemiológica. Así lo demuestra la detección de cepas del serotipo Typhimurium, ST 19 y 34, las cuales se asocian con casos clínicos humanos y con la cepa DT104, ampliamente distribuida a escala global(22). De igual forma, los aislamientos del serotipo Kentucky (ST 198) se han asociado con infecciones en humanos y animales en Estados Unidos(23). Estos hallazgos resaltan la necesidad de continuar investigando las poblaciones de Salmonella de origen no clínico, asociadas con producción animal, por su papel como reservorio de infecciones humanas.
Los resultados apoyan también observaciones previas sobre las mayores tasas de positividad al patógeno en linfonodos superficiales, especialmente en condiciones de clima cálido(7,8). Aunque no se han descifrado los factores ambientales causantes de esta variabilidad, la mayor incidencia de picaduras de moscas y de otros insectos en el verano se ha sugerido como un factor condicionante de esta variación estacional(19,24). No obstante, la escasa evidencia experimental relacionada con este factor no proviene de contextos naturales sino de estudios de desafío con moscas artificialmente infectadas con Salmonella.
Los esfuerzos realizados hasta el momento para prevenir las infecciones asintomáticas con Salmonella en el ganado han sido infructuosos. El uso de vacunas basadas en genes involucrados en la captación de hierro, mineral con un papel central en el proceso infeccioso, no tuvo efecto alguno sobre la frecuencia de contaminación en linfonodos de bovinos de engorda(25). Tal hallazgo es poco sorprendente, considerando la redundancia funcional de Salmonella, la cual posee multiplicidad de genes en materia de captación y transporte de hierro (ej. iroBCDE, fepBCDEG, fhuBCD, exbBD, sitD y tonB)(26).
Por otra parte, la supervivencia intracelular de la bacteria, internalizada en vacuolas de células eucariotas(27), como los macrófagos, sugiere que la suplementación con antibióticos es una estrategia poco factible. En este sentido, la administración de concentraciones crecientes de tilosina en la dieta de ganado Holstein, previamente inoculado con el patógeno, no mostró efecto alguno, pues se continuó recuperando Salmonella a partir de los linfonodos de los animales tratados(28).
Aparentemente, la redundancia funcional de Salmonella y sus mecanismos de supervivencia intracelular indican que la eventual eliminación del patógeno dependerá en última instancia del sistema inmune del hospedero. En animales inoculados experimentalmente con cepas del serotipo Montevideo, la eliminación total de la bacteria tomó cerca de un mes(29). En este contexto, el monitoreo de infecciones subclínicas por Salmonella en granjas de engorda, una medida escasamente aplicada, podría funcionar como método para segregar animales portadores y, de esta forma, limitar la diseminación del patógeno. Adicionalmente, la presencia de cepas de un mismo serotipo y ST en carne molida y en linfonodos, observada en este estudio, sugiere a la remoción de los linfonodos como una estrategia para reducir drásticamente la frecuencia de contaminación con Salmonella en carne de res molida. Esta medida es relativamente fácil de realizar a escala industrial, aunque sólo para los linfonodos superficiales, no para los profundos. No obstante, son precisamente los linfonodos superficiales los de mayor relevancia epidemiológica. Por tanto, el establecimiento de esta medida como obligatoria en la normatividad nacional podría funcionar como estrategia para mitigar los riesgos asociados con la presencia de Salmonella en carne de res molida.
Por otra parte, resulta interesante analizar por qué algunos serotipos sólo se presentaron en muestras de carne (ej. Typhimurium), mientras que otros se detectaron en todas las matrices (ej. Anatum y Reading). Particularmente, el serotipo Anatum se ha reportado previamente como cepa predominante en muestras de origen no clínico, sobre todo en linfonodos(19,20). Estas evidencias llevan a razonar sobre la posibilidad de que algunas cepas de Salmonella estén mejor adaptadas para colonizar y sobrevivir en ciertos nichos ecológicos. En el contexto del presente trabajo, sin embargo, es difícil determinar si la representación relativa de serotipos en los linfonodos depende de factores genéticos específicos. Además, se requiere emplear análisis de mayor poder discriminatorio que MLST, para explorar con mayor precisión las relaciones filogenéticas intra e inter-serotipo, así como la dinámica evolutiva de estas poblaciones. Este tema será materia de futuras contribuciones en el área de genómica comparativa.
Conclusiones e implicaciones
El estudio demuestra que los linfonodos y la carne de res molida, provenientes de animales aprobados para la matanza, funcionan como reservorios de cepas de Salmonella enterica de importancia clínica en humanos. Por tanto, es preciso establecer medidas de control para prevenir la diseminación de este patógeno a lo largo de la cadena productiva.
Agradecimientos y conflictos de interés
Esta investigación fue financiada por la Universidad Nacional Autónoma de México, a través del proyecto PAPIIT IN212817. Se agradece la colaboración del Centro Nacional de Referencia de Plaguicidas y Contaminantes por su apoyo en la secuenciación de los aislamientos y en los análisis bioinformáticos preliminares. También agradecemos la colaboración de los alumnos Tavata Meneses, Rosaurora Medina y Abril Viridiana García, así como del profesor Francisco Ruíz, de la Facultad de Medicina Veterinaria y Zootecnia de la UNAM, por su apoyo en las actividades de muestreo y análisis de laboratorio. Ninguno de los autores tiene conflicto de intereses con respecto a la presente publicación.
Literatura citada:
- WHO. WHO estimates of the global burden of foodborne diseases. Foodborne disease burden epidemiology reference group 2007-2015. World Health Organization. http://www.who.int/foodsafety/areas_work/foodborne-diseases/ferg/en/. Accessed Oct 25, 2018.
- Wilhelm BJ, Young I, Cahill S, Desmarchelier P, Nakagawa R, Rajic A. Interventions to reduce non-typhoidal Salmonella in pigs during transport to slaughter and lairage: Systematic review, meta-analysis, and research synthesis based infection models in support of assessment of effectiveness. Prev Vet Med 2017;145:133-144.
- CDC. Outbreak of Salmonella Infections Linked to Ground Beef. Centers for Disease Control and Prevention (CDC) https://www.cdc.gov/salmonella/newport-10-18/index.html. Accessed Feb 25, 2019.
- Cabrera-Diaz E, Barbosa-Cardenas CM, Perez-Montano JA, Gonzalez-Aguilar D, Pacheco-Gallardo C, Barba J. Occurrence, serotype diversity, and antimicrobial resistance of Salmonella in ground beef at retail stores in Jalisco state, Mexico. J Food Prot 2013;76(12):2004-2010.
- Ballesteros-Nova N, Rubio-Lozano MS, Delgado-Suárez EJ, Méndez-Medina RD, Braña-Varela D, Rodas Suárez O. Perfil de resistencia a antibióticos de serotipos Salmonella spp. aislados de carne de res molida en la Ciudad de México. Salud Pública México 2016;58(3):1-7.
- Gragg SE, Loneragan GH, Nightingale KK, Brichta-Harhay DM, Ruiz H, Elder JR, et al. Substantial within-animal diversity of Salmonella isolates from lymph nodes, feces, and hides of cattle at slaughter. Appl Environ Microbiol 2013;79(15):4744-4750.
- Webb HE, Brichta-Harhay DM, Brashears MM, Nightingale KK, Arthur TM, Bosilevac JM, et al. Salmonella in peripheral lymph nodes of healthy cattle at slaughter. Front Microbiol 2017;8:2214.
- Gragg SE, Loneragan GH, Brashears MM, Arthur TM, Bosilevac JM, Kalchayanand N, et al. Cross-sectional study examining Salmonella enterica carriage in subiliac lymph nodes of cull and feedlot cattle at harvest. Foodborne Pathog Dis 2013;10(4):368-374.
- Koohmaraie M, Scanga JA, De La Zerda MJ, Koohmaraie B, Tapay L, Beskhlebnaya V, et al. Tracking the sources of Salmonella in ground beef produced from nonfed cattle. J Food Prot 2012;75(8):1464-1468.
- Jekel JF, Katz DL, Elmore JG. Epidemiology, biostatistics, and preventive medicine. Third ed. Philadelphia, PA, United States of America: Saunders Elsevier; 2007.
- Brichta-Harhay DM, Arthur TM, Bosilevac JM, Kalchayanand N, Schmidt JW, Wang R, et al. Microbiological analysis of bovine lymph nodes for the detection of Salmonella enterica. J Food Prot 2012;75(5):854-858.
- SSA. NORMA Oficial Mexicana NOM-210-SSA1-2014, Productos y servicios. Métodos de prueba microbiológicos. Determinación de microorganismos indicadores. Determinación de microorganismos patógenos. http://www.dof.gob.mx/nota_detalle.php?codigo=5398468&fecha=26/06/2015. Consultado 25 Feb, 2019.
- Rahn K, De Grandis SA, Clarke RC, Curtiss R, Gyles CL. Amplification of an invA gene sequence of Salmonella typhimurium by polymerase chain reaction as a specific method of detection of Salmonella. Mol Cell Probes 1992;6:271-279.
- Zhang S, Yin Y, Jones MB, Zhang Z, Deatherage Kaiser BL, Dinsmore BA, Fitzgerald C, Fields PI, Deng X. Salmonella serotype determination utilizing high-throughput genome sequencing data. J Clin Microbiol 2015;53(5):1685-1692.
- Achtman M, Wain J, Weill FX, Nair S, Zhou Z, Sangal V, et al, Group SEMS. Multilocus sequence typing as a replacement for serotyping in Salmonella enterica. PLoS Pathog 2012;8(6):e1002776.
- Larsen MV, Cosentino S, Rasmussen S, Friis C, Hasman H, Marvig RL, et al. Multilocus Sequence Typing of Total Genome Sequenced Bacteria. J Clin Microbiol 2012;50(4):1355-1361.
- Alikhan NF, Zhou Z, Sergeant MJ, Achtman M. A genomic overview of the population structure of Salmonella. PLoS Genet 2018;14(4):e1007261.
- Zhou Z, Alikhan NF, Sergeant MJ, Luhmann N, Vaz C, Francisco AP, et al. GrapeTree: visualization of core genomic relationships among 100,000 bacterial pathogens. Genome Res 2018;28(9):1395-1404.
- Belk AD, Arnold AN, Sawyer JE, Griffin DB, Taylor TM, Savell JW, Gehring KB. Comparison of Salmonella Prevalence Rates in Bovine Lymph Nodes across Feeding Stages. J Food Prot 2018;81(4):549-553.
- Nickelson KJ, Taylor TM, Griffin DB, Savell JW, Gehring KB, Arnold AN. Assessment of Salmonella prevalence in lymph nodes of U.S. and Mexican cattle presented for slaughter in Texas. J Food Prot 2019;82(2):310-315.
- Cetin E, Serbetcioglu T, Temelli S, Eyigor A. Nontyphoid Salmonella carriage, serovar profile and antimicrobial resistance phenotypes in slaughter cattle. J Food Safety 2018;39(2).
- Balleste-Delpierre C, Sole M, Domenech O, Borrell J, Vila J, Fabrega A. Molecular study of quinolone resistance mechanisms and clonal relationship of Salmonella enterica clinical isolates. Int J Antimicrob Agents 2014;43(2):121-125.
- Haley BJ, Kim SW, Pettengill J, Luo Y, Karns JS, Van Kessel JA. Genomic and evolutionary analysis of two Salmonella enterica Serovar Kentucky sequence types Isolated from bovine and poultry sources in North America. PLoS One 2016;11(10):e0161225.
- Olafson PU, Brown TR, Lohmeyer KH, Harvey RB, Nisbet DJ, Loneragan GH, Edrington TS. Assessing transmission of Salmonella to bovine peripheral lymph nodes upon horn fly feeding. J Food Prot 2016;79(7):1135-1142.
- Cernicchiaro N, Ives SE, Edrington TS, Nagaraja TG, Renter DG. Efficacy of a Salmonella siderophore receptor protein vaccine on fecal shedding and lymph node carriage of Salmonella in commercial feedlot cattle. Foodborne Pathog Dis 2016;13(9):517-525.
- Delgado-Suárez EJ, Selem-Mojica N, Ortiz-López R, Gebreyes WA, Allard MW, Barona-Gómez F, Rubio-Lozano MS. Whole genome sequencing reveals widespread distribution of typhoidal toxin genes and VirB/D4 plasmids in bovine-associated nontyphoidal Salmonella. Sci Rep 2018;8(1):9864.
- Fabrega A, Vila J. Salmonella enterica serovar Typhimurium skills to succeed in the host: virulence and regulation. Clin Microbiol Rev 2013;26(2):308-341.
- Holzer K, Weissend C, Huebner K, Metcalf J, Geornaras I, Belk K, Morley P, Martin J. Presence and characteristics of Salmonella enterica recovered from subiliac lymph nodes of beef feedlot cattle enrolled in a randomized clinical trial of dietary additives. Meat Muscle Biol 2017;1(3):131.
- Edrington TS, Loneragan GH, Genovese K, Hanson DL, Nisbet DJ. Salmonella persistence within the peripheral lymph nodes of cattle following experimental inoculation. J Food Prot 2016;79(6):1032-1035.
 2=23.7, P<0.0001) y con la época del año (
2=23.7, P<0.0001) y con la época del año ( 2=20.3, P<0.0001), siendo mayor en LNS (9.7 %) y en estación cálida (7.0 %). Los serotipos predominantes fueron Anatum y Reading (n=23 cada uno), Typhimurium (n= 11) y London (n= 9). El perfil de MLST de cepas de los serotipos Typhimurium (ST 19 y 34) y Kentucky (ST 198) se ha reportado previamente en aislamientos involucrados en casos clínicos. Se concluye que los linfonodos y la carne molida de res son reservorios de SE de importancia en salud pública, especialmente durante los meses cálidos del año. Por tanto, es necesario establecer medidas encaminadas a prevenir la diseminación, a lo largo de la cadena productiva, de las cepas asociadas con animales aparentemente sanos.
2=20.3, P<0.0001), siendo mayor en LNS (9.7 %) y en estación cálida (7.0 %). Los serotipos predominantes fueron Anatum y Reading (n=23 cada uno), Typhimurium (n= 11) y London (n= 9). El perfil de MLST de cepas de los serotipos Typhimurium (ST 19 y 34) y Kentucky (ST 198) se ha reportado previamente en aislamientos involucrados en casos clínicos. Se concluye que los linfonodos y la carne molida de res son reservorios de SE de importancia en salud pública, especialmente durante los meses cálidos del año. Por tanto, es necesario establecer medidas encaminadas a prevenir la diseminación, a lo largo de la cadena productiva, de las cepas asociadas con animales aparentemente sanos.






