https://doi.org/10.22319/rmcp.v11i4.5137
Artículo
Detección molecular de coronavirus bovino asociado al complejo respiratorio bovino en ganado de engorda del valle de Mexicali, Baja California, México
Carolina Orozco-Cabrera a
Gilberto López-Valencia a
Luis Mario Muñoz-Del Real a
Soila Maribel Gaxiola-Camacho b
Nohemí Castro-del Campo b
Sergio Arturo Cueto-González a
José Guadalupe Guerrero-Velázquez a
Kattya Moreno-Torres a
Kelvin Orlando Espinoza-Blandón a
Sergio Daniel Gómez-Gómez a
Enrique Trasviña-Muñoz a
Francisco Javier Monge-Navarro a*
a Universidad Autónoma de Baja California. Instituto de Investigaciones en Ciencias Veterinarias. Km. 3.5 Carretera a San Felipe, Fraccionamiento Campestre, 21386, Mexicali, Baja California, México.
b Universidad Autónoma de Sinaloa. Facultad de Medicina Veterinaria y Zootecnia, Culiacán Sinaloa, México.
* Autor de correspondencia: fmonge@uabc.edu.mx
Resumen:
El complejo respiratorio bovino (CRB) es la principal causa de enfermedad y muerte en el ganado de engorda en todo el mundo. Es un síndrome infeccioso multifactorial provocado por distintos virus y bacterias que disminuyen la eficiencia productiva y ocasionan pérdidas económicas. En México, el CRB se ha reportado en todas las regiones donde se engorda ganado; sin embargo, esos reportes carecen de información sobre la presencia del coronavirus respiratorio bovino (CVB), haciendo necesario contar con herramientas de diagnóstico confiables y precisas para detectar la presencia de CVB en el ganado que se engorda en México, para proponer las medidas sanitarias apropiadas para su manejo clínico. En este trabajo, se desarrolló una plataforma de diagnóstico molecular de PCR en tiempo real (rt-PCR) que amplifica un fragmento de la proteína S del CVB en muestras de exudado nasal. Al aplicar la plataforma rt-PCR para CVB en bovinos de engorda en aparente estado de salud y con signos de enfermedad respiratoria asociados a CRB se encontró que 19/50 (38 %) resultaron positivos, confirmando la presencia de ese virus en el ganado de la región. Los resultados de este trabajo significan el primer reporte sobre la presencia del CVB asociado al CRB en la región ganadera del noroeste de México y sienta las bases para futuras investigaciones sobre papel que juega este virus en la presentación de la patología del CRB en los sistemas de explotación de bovinos de engorda en nuestra región y el país.
Palabras clave: Coronavirus respiratorio bovino, Complejo respiratorio bovino, PCR, Proteína S, Bovinos de engorda.
Recibido: 30/10/2018
Aceptado: 09/09/2019
Introducción
El complejo respiratorio bovino (CRB) se considera la principal causa de enfermedad clínica y muerte en ganado de engorda estabulado en todo el mundo, ocasionando pérdidas económicas para el ganadero(1). Tradicionalmente, el CRB se relaciona con enfermedad producida de forma individual o combinada del virus sincitial respiratorio bovino (VSR), herpes virus bovino tipo 1 (RIB), virus de parainfluenza 3 bovina (PI3) y virus de la diarrea viral bovina (DVB), los cuales desarrollan una infección primaria con signos clínicos leves(2). Asimismo, el CRB se asocia a la presencia de las bacterias Pasteurella multocida, Mannheimia haemolytica, Histophilus somni y Mycoplasma bovis, las cuales actúan como patógenos oportunistas durante condiciones de estrés o infección viral primaria(3,4). Además de los virus mencionados, el coronavirus bovino (CVB) también se ha reportado como agente viral asociado al CRB, ocasionando enfermedad respiratoria y reducción en la ganancia de peso en ganado de engorda(5- 9). Las infecciones producidas por el CVB han sido reportadas en todo el mundo y se considera como una enfermedad endémica en explotaciones de bovinos de leche y bovinos de engorda estabulados(7). La principal vía de transmisión del CVB es a través de la ruta fecal-oral, aunque también se ha demostrado el contagio a través de la vía respiratoria por inhalación de aerosoles que contienen las partículas virales. Cuando el CVB ingresa al tracto gastrointestinal, desencadena un cuadro clínico de diarrea, deshidratación, acidosis e hipoglucemia en animales jóvenes(10). Cuando el CVB ingresa por inhalación, infecta el epitelio respiratorio de cornetes nasales, tráquea y pulmones. La replicación conduce a la eliminación del virus en las secreciones nasales y la enfermedad produce un cuadro con signos clínicos que varían de ausentes a graves, incluyendo depresión, fiebre, conjuntivitis, dificultad respiratoria y tos leve a severa(11).
En México, el CRB se ha reportado en todas las regiones donde se engorda ganado(12,13,14). Sin embargo, esos reportes carecen de información sobre la presencia del CVB en las explotaciones de bovinos de engorda; siendo de la mayor importancia contar con herramientas de diagnóstico que permitan confirmar la presencia del CVB en el ganado. El diagnóstico del CVB se logra utilizando distintas técnicas serológicas(10,15) o aislamiento viral a partir de exudado nasal y biopsias de diferentes tejidos(10,16,17). Recientemente, se ha reportado la aplicación de técnicas moleculares incluyendo PCR convencional (PCR) y en tiempo real (rt-PCR) para la detección de CVB asociado al CRB(6,10,14); sobresaliendo la utilización de plataformas que amplifican el gen que codifica para la proteína S del CVB, la cual representa la estructura viral más importante para la producción de anticuerpos neutralizantes(7,18). El gen de la proteína S es altamente conservado entre las cepas de CVB y ha sido ampliamente utilizado como gen objetivo de pruebas moleculares para diagnóstico de esta enfermedad en el ganado y otras especies animales, incluyendo el hombre(6,19,20).
El presente trabajo tuvo como objetivo el desarrollo y utilización de una plataforma de diagnóstico molecular por rt-PCR para detectar un fragmento del gen que codifica para la proteína S del CVB en muestras de exudado nasal de ganado bovino. Los resultados indican que el sistema rt-PCR es altamente sensible y específico para detectar el CVB y puede ser utilizado en en los sistemas de explotación de bovinos de engorda de la región y del país.
Material y métodos
El presente estudio se realizó en la Unidad de Laboratorios de Diagnóstico (ULADI) del Instituto de Investigaciones en Ciencias Veterinarias de la Universidad Autónoma de Baja California, Campus Mexicali.
Se recolectaron 50 muestras de exudado nasal de bovinos de engorda estabulados, pertenecientes a un sistema de explotación de bovinos tecnificado, ubicado en el valle de Mexicali, Baja California. Las muestras se obtuvieron de animales de nuevo ingreso a corral con menos de 30 días de arribo a la explotación y contaban con 18 meses de edad en promedio. Se colectaron 30 muestras de animales que presentaban descarga nasal, tos, depresión o temperatura corporal mayor de 38.5 °C (Grupo 1), los cuales se clasificaron como enfermos, y 20 muestras de animales aparentemente sanos que no mostraban ninguno de los signos mencionados(21). Las muestras de exudado nasal se colectaron por vía intranasal profunda, utilizando hisopos de dacrón tipo escobetilla. Una vez realizada la toma de muestra, la escobetilla del hisopo se sumergió en un tubo que contenía solución salina de fosfatos (PBS) estéril, pH 7.4, recortando el mango de tal forma que el tubo pudiera ser cerrado para proteger la muestra de posible contaminación, e identificando con número progresivo el grupo correspondiente. Una vez colectadas, las muestras se transportaron al laboratorio para su procesamiento.
Para la extracción de ARN se utilizaron los juegos de reactivos Aurum Total RNA Fibrous Tissue (Bio Rad, Hercules, California, EUA) siguiendo las instrucciones del fabricante. De cada extracción se recuperó ARN en un volumen de 50 µl de la solución de elución suministrada en los juegos de reactivos. El ARN extraído se almacenó en congelación a -20 °C hasta el momento de las pruebas rt-PCR.
Se diseñaron oligonucleótidos a partir de la secuencia del gen que codifica para la proteína S del coronavirus bovino cepa R-AH187 (CVB-S), con número de acceso GenBank EF424620.1. El gen corresponde a una molécula de 4,090 pares de bases publicada en julio de 2016. Se utilizó el programa de diseño de oligonucleótidos Primer3Plus versión 2006-2007, que se encuentra disponible en: http://www.bioinformatics.nl/cgi-bin/primer3plus/primer3plus.cgi. A partir de la secuencia del gen CVB-S se generó el oligonucleótido de banda positiva denominado CVBf con secuencia 5’–CTACTTGGAATAGGAGATTTG-3´, mientras que para el oligonucleótido de banda negativa denominado CVBr, la secuencia seleccionada fue 3´-TACACGGACAGAAATTTGTG-5´; la amplificación de esos oligonucleótidos por rt-PCR genera un producto de 132 pares de bases y contenido de GC de 36 %, con temperatura de disociación (Tm) de 77.0 ºC para ese producto de PCR. Las características de los oligonucleótidos se muestran en el Cuadro 1. Los oligonucleótidos se sintetizaron por GenScript LTD (Piscataway, New Jersey, EUA), y fueron reconstituidos con agua grado biología molecular equivalente a 10 veces el valor de la concentración en nano moles (nM) referida por el fabricante, para obtener una concentración estándar de 100 micro molar (µM). Para las pruebas rt-PCR la concentración de trabajo de los oligonucleótidos se estableció en 10 µM.
Cuadro 1. Secuencias y propiedades de los oligonucleótidos diseñados a partir del gen CVB-S con referencia GenBank EF424620.1
Oligonucleótido: Secuencia: | CVBf CTACTTGGAATAGGAGATTTG |
Inicio: nucleótido 1337 Longitud: 21 pb Tm: 55.4 ºC GC: 38% | |
Oligonucleótido: Secuencia: Fin: nucleótido 1469 Producto: | CVBr TACACGGACAGAAATTTGTG |
Longitud: 20 pb Tm: 54.3 ºC GC: 40% | |
132 pb Tm: 77.0 ºC | |
En este trabajo se utilizó la mezcla maestra I Script One-Step RT-PCR (Bio Rad, Hercules, California, EUA) formulada con el fluoróforo SYBR Green I en una solución de mezcla maestra que utiliza ambos oligonucleótidos a una concentración de 400 nM, 2 µl de ARN templete y agua grado biología molecular para un volumen de reacción total optimizado a 10 µl.
Como control positivo se utilizó ARN extraído de la fracción líquida de la vacuna Scourgard 4 K7C (Zoetis, New Jersey, USA), la cual contiene coronavirus bovino cepa Hansen inactivado, además de rotavirus bovino serotipos G6 y G10 inactivados, E. coli enterotoxigénica K 99 y toxoide de Clostridium perfringens tipo C. El procedimiento para extracción de ARN fue aplicando el protocolo de los juegos de reactivos Bio Rad AurumTotal RNA Fibrous Tissue utilizando 300 µl de la vacuna. El ARN extraído fue dividido en alícuotas de 10 µl y almacenado en congelación a -20° C hasta el momento de ejecutar las pruebas RT-PCR.
Las pruebas rt-PCR se realizaron en un termociclador CFX96 de Bio Rad. Los parámetros de desnaturalización, hibridación y extensión se calcularon empleando la herramienta Protocol Autowriter de la paquetería CFX96 integrada al termociclador, tomando en consideración el tamaño del producto del PCR, la secuencia de los oligonucleótidos y el tipo de enzima de la mezcla maestra, resultando un paso inicial a 50 °C por 10 min para transcripción reversa, seguido de un ciclo de desnaturalización de 95 °C durante 3 min, y continuando con 39 ciclos desnaturalización a 95° C por 10 seg, 20 seg a 51.0 °C para hibridación de oligonucleótidos y 15 seg a 72 °C para extensión. Asimismo, para cada corrida se realizó el análisis de curva de disociación a partir de 65 °C y hasta 95 °C para la identificación de curvas de amplificación dentro de la temperatura estimada de 77.0 °C +/- 1 °C del producto del PCR de 132 pb y discriminar entre artefactos distintos a la amplificación del templete de ARN esperado.
Los resultados de las pruebas rt-PCR para CVB fueron considerados positivos cuando la muestra correspondiente obtuvo una señal fluorescente de amplificación antes del ciclo 40, por encima de la línea umbral de control que el programa CFX96 establece de manera automática, y que corresponde a 10 veces la desviación estándar del promedio de índice de fluorescencia generada por todas las muestras durante los 10 primeros ciclos de cada corrida. Los resultados se consideraron negativos cuando la muestra correspondiente no logró desarrollar una señal fluorescente de amplificación por encima de la línea umbral del control negativo de referencia en un máximo de 40 ciclos.
Resultados
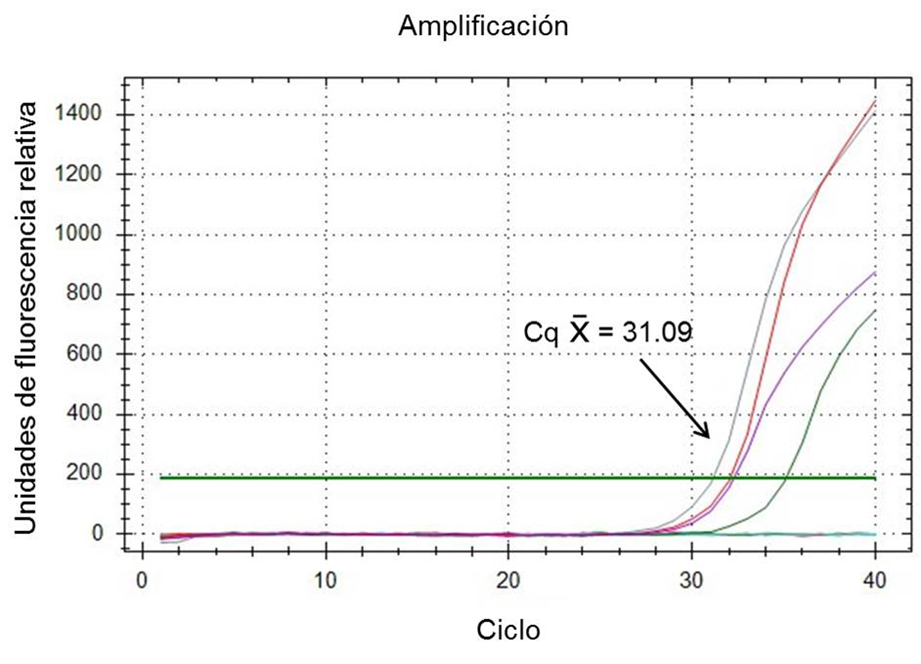
El gráfico de amplificación y la curva de disociación calculados por la paquetería CFX96 de BioRad para el sistema rt-PCR de CVB demostraron que la combinación óptima de reactivos para obtener la máxima amplificación de los oligonucleótidos CVBf y CVBr se logra a una concentración de 400 nM con 2 µl de ARN templete. Bajo estas condiciones los controles positivos extraídos de la vacuna Scourguard 4 K/C desarrollaron una señal por encima de la línea umbral con un ciclo de amplificación (Cq) promedio de 31.09 en 40 ciclos totales de cada corrida de amplificación; los controles negativos no mostraron evidencia de amplificación (Figura 1). Asimismo, el análisis de la curva de disociación (Tm) para el ARN control positivo mostró un rango de temperatura de disociación entre 76.0 y 78.0 °C, con una temperatura promedio de 77.0 °C (Figura 2); estos parámetros permiten considerar a la prueba rt-PCR para CVB como válida.
Se probaron por duplicado 50 muestras de ARN proveniente de exudado nasal de bovinos de engorda estabulados de las cuales 19 (38.0 %) lograron una amplificación por encima de la línea umbral que establece la paquetería CFX96 y por lo tanto fueron consideradas como positivas. De las muestras que resultaron positivas, 5 (10.0 %) pertenecían al Grupo 1, que corresponde a animales con signos y síntomas asociados al CRB y 14 (28.0 %) al Grupo 2, conformado por animales sin signos ni síntomas de enfermedad respiratoria. El Cq promedio de las muestras de ambos grupos fue de 34.60 ciclos con rango de Cq entre 30.87 y 35.95 ciclos y Tm promedio de 77.0 °C (Figura 2).
Discusión
El CVB es un virus patógeno de distribución mundial que produce enfermedades entéricas en becerros jóvenes y disentería de invierno en bovinos adultos. El CVB también está implicado en infecciones asociadas al CRB en ganado de engorda. Aunque las infecciones por CVB producen una mortalidad menor al 2 %, la morbilidad de este virus puede alcanzar el 100 % de los animales de una explotación, ocasionando síndromes respiratorios o digestivos que afectan de forma negativa la tasa de ganancia de peso o de producción de leche, además de los costos por servicios veterinarios, antibióticos y otros medicamentos que en conjunto, ocasionan pérdidas económicas para el sector pecuario(10,15).
El desarrollo e instrumentación de la plataforma rt-PCR para CVB aquí presentada, surge como respuesta a la necesidad de contar con herramientas de diagnóstico confiables, precisas y rápidas para detectar una enfermedad de origen viral que ha sido reportada como parte del CRB pero que debido a la gran cantidad de signos y síntomas que comunes que producen los agentes patógenos, resulta difícil establecer con precisión cuál es el principal agente causal al que puede atribuirse el estado patológico en un animal o hato(22,23); especialmente cuando el proceso infeccioso se desarrolla con síntomas mínimos o imperceptibles, ocasionando retraso en la iniciación de la terapia correspondiente, extendiendo en el tiempo requerido para recuperar el estado de salud y como consecuencia afectando de forma negativa los niveles de productividad de los animales enfermos(20,24).
En este trabajo, se diseñó, desarrolló e instrumentó una plataforma de rt-PCR que detecta y amplifica un fragmento del gen que codifica para la proteína S del CVB, resultando en una plataforma de diagnóstico molecular altamente sensible y específica para la detección de CVB a partir de muestras de exudado nasal. Si bien en este trabajo no se realizó la cuantificación de partículas virales, la sensibilidad de las plataformas rt-PCR para CVB a partir de muestras de exudado nasal ha sido reportada previamente con rangos de detección que van desde 102 copias de cDNA(25) hasta 103 copias de cDNA(26) por reacción, con curvas de amplificación desarrolladas después del ciclo 34 para ambos estudios, lo cual coincide con el Cq promedio de 34.60 desarrollado por las muestras analizadas en este trabajo, por lo que se propone que la plataforma rt-PCR para CVB aquí reportada tiene una sensibilidad estimada entre 102 y 103 copias de cDNA por reacción.
Debido a las propiedades genéticas de la proteína S, caracterizadas por una alta homología ente las distintas cepas virales y su alta reactividad inmunológica(22,24), distintos fragmentos del gen de la proteína S del CVB han sido utilizado como base metodológica de referencia para el desarrollo de plataformas de diagnóstico molecular y serológico para la detección y diagnóstico de este virus de forma rápida y con altos niveles de sensibilidad y especificidad, incluso en muestras que contienen pequeñas cantidades de virus donde las pruebas de diagnóstico convencionales pueden resultar poco concluyentes(23).
Los resultados obtenidos indican que 19 muestras (38 %) provenientes de ambos grupos de estudio resultaron positivas a la plataforma rt-PCR para CVB. Sobresale el hecho que cinco de las muestras del grupo de animales enfermos (n= 30) resultaron positivas, mientras que 14 de las muestras provenientes del grupo de animales aparentemente sanos (n= 20) resultaron positivas a las pruebas. Contrario a lo que se anticipaba, el 70 % de las muestras del grupo de animales aparentemente sanos resultó positivo a la plataforma rt-PCR para CVB. Lo anterior puede deberse a que el CVB puede infectar hasta el 45 % de ganado de reciente arribo a corral sin mostrar signos o síntomas evidentes de enfermedad(27,28); sin embargo, se ha comprobado que la eliminación vía nasal de CVB en ganado aparentemente sano es 1.6 veces más propenso a sufrir al menos un episodio enfermedad respiratoria y 2.2 veces más probabilidad de desarrollar lesiones pulmonares comparado con animales que no eliminan virus por esta vía(7,29), por lo que es posible que esos animales se encontraban incubando el virus y todavía no desarrollaban el cuadro clínico respiratorio característico del CRB.
La prevalencia de 38 % es similar a la reportada en otras regiones del mundo. En un estudio realizado en Australia en ganado de engorda para exportación se analizaron muestras de exudado nasal empleando una plataforma rt-PCR similar a la empleada aquí, encontrando una prevalencia de 40.1 % para CVB, seguido de 0.4 % para DVB, 0.3 % para RIB, 0.3 % para VSR y 0.3 % para PI3, haciendo evidente la magnitud de la influencia del CVB sobre la presentación de enfermedad respiratoria del CRB en ese país(29). Asimismo, los resultados muestran una tasa de positivos superior a la reportada en Irlanda, donde se realizó un estudio para establecer la prevalencia de agentes patógenos asociados al CRB a partir de muestras de exudado nasal empleando rt-PCR, encontrando una tasa de positivos de 22.9 % para CVB, 11.6 % para VSRB, 7.0 % para PI3, 6.1 % para RIB y 5 % para DVB; destacando el hecho que el CVB es el virus asociado al CRB que más frecuentemente se diagnostica en el ganado de engorda de ese país(30). En los Estados Unidos de América, el CVB es el virus asociado al CRB con mayor prevalencia en el ganado que se engorda en ese país. Los reportes de prevalencia de patógenos asociados al CRB en muestras de exudado nasal provenientes de ganado de engorda y analizadas por técnicas rt-PCR reportan una prevalencia de 62.8 % para CVB, seguido de DVB con 15.7 %, IBR con 14.9 %, VSRB con 9.1 % y 8.3 % para PI3(23). La tasa de prevalencia de los EUA supera la reportada para Australia e Irlanda, así como lo reportado para México a través de este trabajo, debido principalmente a que las explotaciones de bovinos de engorda en los EUA alojan cientos de miles de cabezas de ganado en una región determinada, donde el contacto cercano entre animales sanos y enfermos puede favorecer la transmisión y persistencia del CVB entre el ganado(31).
Los resultados aquí presentados significan el primer reporte sobre la presencia del CVB asociado al CRB en la región ganadera correspondiente al noroeste de México y colocan al CVB en la segunda posición de la tabla de prevalencia de virus asociados al CRB detectados previamente en la zona, donde el virus sincitial respiratorio bovino (VSRB) ocupó la primera posición con 80.6 % de prevalencia, seguido del virus de parainfluenza 3 (PI3) con 23.8 %, herpes virus bovino 1 (IBR) con 20.4 % y virus de diarrea viral bovina (DVB) con 11.3 %(15).
Conclusiones e implicaciones
Se concluye que el CVB se encuentra presente en el ganado bovino estabulado del valle de Mexicali, Baja California. La plataforma rt-PCR para CVB aquí reportada es una herramienta de diagnóstico molecular rápida, sensible y específica para detectar el CVB en muestras de exudado nasal proveniente de ganado de engorda estabulado. La prevalencia de 38.0 % para CVB reportada en este trabajo debe ser el punto de partida para futuras investigaciones sobre papel que juega este virus en la presentación de la patología del CRB en los sistemas de explotación de bovinos de engorda en nuestra región y el país.
Los autores de este trabajo declaran que no existe conflicto de interés de ningún tipo.
Literatura citada: