https://doi.org/10.22319/rmcp.v11i4.5124
Artículo
Frecuencia de M. hyopneumoniae, M. hyorhinis y M. hyosynoviae en muestras nasales y de pulmón de cerdos con síntomas de neumonía enzoótica porcina
Rosa Elena Miranda Morales a*
Verónica Rojas Trejo a
Luis Enrique López-Cerino a
Erika Margarita Carrillo Casas b
Rosa Elena Sarmiento Silva a
María Elena Trujillo Ortega c
Rolando Beltrán Figueroa c
Francisco José Trigo Tavera d
a Universidad Nacional Autónoma de México. Facultad de Medicina Veterinaria y Zootecnia, Departamento de Microbiología e Inmunología. UNAM. Ciudad Universitaria, 04519, CDMX, México.
b Hospital General “Dr. Manuel Gea González”, Departamento de Biología Molecular e Histocompatibilidad. CDMX, México.
c Universidad Nacional Autónoma de México, Facultad de Medicina Veterinaria y Zootecnia, Departamento de Cerdos. CDMX, México.
d Universidad Nacional Autónoma de México, Facultad de Medicina Veterinaria y Zootecnia, Departamento de Patología. CDMX, México.
*Autor de correspondencia: roelmimo@yahoo.com.mx
Resumen:
M. hyopneumoniae, M. hyorhinis y M. hyopsynoviae son especies genéticamente relacionadas del género Mycoplasma que afectan la producción porcina. El objetivo de este trabajo fue el aislamiento e identificación por PCR de M. hyopneumoniae, M. hyorhinis y M. hyosynoviae a partir de hisopos nasales y muestras de pulmón de cerdos de diferentes regiones mexicanas para determinar la frecuencia de estas especies y evaluar la PCR como herramienta diagnóstica para NEP. Se incluyeron cerdos de 4 a 8 semanas con diagnóstico clínico de NEP. Se obtuvieron muestras de pulmón e hisopos nasales para el aislamiento de Mycoplasma en medio Friis líquido y se identificaron mediante la PCR especie-específica basada en la subunidad 16S rRNA. El aislamiento se logró en 37.11 % (36/97) muestras. Las tres especies de Mycoplasma se identificaron en muestras de pulmón e hisopos nasales. La co-infección por Mycoplasma se identificó en el 27.77 % (10/36). Los géneros bacterianos asociados a las infecciones por Mycoplasma fueron E. coli, Bordetella, Enterobacter, SCN, Corynebacterium, Pasteurella, Streptococcus, Shigella y Kebsiella. La infección mixta estuvo presente en 26 hisopos nasales (45.61 %) y ausente en pulmones. Se concluyó que la frecuencia de Mycoplasma en las fincas de producción fue mayor a la esperada (40.27 %). También se identificaron otras especies de Mycoplasma involucradas en el desarrollo de la NEP. Por lo tanto, la vigilancia asistida por el aislamiento y las técnicas moleculares pueden ser de gran ayuda para la eliminación de Mycoplasma de las explotaciones porcinas y para los proveedores de pie de cría.
Palabras clave: Micoplasmosis, M. hyopneumoniae, M. hyorhinis, M. hyosynoviae, Neumonía enzoótica porcina.
Recibido: 29/10/2018
Aceptado: 09/09/2019
Introducción
El complejo de enfermedades respiratorias porcinas (CREP) es un problema de salud de importancia para la industria porcina a nivel mundial(1). Se debe a la asociación de infecciones como Mycoplasma, virus del síndrome respiratorio y reproductivo porcino (Por sus siglas en inglés: PRRSV), Circovirus porcino tipo 2 (Por sus siglas en inglés: PCV2), Pasteurella multocida, Actinobacillus pleuroneumoniae, Streptococcus suis, Haemophilus parasuis, Bordetella bronchiseptica y Arcanobacterium pyogenes(1,2). Un factor predisponente es la neumonía enzoótica porcina (NEP), primariamente causada por Mycoplasma hyopneumoniae(3), el cual se adhiere al epitelio respiratorio, daña las células ciliadas de la tráquea, bronquios y bronquiolos(4), y suprime la respuesta inmune del tracto respiratorio superior que favorece el desarrollo del CREP(5,6).
La NEP es una enfermedad respiratoria crónica de alta prevalencia con alta morbilidad y baja mortalidad. Del 30 al 80 % de los animales destinados al abasto presentan lesiones de consolidación típicas(7,8). A lo largo de la vida productiva del cerdo, la prevalencia de M. hyopneumoniae va incrementando hasta llegar a la edad de sacrificio, aun cuando se trate de animales vacunados(9). Las hembras reproductoras son un reservorio que perpetúa la continua circulación de los patógenos respiratorios asociados a NEP(10,11).
La gravedad de la enfermedad difiere entre los hatos, y presenta una alta prevalencia en las granjas porcinas convencionales(12). El signo clínico más significativo de la NEP es una tos crónica, seca y no productiva, que se produce en cerdos de engorda entre las 16 y 22 semanas de edad. La lesión macroscópica principal es una consolidación pulmonar craneo-ventral(5), que se caracteriza histológicamente por una neumonía bronco-intersticial con hiperplasia del tejido linfoide-alveolar (Por sus siglas en inglés: BALT)(13). El principal factor de riesgo para NEP es la transmisión vertical de la cerda al lechón durante la lactancia. Debido a que la vacunación no garantiza la protección(14) y a que M. hyopneumoniae es circulante en animales vacunados(15) y en animales de vida libre como el jabalí, con el que se comparte la vulnerabilidad a M. hyopneumoniae, y puede ser un reservorio(16). La severidad de la enfermedad, al momento del sacrificio, es un predictor de la prevalencia inicial al destete con base en las variables indicativas de infección (promedio de lesiones pulmonares, porcentaje de tejido pulmonar afectado, presencia de M. hyopneumoniae en el epitelio bronquial y seroconversión), ya que entre estas dos variables existe una correlación positiva(17).
La mayoría de las infecciones por Mycoplasma permanecen subclínicas(18) y pueden involucrar otras especies del mismo género bacteriano como M. hyorhinis, un habitante comensal de la mucosa del tracto respiratorio superior y de las amígdalas(19). M hyosynoviae, una especie asociada principalmente a artritis aguda y en menor grado a neumonía supurativa con consolidación pulmonar severa y pleuritis(20,21,22). M. hyopneumoniae, M. hyorhinis y M. hyosynoviae son especies de interés porcino genéticamente relacionadas(23), que pueden ser discriminadas por PCR con base en las regiones hipervariables de la subunidad 16S del género(23,24).
El objetivo de este trabajo fue el aislamiento e identificación por PCR de M. hyopneumoniae, M. hyorhinis y M. hyosynoviae a partir de hisopos nasales y muestras de cerdos de distintas regiones de la República Mexicana, para determinar la frecuencia de estas especies y evaluar la PCR como herramienta diagnóstica de la neumonía enzoótica porcina.
Material y métodos
Animales y colección de muestras
Cerdos de 4 a 8 semanas con diagnóstico de NEP de acuerdo a los signos clínicos y con lesiones gruesas en pulmón (áreas de tejido consolidado de tonos purpura a gris en el lóbulo pulmonar cráneo-ventral) fueron incluidos en este estudio. Se obtuvieron asépticamente 40 muestras de pulmón y 57 hisopos nasales con presión en la pared estructural del tejido(25). La recolección de muestras se realizó en granjas de cuatro regiones de México, de mayo 2015 a enero 2016 (Cuadro 1). Cada muestra se colectó por duplicado para el aislamiento de Mycoplasma y para la bacteriología tradicional. Todos los procedimientos animales fueron aprobados por el Comité Institucional para el Cuidado y Uso de Animales de Experimentación de la Universidad Nacional Autónoma de México (CICUAE), en seguimiento de los estándares éticos internacionales.
Cuadro 1: Regiones de procedencia de las muestras de pulmón e hisopos nasales incluidos en este trabajo
Muestras | Región geográfica | Número de muestras |
40 pulmones | México | 12 |
Veracruz | 28 | |
57 hisopos nasales | Hidalgo Guanajuato | 25 32 |
Total | 97 | |
Aislamiento de Mycoplasma
Para el aislamiento de Mycoplasma, los hisopos nasales se resuspendieron en 2 ml de medio Friis. Las muestras de pulmón se congelaron a -20 °C hasta su seguimiento en el laboratorio. Las muestras de pulmón se procesaron de forma rutinaria por maceración en 3 ml de medio Friis para el aislamiento(18,26,27). De cada muestra macerada, 200 μl de la suspensión en medio Friis se inocularon en 1.8 ml de medio Friis suplementado con suero de cerdo (10 %), suero de caballo (10 %), y penicilina (100 μg/ml) para optimizar la recuperación de M. hyopneumoniae(28), y suplementado con L-arginina (0.05 %) para la recuperación de M. hyosynoviae(29). Posteriormente, se realizaron diluciones seriadas hasta 10−6 y finalmente 10 μl se sembraron en agar Friis(27). Los tubos se incubaron a 37 °C hasta que se observó un cambio de color en el medio, o hasta 30 días, antes de ser descartados. Las muestras positivas fueron aquellas que desarrollaron al menos una unidad de cambio de color, las muestras negativas se consideraron aquellas que después de 30 días no tuvieron cambio de color. Las placas de agar se incubaron a 37 °C con 5 % CO2 durante 1 a 2 semanas. Cada colonia aislada, posteriormente se inoculó en 2 ml de medio Friis e incubada posteriormente. Tras observar el cambio de color, los cultivos se evaluaron para confirmar su pureza y posterior uso para discriminación de la especie por PCR.
PCR especie-específica para la identificación de Mycoplasma
La PCR basada en la subunidad 16S rRNA para la identificación de las tres especies de Mycoplasma fue aplicada a cada uno de los aislamientos. Las cepas de referencia M. hyopneumoniae ATCC 25617, M. hyorhinis ATCC 17981, M. hyosynoviae cepa S-16, y M. bovis Donetta PG45, ambas gentilmente donados por la Universidad de Aarhus, en Aarhus, Dinamarca, se cultivaron en 50 ml de medio, concentradas por centrifugación para la extracción de ADN de acuerdo al protocolo con tiocianato de guanidinio(30). Cada aislamiento fue también procesado para la extracción de ADN y conservado a -70 °C para posterior análisis.
La amplificación de la subunidad 16S rRNA se realizó en un volumen de reacción total de 25 µl conteniendo 0.25 µl de Taq PCR Reaction Mix (Sigma-Aldrich, Austria), 10 pmol de cada iniciador sentido y antisentido (Cuadro 2)(24), y 10 µl de ADN(31). Las condiciones de reacción fueron: desnaturalización inicial a 96 °C, por 5 min, seguido de 30 ciclos de desnaturalización a 94 °C por 45 seg, alineación a 72 °C por 2 min, y extensión a 72 °C por 4 min. ADN de cultivos puros de M. hyopneumoniae ATCC 25617, M. hyorhinis ATCC 17981 y M. hyosynoviae cepa S-16 se aplicaron como controles positivos y M. bovis Donetta PG45, como control negativo.
Cuadro 2: Iniciadores de PCR basados en 16S rRNA de M. hyopneumoniae, M. hyorhinis y M. hyosynoviae
Especie de Mycoplasma | Secuencia (5 -3) | Producto (bp) | Referencia |
M. hyopneumoniae | F 5'-TTC AAA GGA GCC TTC AAG CTT C-3' R 5'-GAC GTC AAA TCA TCA TGC CTC T-3' | 1000 | 30 |
M. hyorhinis | F 5' CGGGATGTAGCAATACATTCAG 3' R 5' GACGTCAAATCATCATGCCTCT 3' | 1129 | 30 |
M. hyosynoviae | F 5' CAGGGCTCAACCCTGGCTCGC 3' R 5' GACGTCAAATCATCATGCCTCT 3' | 585 | Este trabajo Gen Bank No de acceso NR029183.1 |
Resultados
Aislamiento de Mycoplasma
Se colectaron 97 muestras: 40 de pulmón con lesiones típicas de Mycoplasma sugerentes de NEP (Figura 1) y 57 hisopos nasales de porcinos de distintas regiones geográficas de la república mexicana (Cuadro 1). De las muestras de pulmón 22.5 % (9/40) fueron positivas al aislamiento de Mycoplasma spp y 77.5 % (31/40) fueron negativas. De los hisopos nasales 47.36 % (27/57) fueron positivos, y 52.63 % (30/57) fueron negativos. En las muestras positivas, el cambio de color del medio de cultivo, se observó tan temprano como el 5.o día o hasta el 12.o día. En promedio, el cambio de color se observó al 7.o día. Las muestras restantes fueron determinadas negativas después de 30 días sin cambio de color.
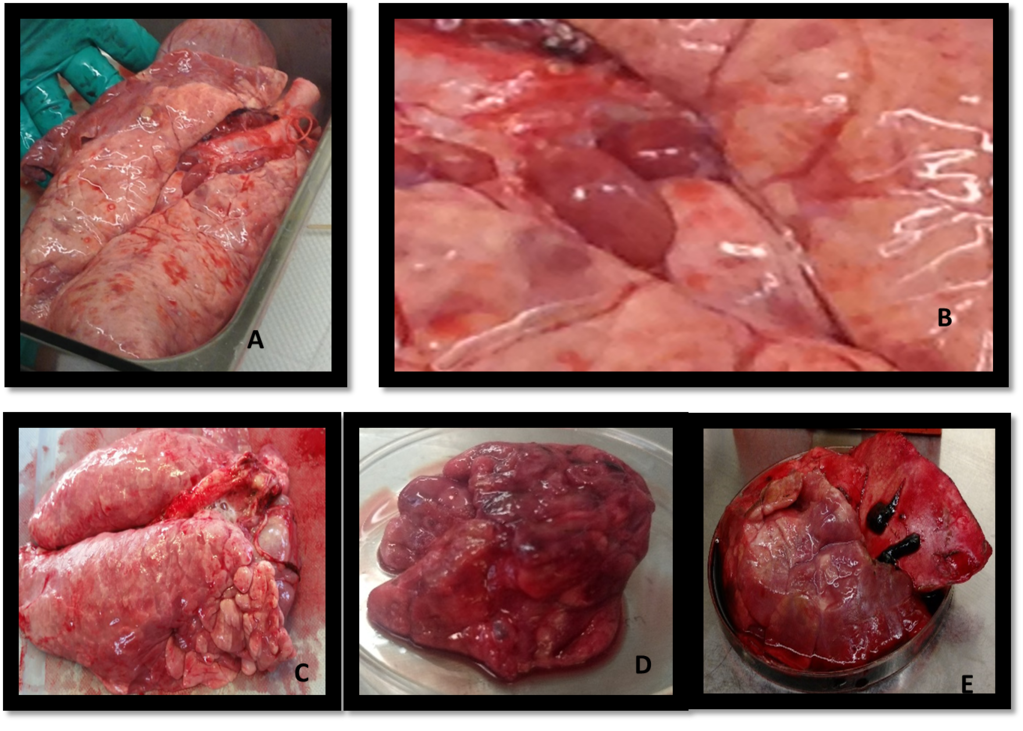
Figura 1: Lesiones típicas de Mycoplasma en los pulmones recolectados para este estudio
En (A) pulmón con lesión neumónica típica de NEP, distribuida en todos los lóbulos pulmonares, (B) acercamiento a la consolidación pulmonar, (C) Pulmón con mayor grado de consolidación pulmonar, (D) Secuestro pulmonar resultante de la evolución de la lesión, (E) Evidencia de cicatrices en el tejido pulmonar.
Resultados de PCR
Los fragmentos amplificados de 1,000 pb de M. hyopneumoniae, 1,129 pb de M. hyorhinis, y 585 pb de M. hyosynoviae empleando las cepas de referencia (ATCC 25617, ATCC 17981, y M. hyosynoviae cepa S-16), se visualizaron por electroforesis en gel de agarosa al 1.5 % a 80 V por 60 min, teñidos con bromuro de etidio y visualizados en un transiluminador UV, como se muestra en la Figura 2. Los aislamientos de muestras de pulmón (AMP) de Mycoplasma fueron positivas en 22 % (2/9) a M. hyopneumoniae, 55.5 % (5/9) a M. hyorhinis y 44 % (4/9) a M. hyosynoviae. Los AMP negativos a la PCR especie-específica fueron 44 % (4/9). Los aislamientos de hisopo nasal (AHN) fueron positivos en 7.40 % (2/ 27) a M. hyopneumoniae, en 51.85 % (14/27) a M. hyorhinis y en 33.3 % (9/27) a M. hyosynoviae. Los AHN negativos a la PCR especie-específica fueron 22.22 % (6/27) (Cuadro 3). A pesar de obtenerse el aislamiento, cuatro AMP y seis AHN no fueron identificados con la PCR especie-específica.
Figura 2: Perfiles electroforéticos de los fragmentos amplificados de M. hyopneumoniae, M. hyorhinis 16S rRNA
Carril 1, Marcador de peso molecular (1 Kb plus Invitrogen), Carril 2, M. hyopneumoniae ATCC 25617, 1000 pb; Carril 3. M. bovis Donetta PG45, donada por la Universidad de Aarhus, Dinamarca, Carril 4, M. hyorhinis, ATCC17981, 585 pb, Carril 6, Producto no relacionado a este trabajo de 685 pb de p97 de M. hyopneumoniae, ATCC25617, Carril 7, M. hyosynoviae, cepa S-16, 1129 pb, también donada por la Universidad de Aarhus, Dinamarca, Carril 8, Marcador de peso molecular (1 Kb plus Invitrogen).
Cuadro 3: Relación de aislados identificados por PCR especie-específica para M. hyopneumoniae, M. hyorhinis, y M hyosynoviae
Muestra | Aislamiento positivo (%) | Mycoplasma spp | ||
M. hyopneumoniae | M. hyorhinis | M. hyosynoviae | ||
Pulmón | 2/9 (22.0) | 5/9 (55.5) | 4/9 (44.0) | 4/9 (44.0) |
Hisopo nasal | 2/27 (7.4) | 14/27 (51.8) | 9/27 (33.3) | 6/27 (22.2) |
Total | 4/36 (11.1) | 19/36 (52.7) | 13/36 (36.1) | 10/36 (27.7) |
La coexistencia de M. hyopneumoniae, M. hyorhinis y M hyosynoviae se detectó en diez muestras que representan el 27.77 % (10/36): en dos pulmones las tres especies, en otros dos pulmones y cinco hisopos nasales se identificó a M. hyorhinis y M. hyosynoviae, y solo un hisopo contenía M. hyopneumoniae y M. hyorhinis (Cuadro complementario 1). Adicionalmente, los géneros bacterianos asociados identificados por bacteriología general en hisopos nasales fueron E. coli, Enterobacter, Staphylococcus coagulasa-negativos, Klebsiella, Bordetella, Corynebacterium, Pasteurella, Shigella y Streptococcus. En muestras de pulmón, no se identificó crecimiento bacteriano.
Cuadro suplementario 1: Identificación de los aislamientos de Mycoplasma por PCR especie-específico
Número | Descripción | Tipo de muestra* | M. hyop | M. hyor | M. hyos | Géneros bacterianos |
1 | 111 | HN | - | + | - | SCN |
2 | 112 | HN | - | + | + | E. coli , Shigella |
3 | 113 | HN | - | - | - | Pasteurella |
4 | 114 | HN | - | - | - | Klebsiella, SCN |
5 | 115 | HN | - | + | + | Klebsiella, E. coli, SCN |
6 | 116 | HN | - | + | - | E. coli, SCN, Bordetella, Corynebacterium |
7 | 117 | HN | - | + | - | SCN, Corynebacterium |
8 | 118 | HN | - | + | - | SCN, Corynebacterium |
9 | 119 | HN | - | - | + | Enterobacter |
10 | 120 | HN | - | + | - | Corynebacterium |
11 | 121 | HN | - | - | - | Klebsiella, SCN |
12 | 122 | HN | - | + | + | Corynebacterium |
13 | 123 | HN | - | - | + | Klebsiella, SCN |
14 | 124 | HN | - | - | + | SCN |
15 | 125 | HN | - | + | - | Corynebacterium |
16 | 126 | HN | - | - | - | Corynebacterium |
17 | 127 | HN | - | - | - | Klebsiella, Corynebacterium |
18 | 130 | HN | + | - | - | SCN |
19 | 133 | HN | - | - | - | E. coli |
20 | 148 | HN | - | + | - | SCN |
21 | 159 | HN | + | + | - | Sin crecimiento bacteriano |
22 | 160 | HN | - | + | - | SCN |
23 | 161 | HN | - | - | + | Pasteurella |
24 | 162 | HN | - | + | + | E. coli, SCN |
25 | 165 | HN | - | + | - | Streptococcus, SCN |
26 | 168 | HN | - | + | - | E. coli |
27 | 170 | HN | - | + | + | SCN |
28 | 182 | P | - | + | + | Sin crecimiento bacteriano |
29 | 183 | P | - | + | + | Sin crecimiento bacteriano |
30 | 186 | P | - | - | - | Sin crecimiento bacteriano |
31 | 188 | P | - | - | - | Sin crecimiento bacteriano |
32 | 194 | P | + | + | + | Sin crecimiento bacteriano |
33 | 206 | P | + | + | + | Sin crecimiento bacteriano |
34 | 207 | P | - | + | - | Sin crecimiento bacteriano |
35 | 208 | P | - | - | - | Sin crecimiento bacteriano |
36 | 210 | P | - | - | - | Sin crecimiento bacteriano |
M. hyop= M. hyopneumoniae; M. hyor= M. hyorhinis; M. hyos= M. hyosynoviae; *HN= hisopo nasal, P= pulmón, SCN= Staphylococcus coagulasa-negativos.
Discusión
En México hay pocos estudios sobre la asociación de estas tres especies de Mycoplasma a NEP, principalmente por las dificultades del aislamiento y las inherentes a la muestra biológica. La concentración de microorganismos frecuentemente está por debajo del límite de detección como consecuencia del uso generalizado de antibióticos para el control de la micoplasmosis porcina. Por lo tanto, los procedimientos de aislamiento son necesarios para favorecer su crecimiento e identificación para fines de investigación y vigilancia. El procedimiento utilizado permitió identificar la asociación de las tres especies relacionadas con la producción porcina.
M. hyopneumoniae es la especie de Mycoplasma más frecuentemente aislada de cerdos con signos clínicos de neumonía y tiene una baja tasa de transmisión. Sin embargo, en asociación es capaz de aumentar la gravedad de las infecciones causadas por virus y bacterias(32).
M. hyorhinis ha pasado de ser un patógeno secundario(33,34), a ser considerado agente causal de NEP y CREP(35). En este estudio, esta especie de Mycoplasma es la más prevalente en los hisopos nasales 51.85 % (14/57). Esta observación puede ser explicada por el éxito de las medidas de control que se han implementado en las granjas de producción porcina.
Aquí se reporta que M. hyosynoviae está en estrecha interacción con las otras dos especies de Mycoplasma en pulmones con lesiones típicas de NEP. M. hyosynoviae se presentó en los hisopos nasales como un microorganismo asociado en alto porcentaje 33 % (9/27). Por lo que este Mycoplasma comensal podría tener potencial patógeno y se requerirán estudios adicionales para evaluar su papel en el desarrollo de la NEP.
El cultivo bacteriológico es el “estándar de oro” para el diagnóstico. Entre sus desventajas están el ser muy laborioso, rara vez usado como método de rutina, y no distingue entre las especies asociadas a la NEP. La PCR basada en la subunidad 16S rRNA permitió discernir entre M. hyopneumoniae y M. hyorhinis con precisión y rapidez. Por otro lado, se identificaron 10 casos, en los que las especies evaluadas en este trabajo, no estuvieron involucradas. Este resultado plantea la posibilidad de que otras especies de Mycoplasma pueden estar involucradas.
El método de colección (hisopo nasal, moco traqueobronquial, hisopado profundo postmortem, lavado broncoalveolar o tejido pulmonar) tiene un efecto significativo en la frecuencia de M. hyopneumoniae, ya que la frecuencia reportada varía de acuerdo al método del 3 al 40 %(36). Previamente en lechones, los hisopos nasales han sido el método de recolección de muestras antemortem a nivel de hato(28,37). Pieters et al(38) sugieren que los hisopos laríngeos son útiles en la etapa temprana de la infección en lechones. Otros autores afirman que el sitio de muestreo óptimo para la detección por métodos moleculares para M. hyopneumoniae es la colección de moco traqueobronquial (CMTB), ya que su sensibilidad es 3.5 veces más sensible en lechones de menos de 25 días(36).
El tracto respiratorio superior (cavidad nasal y faringe) desempeña un papel importante en el monitoreo y limpieza de microrganismos patógenos y también en la inducción de la respuesta inmune apropiada. M. hyopneumoniae coloniza principalmente los cilios del tracto respiratorio del cerdo(39). En animales adultos de granjas de producción, el CMTB se vuelve difícil y costoso de obtener. El manejo de animales es restringido en concordancia con las medidas vigentes para la prevención de la influenza porcina y de acuerdo a nuestra experiencia, recomendamos el uso de hisopos nasales como la técnica de muestreo adecuada.
La prevalencia de M. hyopneumoniae en cerdas infectadas naturalmente es de 36.4 %(40), en lechones puede variar de 3.6 a 16 %. La frecuencia aquí determinada de Mycoplasma fue mayor a la esperada 37.11 % (36/97): La presencia de Mycoplasma en pulmón fue de 22.50 % (9/40) y en hisopos nasales 47.36 % (27/57). La infección por una sola especie de Mycoplasma fue 44.44 % (16/36): en AMP 11.11 % (1/9) y en AHN 55.55 % (15/27). La asociación de más de una especie de Mycoplasma estuvo presente en el 27.77 % (10/36): la asociación de las tres especies representó el 2 % (2/10), y la asociación de M. hyorhinis y M. hyosynoviae el 5.15 % (5/10). La co-infección de M. hyopneumoniae y M. hyorhinis, y la de M. hyosynoviae y M. hyorhinis han sido previamente asociadas con problemas articulares. En este trabajo se identificaron ambas asociaciones en el tracto respiratorio de animales en las granjas porcinas con NEP.
La tasa de Mycoplasma asociados a NEP es variable, independientemente de que la tasa de infecciones mixtas permanezca constante(35). En este estudio, los géneros bacterianos asociados en infecciones mixtas fueron similares a los previamente reportados(41). La PCR puede ser complemento o alternativa al diagnóstico histopatológico, y representa una opción para la vigilancia epidemiológica e investigación. Además de que puede asistir en la eliminación de Mycoplasma spp de las granjas de producción porcina, ya que hasta el momento es la mejor estrategia de control a largo plazo, para muchos productores porcinos y proveedores de pie de cría(42).
Conclusiones e implicaciones
La frecuencia de Mycoplasma en granjas porcinas en los estados de Hidalgo, Guanajuato, Veracruz y México fue mayor a la esperada (40.27 %). Existen otras especies de Mycoplasma que pueden estar involucradas en el desarrollo de NEP y este trabajo agrega evidencia de M. hyorhinis como agente causal de NEP.
Agradecimientos
Este trabajo fue financiado dentro del Proyecto DGAPA UNAM PAPITT IN 222515.
Literatura citada: