https://doi.org/10.22319/rmcp.v11i4.5111
Artículo
Frecuencia de contaminación y de serotipos de Salmonella enterica y Escherichia coli en una operación integrada de matanza y deshuese de bovinos
Jorge Alfredo de la Garza-García a
María Salud Rubio Lozano a
María del Carmen Wacher-Rodarte b
Armando Navarro Ocaña c
Rigoberto Hernández-Castro d
Juan Xicohtencatl-Cortes e
Enrique Jesús Delgado Suárez a*
a Universidad Nacional Autónoma de México (UNAM). Facultad de Medicina Veterinaria y Zootecnia, Ciudad de México, México.
b UNAM. Facultad de Química. Ciudad de México. México.
c UNAM. Facultad de Medicina. Ciudad de México. México.
d Hospital General Dr. Manuel Gea González, Departamento de Ecología de Agentes Patógenos. Ciudad de México. México.
e Hospital Infantil de México Dr. Federico Gómez, Laboratorio de Bacteriología Intestinal. Ciudad de México. México.
* Autor de correspondencia: enriquedelgado.suarez@gmail.com
Resumen:
El objetivo fue determinar la frecuencia de contaminación y la diversidad de serotipos de Salmonella enterica (SE) y Escherichia coli (EC) en diferentes etapas de los procesos de matanza y deshuese de bovinos. Se tomaron muestras de heces, canales y cortes primarios (100 de cada tipo) en un rastro Tipo Inspección Federal ubicado en Mexicali, Baja California. EC no se analizó en heces, por ser esta bacteria parte de la microbiota intestinal. La identidad de las cepas se confirmó por métodos bioquímicos y por PCR: genes taxonómicos invA y gadA, para SE y EC, respectivamente. En EC se investigó también la presencia de genes asociados con los principales patotipos. SE tuvo una frecuencia de 34 % en heces, 3 % en canales y 2 % en cortes, siendo Montevideo el serotipo predominante (72.5 % del total de cepas). EC se detectó en canales (34 %) y en cortes (11 %), en concentraciones promedio de 0.012 y 0.33 log UFC cm-2, respectivamente. Aunque varios de los serotipos de EC identificados están asociados con cepas enterotoxigénicas o productoras de toxinas tipo Shiga, ninguno portaba factores de virulencia característicos de estos patotipos. En resumen, las canales y cortes de bovino no son una fuente relevante de cepas patógenas de EC. En contraste, los bovinos constituyen un importante reservorio de SE, lo cual representa un riesgo para la salud pública. Se requieren estudios genómicos sobre el perfil de virulencia y los genes asociados con infecciones subclínicas en cepas de SE provenientes de animales aparentemente sanos.
Palabras clave: Escherichia coli, Salmonella spp., Bovinos, Sacrificio, Serotipos, Patotipos.
Recibido: 16/10/2018
Aceptado: 23/09/2019
Introducción
Las infecciones intestinales causadas por Salmonella enterica y los distintos patotipos de E. coli, como los productores de las toxinas tipo Shiga (STEC), constituyen un problema de salud pública a escala global(1). Ambos patógenos suelen contaminar la carne de diferentes especies, entre ellas la de bovino(2,3), el segundo tipo de carne de mayor consumo en México(4). Por ello, la caracterización de las cepas circulantes de S. enterica y E. coli en la cadena productiva de bovinos de carne es de vital importancia para mejorar la gestión de los riesgos asociados con ambos patógenos.
La mayoría de los estudios realizados en este campo en México se concentran en un solo punto de la cadena productiva. Por ejemplo, varios autores han observado frecuencias moderadas (8 a 15 %) de Salmonella spp. en canales de bovino(5-7), aunque no en todos los casos se reportan los serotipos representados. En estudios más completos se reportan niveles de contaminación más elevados (25 a 100 %) en piel, heces, linfonodos, canales no refrigeradas y carne(8-10), detectándose predominancia de ciertos serotipos en algunas de las matrices analizadas. Sin embargo, la comparación entre estudios es difícil, debido a variaciones en cuanto al tipo de muestra analizada, segmento de la cadena productiva, metodología de análisis empleada, así como zona geográfica, sistema de producción animal y condiciones sanitarias del proceso en estudio.
En el caso de E. coli, la situación es similar. La mayoría de los estudios se enfoca en un segmento de la cadena productiva y en cepas STEC entero-hemorrágicas, tales como E. coli O157:H7(6,11,12). Aunque en estudios previos se ha reportado baja frecuencia (1a 3 %) de cepas patógenas de E. coli en canales y en heces de bovinos(11-13), no se ha explorado a fondo la distribución de éstas a lo largo de la cadena productiva. Esta información puede contribuir a identificar patrones de diseminación en diferentes procesos y en distintas zonas geográficas, así como medidas encaminadas a garantizar la inocuidad de los alimentos y a proteger la salud pública. Por tanto, el presente estudio tiene como objetivo determinar la frecuencia de contaminación y diversidad de serotipos de S. enterica y E. coli en un rastro Tipo Inspección Federal con integración horizontal de los procesos de matanza y deshuese de bovinos.
Material y métodos
Diseño del estudio y determinación del tamaño de muestra
Se realizó un muestreo en tres etapas del proceso de transformación del bovino desde la matanza hasta el deshuese: 1) contenido rectal obtenido después de la evisceración, 2) canales calientes y 3) cortes primarios. Cada etapa se consideró como un muestreo independiente, pues no fue posible determinar con anticipación el destino de los animales, los cuales se vendieron como canales enteras en algunos casos y en otros como cortes primarios. El tamaño de muestra para cada etapa a evaluar se calculó mediante la fórmula estadística para determinar el tamaño de muestra para una proporción de una población, cuando no se conoce el número de elementos en esa población(14):
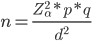 ; n= tamaño de muestra; Zα2 = valor de Z en una distribución normal Zα = 1.96 cuando α= 0.05; p= proporción de la población con la característica estudiada (si se desconoce se emplea 0.5, como en este caso); q= proporción aproximada de la población carente de la característica estudiada(1-p); d= error o precisión deseada, la cual se fijó en 10% (0.1).
; n= tamaño de muestra; Zα2 = valor de Z en una distribución normal Zα = 1.96 cuando α= 0.05; p= proporción de la población con la característica estudiada (si se desconoce se emplea 0.5, como en este caso); q= proporción aproximada de la población carente de la característica estudiada(1-p); d= error o precisión deseada, la cual se fijó en 10% (0.1).
Con dicha fórmula se obtuvo un tamaño de muestra por etapa de 96, el cual se redondeó a 100, para un total de 300 muestras en el estudio. El estudio se realizó en septiembre de 2013, en una empresa integrada de producción de carne de bovino situada en Mexicali, Baja California, con operaciones de engorda intensiva en corral, matanza y deshuese. Las canales muestreadas provenían de bovinos machos enteros, cruzas de Bos indicus, con una edad promedio de 24 a 30 meses, originarios de ocho estados de la República Mexicana y finalizados durante un promedio de 190 días en la engorda. La empresa fue seleccionada por sus características integrales, las cuales permiten tener un modelo de la cadena productiva en un solo sitio. El rastro se encuentra a 1 km de los corrales de engorda y tiene capacidad para procesar 300 bovinos por turno de 8 h.
Toma de muestras
Contenido rectal
Las muestras de heces se tomaron del recto, después de la evisceración, a los 20 min post mortem. De cada recto se tomaron aproximadamente 100 g de heces. Para ello, se retuvieron momentáneamente los paquetes de vísceras en la rampa de eviscerado, se abrió la ligadura del recto y se usaron guantes nuevos de nitrilo para recolectar las muestras de heces, las cuales se depositaron en bolsas estériles y se mantuvieron en hieleras con geles refrigerantes ( hasta su posterior procesamiento en el laboratorio. Cada muestra se tomó con un nuevo par de guantes. Como E. coli forma parte de la microbiota intestinal del bovino, se asumió que todas las muestras resultarían positivas y con altas concentraciones. Por esta razón, no se hizo el muestreo de rectos para E. coli, solamente para S. enterica.
hasta su posterior procesamiento en el laboratorio. Cada muestra se tomó con un nuevo par de guantes. Como E. coli forma parte de la microbiota intestinal del bovino, se asumió que todas las muestras resultarían positivas y con altas concentraciones. Por esta razón, no se hizo el muestreo de rectos para E. coli, solamente para S. enterica.
Canales sin refrigerar
Se empleó la metodología utilizada por el Departamento de Agricultura de los Estados Unidos en sus estudios microbiológicos de línea base para la especie bovina(15) con ligeras modificaciones. En lugar de canales refrigeradas, se muestrearon canales calientes y se empleó el agua peptonada de una misma canal para la detección de E. coli y S. enterica. En todos los casos, se muestrearon medias canales (lado derecho) empleando esponjas pre-humedecidas con 10 ml de agua peptonada tamponada y marcos estériles desechables de 10 x 10 cm2 (Meat/Turkey Carcass Sampling Kit, NASCO®, EEUU) en tres puntos de la canal (pierna, falda y pecho) con una superficie total de muestreo de 300 cm2 por canal.
Cortes
En la sala de cortes, una vez obtenidos los cortes primarios y antes del empacado, se seleccionaron al azar piernas, faldas y pechos para la toma de muestras. Para ello, se utilizó el mismo método descrito anteriormente para las canales, excepto porque se tomó un solo marco de 100 cm2 por corte.
Análisis microbiológicos
Una vez tomadas las muestras, las esponjas se sellaron en bolsas estériles de plástico y se mantuvieron en hieleras con geles refrigerantes ( para su traslado al laboratorio de la planta. De los 10 ml de agua peptonada se inocularon por triplicado 100 μl en placas con agar Salmonella-Shigella (SS) (MCD Lab®, PRONADISA-CONDA®, España), se incubaron a 37 ºC y se revisaron a las 24, 48 y 72 h. Las placas sin crecimiento de colonias con morfología típica de Salmonella spp. a las 72 h se consideraron negativas. Para las muestras de heces, se emplearon hisopos estériles para realizar la siembra directa en medio SS y se procedió de igual forma para la incubación y lectura de las placas. Cuando se obtuvieron colonias con morfología sugerente de Salmonella spp. (con producción de ácido sulfhídrico, colonias redondeadas, convexas de borde regular), éstas fueron resembradas en medio CHROMAgar Salmonella Plus (CHROMAgar®, Francia) para su purificación e identificación. Todas las colonias sugerentes a Salmonella spp. (productoras de ácido sulfhídrico o de coloración morada en CHROMAgar Salmonella Plus) fueron recuperadas. Los aislamientos puros y confirmados con el medio selectivo y diferencial fueron sembrados en agar tripticaseína de soya (TSA, MCD Lab®, PRONADISA-CONDA®, España) para su identificación por métodos bioquímicos y por PCR. Del volumen restante del agua peptonada, se tomó 1 ml para cada placa rehidratable 3M Petrifilm® E. coli/Coliformes (3M, USA), para estimar la concentración de Escherichia coli genérica. Las placas 3M Petrifilm fueron incubadas a 37 °C y analizadas a las 24 y 48 h, de acuerdo con las instrucciones del fabricante. Se empleó el medio CHROMAgar ECC (CHROMAgar®, Francia) para aislar las cepas identificadas en las placas 3M Petrifilm.
para su traslado al laboratorio de la planta. De los 10 ml de agua peptonada se inocularon por triplicado 100 μl en placas con agar Salmonella-Shigella (SS) (MCD Lab®, PRONADISA-CONDA®, España), se incubaron a 37 ºC y se revisaron a las 24, 48 y 72 h. Las placas sin crecimiento de colonias con morfología típica de Salmonella spp. a las 72 h se consideraron negativas. Para las muestras de heces, se emplearon hisopos estériles para realizar la siembra directa en medio SS y se procedió de igual forma para la incubación y lectura de las placas. Cuando se obtuvieron colonias con morfología sugerente de Salmonella spp. (con producción de ácido sulfhídrico, colonias redondeadas, convexas de borde regular), éstas fueron resembradas en medio CHROMAgar Salmonella Plus (CHROMAgar®, Francia) para su purificación e identificación. Todas las colonias sugerentes a Salmonella spp. (productoras de ácido sulfhídrico o de coloración morada en CHROMAgar Salmonella Plus) fueron recuperadas. Los aislamientos puros y confirmados con el medio selectivo y diferencial fueron sembrados en agar tripticaseína de soya (TSA, MCD Lab®, PRONADISA-CONDA®, España) para su identificación por métodos bioquímicos y por PCR. Del volumen restante del agua peptonada, se tomó 1 ml para cada placa rehidratable 3M Petrifilm® E. coli/Coliformes (3M, USA), para estimar la concentración de Escherichia coli genérica. Las placas 3M Petrifilm fueron incubadas a 37 °C y analizadas a las 24 y 48 h, de acuerdo con las instrucciones del fabricante. Se empleó el medio CHROMAgar ECC (CHROMAgar®, Francia) para aislar las cepas identificadas en las placas 3M Petrifilm.
Identificación bioquímica
Las cepas de Salmonella se identificaron con substratos preparados en el laboratorio, de acuerdo con los resultados de las siguientes pruebas(16): triple azúcar hierro (TSI, por sus siglas inglesas); ácido sulfhídrico, indol y motilidad (SIM, por sus siglas en inglés); citrato de Simmons; urea; rojo de metilo-Vosges-Proskauer; malonato-fenilalanina; gluconato; enzimas descarboxilasas de arginina, ornitina, lisina y testigo. Se utilizó Salmonella enterica subsp. enterica ser. Typhimurium ST19, como control positivo, la cual se obtuvo del cepario del Hospital General Dr. Manuel Gea González, de la Ciudad de México, aislada y caracterizada por VITEK 2 (bioMerieiux, Francia)(17). Para E. coli se usaron las mismas pruebas, excepto la de descarboxilación de aminoácidos(16) y se empleó como control positivo una cepa de E. coli K12.
Identificación molecular
La identificación molecular se realizó mediante PCR de punto final, empleando iniciadores de secuencias específicas de genes típicos de cada especie (Cuadro 1). El ADN genómico se extrajo con el kit “DNeasy Blood & Tissue Kit” (Qiagen, Inc., USA), según instrucciones del fabricante, a partir de las cepas puras, previamente refrescadas en caldo tripticasa soya (MCD Lab®, PRONADISA-CONDA®, España) por 18 a 24 h. Para S. enterica se utilizó el gen invA(18) y para E. coli, el gen gadA(19), el cual codifica para la subunidad alfa de la glutamato descarboxilasa, perteneciente al sistema de resistencia al estrés ácido en E. coli. Además, para identificar los patotipos presentes, se incluyeron seis genes asociados con cepas enteropatogénicas (EPEC), enterotoxigénicas (ETEC) y productoras de toxina tipo Shiga (STEC). Entre estos, se seleccionó el gen eaeA, el cual codifica para una intimina, proteína importante para la adhesión mediante el receptor translocado de intimina(20) y se encuentra presente en el genoma de los patotipos EPEC y STEC. Por su parte, los genes codificantes de las toxinas tipo Shiga 1 (stx1) y 2 (stx2) suelen presentarse en las cepas STEC, las cuales manifiestan el mismo fenotipo cuando portan uno de estos genes o ambos(20). También se investigó la presencia de genes codificantes para las toxinas termoestable (estA) y termolábil (eltA), asociados con cepas ETEC(21); así como el gen bfp (del inglés bundle forming pilus), codificante de los pili formadores de penachos, involucrados en la adhesión al epitelio intestinal, el cual se encuentra en el genoma de cepas EPEC(2). Las reacciones de PCR se realizaron en un volumen total de 25 μl y se utilizaron los reactivos del Top Taq Master Mix Kit (QIAGEN®, USA) con las siguientes concentraciones finales: 1.25 Unidades de Taq Polimerasa, 1.5 mM MgCl2, 1x Buffer para PCR, 200 μM de cada dNTP. Las condiciones empleadas para cada reacción fueron las mismas descritas en publicaciones anteriores (Cuadro 1).
Cuadro 1: Genes e iniciadores empleados para la caracterización molecular de Salmonella spp. y Escherichia coli
Patógeno |
Gen
| Fragmento amplificado (pb) | Secuencia de Iniciadores 5´🡪3´ | Ref. |
Salmonella spp. | invA | 284 | 139 GTGAAATTATCGCCACGTTCGGGCAA 141 TCATCGCACCGTCAAAGGAACC | (19) |
E. coli | gadA | 670 | gadA1: ACCTGCGTTGCGTAAATA gadA2: GGGCGGGAGAAGTTGATG | (20) |
eaeA | 890 | EAE1: GTGGCGAATACTGGCGAGACT EAE2: CCCCATTCTTTTTCACCGTCG | (21) |
stx1 | 582 | STX1F: ACACTGGATGATCTCAGTGG STX1R: CTGAATCCCCCTCCATTATG | (21) |
stx2 | 255 | STX2F: GGCACTGTCTGAAACTGCTCC STX2R: TCGCCAGTTATCTGACATTCTG | (21) |
estA | 190 | STa-F CTAATGTTGGCAATTTTTATTTCTGTA STa-R AGGATTACAACAAAGTTCACAGCAGTAA | (22) |
eltA | 132 | LT-1 AGCAGGTTTCCCACCGGATCACCA LT-2 GTGCTCAGATTCTGGGTCTC | (22) |
bfp | 324 | EP1, CAATGGTGCTTGCGCTTGCT EP2, GCCGCTTTATCCAACCTGGT | (2) |
Los productos de amplificación por PCR fueron procesados por electroforesis en geles de agarosa (SeaKem® LE Agarose, Lonza, ME, USA) al 1% para los productos de alto peso molecular (gadA, eaeA, stx1) y al 2% para los más pequeños (eltA y estA). Los geles se corrieron en buffer tris/borato/EDTA (TBE 1x), a 80 V por 50 min. Se usó SYBR Safe DNA Gel Stain (Invitrogen, USA) para revelar los fragmentos de ADN. La visualización y digitalización de imágenes se realizó en un fotodocumentador Gel Logic 2200 (Kodak, USA) con el software Care Stream® (Carestream Health, Inc., USA). Se utilizaron como controles positivos las mismas cepas de ambos patógenos referidas en la identificación bioquímica. Además, para la identificación de los patotipos de E. coli se incluyeron como controles cepas de cada uno de los patotipos investigados (EPEC, ETEC y STEC), también provenientes del cepario del laboratorio del Hospital General Dr. Manuel Gea González, previamente identificados por medio del sistema VITEK 2.
Serotipificación
Salmonella spp.
Tipificación serológica del antígeno somático (O). La identificación serológica de las cepas
de Salmonella se realizó de acuerdo al esquema de Kauffmann–White(22,23). La obtención del antígeno somático (O) se llevó a cabo hirviendo los cultivos bacterianos ( durante 1 h. El antígeno O se determinó utilizando sueros polivalentes anti-O A, B, D, D, E, F y G de (DIFCO, BD) y sueros anti-O monovalentes (específicos) de los serogrupos A, C, D, E y F (DIFCO, BD).
durante 1 h. El antígeno O se determinó utilizando sueros polivalentes anti-O A, B, D, D, E, F y G de (DIFCO, BD) y sueros anti-O monovalentes (específicos) de los serogrupos A, C, D, E y F (DIFCO, BD).
Tipificación serológica del antígeno flagelar (H). Este antígeno se obtuvo inoculando las
cepas en medio semisólido en tubos de Cragie´s y resembrando en caldo nutritivo. El antígeno H de fase I y II de las cepas se determinó utilizando el sistema de antisueros-H Spicer-Edwars (DIFCO) y sueros monovalentes (específicos) de los serogrupos A, B, C, D, E y F.
Aunque la determinación del serotipo no se realizó en un laboratorio de referencia, el genoma completo de las cepas obtenidas fue secuenciado, como parte de otra investigación(24). Ello permitió confirmar, mediante análisis in silico de las secuencias crudas, los resultados preliminares de serotipificación y determinar el serotipo de las cepas no tipificables por métodos bioquímicos.
E. coli
Las cepas de E. coli fueron serotipificadas utilizando microaglutinación en placa de 96 pozos y 187 sueros anti-antígeno somático (O) y 53 sueros anti-antígeno flagelar (H) obtenidos en conejo (SERUNAM), empleando el método descrito por Ørskov y Ørskov(25), con modificaciones menores.
Clasificación por filogrupos. Como ciertos filogrupos de E. coli están asociados con animales o con humanos, así como con diferentes patotipos de la bacteria, se decidió realizar la clasificación en grupos filogenéticos mediante PCR, según el esquema de Clermont(26). Esta técnica permite dividir a los aislamientos de E. coli en siete grupos filogenéticos característicos de la especie (A, B1, B2, C, D, E y F) y un grupo adicional, correspondiente al Clado Críptico I. La prueba se realizó mediante una reacción de PCR cuádruple inicial, para detectar los genes arpA, chuA, yjaA y TSPE4.C2. Además, en caso de obtener resultados sugerentes de filogrupos E y C, se realiza un PCR dúplex adicional dirigida hacia una variante alélica del gen arpA (específica del grupo E) o bien, a una variante alélica del gen trpA (específica para el grupo C), más un control interno dirigido para el gen trpBA. Las reacciones se llevaron a cabo directamente, a partir de colonias frescas crecidas 24 h en agar TSA. Las reacciones de PCR se realizaron a un volumen de 25 μl, bajo las mismas condiciones descritas previamente(26). Los productos de amplificación por PCR fueron procesados por electroforesis en gel de agarosa (SeaKem® LE Agarose, Lonza, EEUU) al 2%, corriendo a voltaje constante de 80 V por 50 min. La visualización y digitalización de imágenes se realizó de la misma forma antes descrita para S. enterica. Se incluyeron como controles positivos E. coli K12 y cepas representativas de cada uno de los filogrupos de E. coli, provenientes del cepario del Hospital General Dr. Manuel Gea González, clasificadas previamente según el esquema de Clermont(26).
Análisis de la información
Se calculó la frecuencia de positividad para ambos patógenos en las muestras evaluadas, así como la concentración, sólo en el caso de E. coli. Se utilizó la prueba de Ji cuadrada y la razón de momios para detectar asociación entre la positividad a los patógenos y el tipo de muestra
Resultados
De las muestras analizadas (300 para Salmonella spp. y 200 para E. coli) se obtuvieron 84 aislamientos, 39 de ellos fueron identificados como Salmonella spp. y otros 45 como E. coli, con una frecuencia global de 13.0 y 22.5 %, respectivamente. Sólo una de las muestras, proveniente de una canal, fue positiva para ambas bacterias.
Salmonella spp.
La frecuencia de contaminación con Salmonella spp. fue de 34 % en heces y de 3 y 2 % en canales y en cortes, respectivamente (Figura 1). Todos los aislamientos obtenidos fueron identificados como Salmonella spp. mediante pruebas bioquímicas y PCR. Inicialmente se habían aislado otras dos cepas, con resultados positivos basados en pruebas bioquímicas y en PCR, pero resultaron no tipificables. No obstante, al confirmar el serotipo, por análisis in silico con las lecturas crudas de la secuenciación del genoma, estas dos cepas fueron descartadas, pues se identificaron como Pseudomonas putida, una especie también portadora del gen invA(27).
Figura 1: Presencia de Salmonella spp. en heces, canales y cortes de bovino (n=100 por tipo de muestra)

La prueba de Ji cuadrada evidenció la existencia de una fuerte asociación ( 2=58.5, P<0.0001) entre el tipo de muestra y la frecuencia de contaminación con Salmonella spp (Cuadro 2). Esto fue confirmado por la razón de momios, según la cual la probabilidad de encontrar muestras positivas a Salmonella spp. en heces fue 20.1 veces mayor en relación con las demás matrices.
2=58.5, P<0.0001) entre el tipo de muestra y la frecuencia de contaminación con Salmonella spp (Cuadro 2). Esto fue confirmado por la razón de momios, según la cual la probabilidad de encontrar muestras positivas a Salmonella spp. en heces fue 20.1 veces mayor en relación con las demás matrices.
Cuadro 2: Asociación entre la frecuencia de contaminación con Salmonella spp. y Escherichia coli y el tipo de muestra
Tipo de muestra | n | % positividad | Razón de momios | I. C. al 95%1 |  2 2
| P2 |
Salmonella spp. |
|
|
|
|
|
|
Heces | 100 | 34 | 20.1 | 7.5-53.5 | 58.5 | <0.0001 |
Canales/cortes | 200 | 5 |
E. coli |
|
|
|
|
|
|
Canales | 100 | 34 | 4.2 | 2.0-8.8 | 15.2 | <0.0001 |
Cortes | 100 | 11 |
1Intervalo de confianza al 95% para la razón de momios.
2Nivel de significancia (probabilidad).
Con respecto a los serotipos presentes (Figura 2), se logró tipificar 35 de los 39 aislamientos por métodos serológicos. Las otras cuatro cepas sólo pudieron ser caracterizadas parcialmente, pues presentaron un antígeno O rugoso, por lo que sólo fue posible obtener una fórmula antigénica parcial, basada en el antígeno flagelar. No obstante, el análisis in silico, con las lecturas crudas de secuenciación genómica completa reportado en otra investigación(24), permitió determinar el serotipo del 100 % de los aislamientos. En total, se identificaron cinco serovariedades: Bergen (n= 1), Reading (n= 2), Muenster (n= 3), Newport (n= 4) y Montevideo (n= 29). Todos los aislamientos correspondientes al serotipo Montevideo fueron monofásicos para el antígeno H, aunque las fórmulas antigénicas permitieron identificar dos subgrupos dentro de este serotipo, 22 de ellos provenientes de heces, canales y cortes, con la fórmula 6,7:g,m,s:-. Las siete cepas restantes, todas provenientes de heces, presentaron la fórmula 6,7:g,m,p,s:-.
Figura 2: Distribución de serotipos de Salmonella enterica subsp. enterica de acuerdo con el origen de la muestra (n=100 por tipo de muestra)

La distribución de serotipos por clase de muestra evidenció la presencia de Salmonella enterica subsp. enterica ser. Montevideo en todas las matrices analizadas. Por el contrario, las cepas de Salmonella enterica subsp. enterica ser. Newport y Reading, detectadas en menor frecuencia que Salmonella Montevideo, sólo se detectaron en las heces (Figura 2).
E. coli
E. coli se encontró en 34 % de las canales y en 11 % de los cortes. Se evidenció una fuerte asociación ( 2=15.2, P<0.0001) entre el tipo de muestra y la frecuencia de contaminación con E. coli (Cuadro 2). Esto fue confirmado por la razón de momios, la cual mostró que la probabilidad de encontrar muestras positivas en canales fue mayor que en cortes (razón de probabilidades 4.2, intervalo de confianza al 95% 2.0-8.8, P<0.0001.
2=15.2, P<0.0001) entre el tipo de muestra y la frecuencia de contaminación con E. coli (Cuadro 2). Esto fue confirmado por la razón de momios, la cual mostró que la probabilidad de encontrar muestras positivas en canales fue mayor que en cortes (razón de probabilidades 4.2, intervalo de confianza al 95% 2.0-8.8, P<0.0001.
En los cortes, las muestras positivas se distribuyeron de forma relativamente homogénea, obteniéndose cinco cepas del pecho, tres de la falda y tres de la pierna. La concentración de esta bacteria fue baja, tanto en canales como en cortes, con valores entre 1 y 8 UFC cm-2. De los 45 aislamientos obtenidos, identificados mediante placas 3M petrifilm y CHROMAgar ECC, 41 mostraron un fenotipo característico de la especie. Las cuatro cepas restantes, todas provenientes de canales, presentaron resultados atípicos; tres de ellas indol negativas y con lenta fermentación de lactosa y una más resultó positiva a citrato, malonato y celobiosa. No obstante, todas las cepas fueron confirmadas molecularmente por PCR, empleando el gen gadA como marcador taxonómico.
Se identificaron total o parcialmente 31 serotipos de E. coli (Cuadro 3). Los serogrupos más frecuentes fueron O8 (29%) y O71 (19.4%) y el serotipo más comúnmente encontrado fue el O1:H6 (9.7 %).
Cuadro 3: Frecuencia de serotipos de Escherichia coli parcial o totalmente identificados por tipo de muestra
Tipo de muestra | n | Serotipo |
Canal | 1 3 1 3 1 1 1 1 1 1 5 1 1 1 | O28ab:- -:H30 -:H32 O1:H6 O113:- O154:H21 O156:- O166:H21 O32:- O6:- O8:- O8:H19 O8:H2 O8:H21 |
Pierna | 1 | -:H32 |
Pecho | 1 1 2 1 | O124:- O71:- O71:H12 O8:H8 |
Falda | 1 2 | O7:H39 O71:H12 |
Los grupos filogenéticos predominantes fueron A (60 %) y B1 (26.7 %), el grupo B2 estuvo ausente y los grupos C y D se presentaron en frecuencias bajas (2.2 y 6.7 %). Hubo dos cepas con resultados no concluyentes, por lo cual no se asignaron a un filogrupo. Resultó interesante observar cómo algunos serogrupos estuvieron fuertemente asociados con ciertos grupos filogenéticos. En el serogrupo O8, 8 de 9 cepas pertenecen al filogrupo B1. De igual forma, 4 de 5 cepas del serogrupo O71 pertenecen al filogrupo A y todas las cepas del serogrupo O1 pertenecen al filogrupo D (Figura 3).
Figura 3: Grupos filogenéticos de E. coli, según el esquema de Clermont(26), representados en cada uno de los serogrupos identificados (n=43)

Discusión
En varios países desarrollados, con sistemas de explotación intensiva de ganado bovino similares a los que existen en naciones en desarrollo, como México, los niveles de contaminación con Salmonella spp. suelen ser bajos, tanto en canales y carne como en muestras de heces(28-30). Sin embargo, en este trabajo se observó una frecuencia moderadamente alta de contaminación en heces, la cual coincide con la reportada en otros rastros TIF del país(9). Esto indica que, en México, las granjas de bovinos de engorda pueden constituir un importante reservorio de este patógeno. Sin duda, esto representa un desafío considerable para las intervenciones aplicadas durante la matanza, donde se reduce drásticamente la contaminación de las canales en relación con las excretas, pero no alcanzan a controlar por completo el patógeno. Asimismo, Salmonella también fue detectada en cortes primarios, evidencia del potencial de este organismo para diseminarse a lo largo de la cadena productiva. Esto se demuestra al detectar cepas de un mismo serotipo en heces, canales y cortes. Además, los resultados son similares a los de estudios previos (2 a 30 % de positividad a Salmonella) en muestras de carne en supermercados(31,32), los cuales sólo expenden carne proveniente de rastros TIF. Esta situación implica un panorama epidemiológico más preocupante en las cadenas de comercialización asociadas con rastros municipales, los cuales carecen de la infraestructura y de las condiciones sanitarias existentes en rastros TIF(33). De hecho, la frecuencia de positividad a Salmonella en puntos de venta de carne de rastros municipales por lo general supera el 50%(10,34).
Lo analizado hasta el momento evidencia la necesidad de reforzar las medidas de control de Salmonella spp. en animales vivos, ya que las intervenciones aplicadas en granja son limitadas. En este sentido, la evaluación de la prevalencia de Salmonella spp. en los becerros destinados a las granjas de engorda, el monitoreo de animales infectados, su manejo separado y así la detección de posibles reservorios del patógeno, son sólo algunas de las medidas favorables para disminuir el porcentaje de animales recibidos en el rastro portadores de esta bacteria.
Con respecto a los serotipos detectados, todos han sido relacionados previamente con infecciones humanas en México(35), por lo cual el riesgo de estas cepas para la salud pública no debe minimizarse. El claro predominio de Salmonella Montevideo en los procesos estudiados es notorio, además de sorpresivo, tomando en cuenta que la empresa participante engorda animales provienentes de ocho Estados del país. También resulta interesante la ausencia de serotipos anteriormente comunes en muestras asociadas con ganado bovino en México, tales como Typhimurium, Anatum y Agona, entre otros(36). Aunque se ha reportado una distribución variable de serotipos de Salmonella spp. en función del tiempo, así como entre zonas geográficas y estudios, la predominancia de Salmonella Montevideo en la muestra estudiada es consistente con la creciente prevalencia de este serotipo en América del Norte(37,38). Asimismo, en estudios recientes realizados en México se reportan serotipos como Montevideo y Reading, pero no Typhimurium, en cepas obtenidas de heces, canales y linfonodos de bovinos(8, 9). En cualquier caso, resulta difícil determinar los factores asociados con el predominio de ciertas cepas en sistemas de producción animal sin recurrir a estudios moleculares en los cuales se aborde la diversidad genética de las poblaciones, así como la presencia de genes asociados con virulencia, persistencia ambiental e infecciones subclínicas. No obstante, los resultados indican que bovinos aparentemente sanos pueden portar Salmonella spp. en frecuencias moderadamente altas, así como la capacidad del patógeno para diseminarse más allá del proceso de matanza y faenado, con los riesgos consecuentes en términos de inocuidad de alimentos.
En relación con E. coli, aunque se encontró con una frecuencia similar a la de Salmonella en canales y en un porcentaje más elevado en cortes, esta bacteria se presentó en bajas concentraciones (<8 UFC cm-2). A pesar de que E. coli es parte de la microbiota intestinal, las intervenciones aplicadas en el rastro lograron reducir tres veces la frecuencia de esta bacteria en los cortes, en los cuales se observó una menor probabilidad de encontrar muestras positivas en relación con las canales. Además, al parecer existe en el país una escasa circulación de cepas patógenas en bovinos, a diferencia de otros países, como Estados Unidos de América, en los que éstas se consideran un problema de salud pública(39). Así lo demostró la ausencia de factores de virulencia asociados con los patotipos STEC, EPEC y ETEC en las muestras estudiadas. Estos hallazgos coinciden con investigaciones previas(13) en las que se detectaron serovariedades (O157 y no O157) asociadas con cepas STEC (n= 146), pero sólo dos de éstas portaban los factores de virulencia característicos. Esta tendencia se mantiene en estudios posteriores, en los cuales se reportan tasas bajas (<1 %) de contaminación con cepas patógenas de E. coli en canales y carne molida de bovinos en México(9,40,41). Tal comportamiento podría obedecer a múltiples factores. Entre estos, el fotoperíodo, más largo durante el verano en los países situados más al norte, se ha considerado como responsable del marcado efecto estacional en la prevalencia de E. coli patogénica en ganado bovino en estas naciones. Otros autores han sugerido que la circulación de otras enterobacterias, con reactividad cruzada de antígenos somáticos (O), en las poblaciones bovinas de México, podría ser un factor de selección negativa de patotipos de E. coli(42). Ello coincide con el alto porcentaje de muestras de suero bovino, proveniente de animales aparentemente sanos, con respuesta bactericida contra E. coli O157 (71 %) en hatos del centro de México(43).
En cuanto a los filogrupos de E. coli identificados, el predominio de los grupos A y B1 es consistente con lo comúnmente observado en cepas de origen animal(44,45). En línea con la ausencia de genes de virulencia asociados con patotipos, solamente una cepa fue clasificada en el grupo C, al cual pertenecen otras cepas STEC de origen animal(44,46). No obstante, prácticamente todos los serotipos identificados han sido asociados con los patotipos STEC o ETEC, ambos de importancia en enfermedades transmitidas por alimentos(47-49). Aunque no se ha estudiado el riesgo a la salud en cepas de serotipos carentes de genes de virulencia, éste no debe descartarse del todo, puesto que cepas no patógenas podrían adquirir dichos factores mediante la incorporación de plásmidos y/o fagos(50,51), un área donde se requiere de más investigación.
Conclusiones e implicaciones
En este estudio se comprobó que más de la tercera parte de los bovinos aprobados para la matanza portan diferentes serotipos de Salmonella enterica en las heces, a pesar de ser animales aparentemente sanos. Además, los resultados evidencian la capacidad del patógeno para diseminarse hacia los siguientes segmentos de la cadena productiva, con los riesgos consecuentes para la salud pública. De ahí la importancia de realizar estudios posteriores sobre los factores genéticos de S. enterica asociados con el establecimiento de infecciones subclínicas en el bovino, así como con su persistencia en las poblaciones ganaderas. Por otra parte, los resultados para E. coli evidencian, al igual que en otras zonas del país, una escasa circulación de cepas patógenas de E. coli en canales y cortes de bovino. No obstante, las muestras analizadas provienen de un solo rastro y el alcance del presente trabajo, para E. coli, no considera muestras de piel o de heces, en las cuales existe mayor probabilidad de encontrar cepas patógenas.
Agradecimientos y conflictos de interés
Este estudio fue realizado con recursos del fondo sectorial SAGARPA-CONACYT, proyecto 109127. Se agradece el apoyo técnico de los profesionales del Hospital General “Dr. Manuel Gea González”, de la Facultad de Medicina, Universidad Nacional Autónoma de México y de la Universidad Autónoma de Baja California, por su asistencia en la conducción de los experimentos y en los análisis de laboratorio. Los autores declaran que no tienen conflictos de interés con respecto a la presente publicación.
Literatura citada:
- WHO. WHO estimates of the global burden of foodborne diseases. Foodborne disease burden epidemiology reference group 2007-2015. World Health Organization. http://www.who.int/foodsafety/areas_work/foodborne-diseases/ferg/en/. Accessed Oct 25, 2018.
- Croxen MA, Law RJ, Scholz R, Keeney KM, Wlodarska M, Finlay BB. Recent advances in understanding enteric pathogenic Escherichia coli. Clin Microbiol Rev 2013;26(4):822-880.
- Koohmaraie M, Scanga JA, De La Zerda MJ, Koohmaraie B, Tapay L, Beskhlebnaya V, et al. Tracking the sources of Salmonella in ground beef produced from nonfed cattle. J Food Protect 2012;75(8):1464-1468.
- FAOSTAT. Food Balance Sheets. Food and Agricultural Organization of the United Nations, Statistics Division. http://faostat3.fao.org/download/FB/FBS/E. Consultado 25 Feb, 2019.
- Varela-Guerrero JA, Talavera-Rojas M, Gutiérrez-Castillo AC, Reyes-Rodríguez NE, Vázquez-Guadarrama J. Phenotypic-genotypic resistance in Salmonella spp. isolated from cattle carcasses from the north central zone of the State of Mexico. Trop Anim Health Prod 2013;45(4):995-1000.
- Hernández-San Juan S, Zúñiga-Estrada A, Sánchez-Ortega I, Castro-Rosas J, Román-Gutiérrez AD, Santos-López EM. Condiciones microbiológicas en el proceso de sacrificio en un rastro municipal del estado de Hidalgo, México. Vet Mex 2007;38(2):187-194.
- Pérez-Montaño JA, González-Aguilar D, Barba J, Pacheco-Gallardo C, Campos-Bravo CA, García S, Heredia NL, Cabrera-Díaz E. Frequency and antimicrobial resistance of Salmonella serovars on beef carcasses at small abattoirs in Jalisco State, Mexico. J Food Prot 2012;75(5):867-873.
- Gragg SE, Loneragan GH, Nightingale KK, Brichta-Harhay DM, Ruiz H, Elder JR, et al. Substantial within-animal diversity of Salmonella isolates from lymph nodes, feces, and hides of cattle at slaughter. Appl Environ Microbiol 2013;79(15):4744-50.
- Narvaez-Bravo C, Miller MF, Jackson T, Jackson S, Rodas-Gonzalez A, Pond K, Echeverry A, Brashears MM. Salmonella and Escherichia coli O157:H7 Prevalence in cattle and on carcasses in a vertically integrated feedlot and harvest plant in Mexico. J Food Prot 2013;76(5):786-795.
- Zaidi MB, McDermott P, Fedorka-Cray P, Leon V, Canche C, Hubert S, et al. Nontyphoidal Salmonella from human clinical cases, asymptomatic children and raw retail meats in Yucatan, Mexico. Clin Infect Dis 2006;42:21-28.
- Callaway TR, Anderson RC, Téllez G, Rosario C, Nava GM, Eslava C, et al. Prevalence of Escherichia coli O157 in cattle and swine in central Mexico. J Food Prot 2004;67(10):2274-2276.
- Amézquita-López BA, Quinones B, Cooley MB, Leon-Felix J, Castro-del Campo N, Mandrell RE, et al. Genotypic analyses of shiga toxin-producing Escherichia coli O157 and non-O157 recovered from feces of domestic animals on rural farms in Mexico. PLoS One 2012;7(12):e51565.
- Varela-Hernández JJ, Cabrera-Díaz E, Cardona-López MA, Ibarra-Velázquez LM, Rangel-Villalobos H, Castillo A, et al. Isolation and characterization of Shiga toxin-producing Escherichia coli O157:H7 and non-O157 from beef carcasses at a slaughter plant in Mexico. Int J Food Microbiol 2007;113(2):237-41.
- Jekel JF, Katz DL, Elmore JG. Epidemiology, biostatistics, and preventive medicine. Third ed. Philadelphia, PA, United States of America: SAUNDERS ELSEVIER; 2007.
- USDA/FSIS. Nationwide Sponge Microbiological Baseline Data Collection Program: Cattle. https://www.fsis.usda.gov/wps/wcm/connect/197ddb9c-1fa5-411e-ab3f-3d253c879370/Baseline_Data_Cattle.pdf?MOD=AJPERES. Accessed Mar 5, 2019.
- Barrow GI, Feltham RKA. Cowan and Steel's manual for the identification of medical bacteria. 3rd Ed. ed. Cambridge, United Kingdom: Cambridge University Press; 1993.
- Bruins MJ, Bloembergen P, Ruijs GJHM, Wolfhagen MJHM. Identification and susceptibility testing of Enterobacteriaceae and Pseudomona aeruginosa by direct inoculation from positive BACTEC blood culture bottles into Vitek 2. J Clin Microbiol 2004;42(1):7-11.
- Rahn K, De Grandis SA, Clarke RC, Curtiss R, Gyles CL. Amplification of an invA gene sequence of Salmonella typhimurium by polymerase chain reaction as a specific method of detection of Salmonella. Mol Cell Probes 1992;6:271-279.
- McDaniels AE, Rice EW, Reyes AL, Johnson CH, Haugland RA, Stelma GNJ. Confirmational identification of Escherichia coli, a comparison of genotypic and phenotypic assays for glutamate decarboxylase and β-d-glucuronidase. Appl Environ Microbiol 1996;62(9):3350-3354.
- DebRoy C, Roberts E. Screening petting zoo animals for the presence of potentially pathogenic Escherichia coli. J Vet Diagn Invest 2006;18(6):597-600.
- Bisi-Johnson MA, Obi CL, Vasaikar SD, Baba KA, Hattori T. Molecular basis of virulence in clinical isolates of Escherichia coli and Salmonella species from a tertiary hospital in the Eastern Cape, South Africa. Gut Pathog 2011;3(9).
- Grimont PAD, Weill F. Antigenic formulae of the Salmonella serovars. WHO Collaborating Centre for Reference and Research on Salmonella, Institut Pasteur. https://www.pasteur.fr/sites/default/files/veng_0.pdf. Accessed Feb 25, 2019.
- Popoff MY, Le Minor LE. Salmonella. In: Bergey's Manual of Systematics of Archaea and Bacteria. Whitman FR, et al editors. Hoboken, New Jersey: John Wiley & Sons, Inc.; 2015:764-799
- Delgado-Suárez EJ, Selem-Mojica N, Ortiz-López R, Gebreyes WA, Allard MW, Barona-Gómez F, Rubio-Lozano MS. Whole genome sequencing reveals widespread distribution of typhoidal toxin genes and VirB/D4 plasmids in bovine-associated nontyphoidal Salmonella. Sci Rep 2018;8(1):9864.
- Ørskov F, Ørskov I. Serotyping of Escherichia coli. Method Microbiol 1984;14:43-112.
- Clermont O, Christenson JK, Denamur E, Gordon DM. The Clermont Escherichia coli phylo-typing method revisited: improvement of specificity and detection of new phylogroups. Environ Microbiol Rep 2013;5(1):58-65.
- Duan J, Jiang W, Cheng Z, Heikkila JJ, Glick BR. The complete genome sequence of the plant growth-promoting bacterium Pseudomonas sp. UW4. PLoS One 2013;8(3):e58640.
- Eguale T, Engidawork E, Gebreyes WA, Asrat D, Alemayehu H, Medhin G, Johnson RP, Gunn JS. Fecal prevalence, serovar distribution and antimicrobial resistance of Salmonellae in dairy cattle in central Ethiopia. BMC Microbiol 2016;16:20.
- Sandt CH, Fedorka-Cray PJ, Tewari D, Ostroff S, Joyce K, M'Ikanatha NM. A comparison of non-typhoidal Salmonella from humans and food animals using pulsed-field gel electrophoresis and antimicrobial susceptibility patterns. PLoS One 2013;8(10):e77836.
- Khen BK, Lynch OA, Carroll J, McDowell DA, Duffy G. Prevalence and characteristics of Salmonella in the beef chain in the Republic of Ireland. Zoonoses Public Health 2014;61(8):534-6.
- Ballesteros-Nova N, Rubio-Lozano MS, Delgado-Suárez EJ, Méndez-Medina RD, Braña-Varela D, Rodas Suárez O. Perfil de resistencia a antibióticos de serotipos Salmonella spp. aislados de carne de res molida en la Ciudad de México. Salud Pública México 2016;58(3):1-7.
- Rubio-Lozano MS, Martínez-Bruno JF, Hernández-Castro R, Bonilla-Contreras C, Méndez MRD, Núñez-Espinosa JF, et al. Detección de Listeria monocytogenes, Salmonella y Yersinia enterocolitica en carne de res en puntos de venta en México. Rev Mex Cienc Pecu 2013;4(1):107-115.
- Signorini M. Evaluación de riesgos de los rastros y mataderos municipales. Nacameh 2007;1(2):118-141.
- Cabrera-Díaz E, Barbosa-Cárdenas CM, Pérez-Montano JA, González-Aguilar D, Pacheco-Gallardo C, Barba J. Occurrence, serovar diversity, and antimicrobial resistance of Salmonella in ground beef at retail stores in Jalisco state, Mexico. J Food Prot 2013;76(12):2004-2010.
- Gutiérrez-Cogco L, Montiel-Vázquez E, Aguilera-Pérez P, González-Andrade MC. Serotipos de Salmonella identificados en los servicios de salud de México. Salud Publica Mex 2000;42(6):490-495.
- Zaidi MB, Calva JJ, Estrada-García MT, León V, Vázquez G, Figueroa G, et al. Integrated food chain surveillance system for Salmonella spp. in Mexico. Emerg Infect Dis 2008;14(3):429-435.
- Habing GG, Manning S, Bolin C, Cui Y, Rudrik J, Dietrich S, Kaneene JB. Within-Farm Changes in dairy farm-associated Salmonella subtypes and comparison to human clinical isolates in michigan, 2000-2001 and 2009. Appl Environ Microbiol 2015;81(17):5724-35.
- Webb HE, Brichta-Harhay DM, Brashears MM, Nightingale KK, Arthur TM, Bosilevac JM, et al. Salmonella in peripheral lymph nodes of healthy cattle at slaughter. Front Microbiol 2017;8:2214.
- Scallan E, Hoekstra RM, Angulo FJ, Tauxe RV, Widdowson M, Roy SL, et al. Foodborne illness acquired in the united states—major pathogens. Emerg Infect Dis 2011;17(1):7-15.
- Gómez-Aldapa CA, Díaz-Cruz CA, Cerna-Cortés JF, Torres-Vitela MR, Villarruel-López A, Rangel-Vargas E. Escherichia coli O157 in ground beef from local retail markets in Pachuca, Mexico. J Food Prot 2013;76(4):680-684.
- Gallegos M, Morales A, Álvarez G, Vázquez J, Morales L, Martínez I, Maldonado J. Caracterización de aislados de Escherichia coli O157:H7 en canales de bovinos y porcinos mediante PCR. Revista Científica, FCV-LUZ 2009;XIX(2):139-146.
- Chart H, Okubadejo A, Rowe B. The serological relationship between Escherichia coli O157 and Yersinia enteocolitica O9 using sera from patients with brucellosis. Epidemiol Infect 1992;108(1):77-85.
- Navarro A, Eslava C, García de la Torre G, León LA, Licona D, León L, Zarco LA, Cravioto A. Common epitopes in LPS of different Enterobacteriaceae are associated with an immune response against Escherichia coli O157 in bovine serum samples. J Med Microbiol 2007;56:1447-1454.
- Clermont O, Olier M, Hoede C, Diancourt L, Brisse S, Keroudean M, et al. Animal and human pathogenic Escherichia coli strains share common genetic backgrounds. Infect Genet Evol 2011;11(3):654-662.
- Carlos C, Pires MM, Stoppe NC, Hachich EM, Sato MI, Gomes TA, Amaral LA, Ottoboni LM. Escherichia coli phylogenetic group determination and its application in the identification of the major animal source of fecal contamination. BMC Microbiol 2010;1(10):161.
- CDC. Center for Disease Control and Prevention. Multistate outbreak of shiga toxin-producing Escherichia coli O145 infections (final update). https://www.cdc.gov/ecoli/2012/o145-06-12/index.html. Accessed Mar 5, 2019.
- Blanco M, Blanco JE, Mora A, Dhabi E, Alonso MP, González EA, et al. Serovars, virulence genes, and intimin types of Shiga toxin (verotoxin)-producing Escherichia coli isolates from cattle in Spain and identification of a new intimin variant gene (eae-xi). J Clin Microbiol 2004;42(2):645-651.
- Bettelheim KA. The Non-O157 Shiga-Toxigenic (Verocytotoxigenic) Escherichia coli; under-rated pathogens. Crit Rev Microbiol 2007;33(1):67-87.
- De Moura C, Ludovico M, Valadares GF, Gatti MSV, Leite DS. Detection of virulence genes in Escherichia coli strains isolated from diarrheic and healthy feces of dairy calves in Brazil. Arq Inst Biol 2012;79(2):273-276.
- Martínez-Castillo A, Muniesa M. Implications of free Shiga toxin-converting bacteriophages occurring outside bacteria for the evolution and the detection of Shiga toxin producing Escherichia coli. Front Cell Infect Microbiol 2014;4:46.
- Hallewell J, Niu YD, Munns K, McAllister TA, Johnson RP, Ackermann HW, Thomas JE, Standford K. Differing populations of endemic bacteriophages in cattle shedding high and low numbers of Escherichia coli O157:H7 Bacteria in feces. Appl Environ Microbiol 2014;80(13):3819-3825.
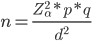 ; n= tamaño de muestra; Zα2 = valor de Z en una distribución normal Zα = 1.96 cuando α= 0.05; p= proporción de la población con la característica estudiada (si se desconoce se emplea 0.5, como en este caso); q= proporción aproximada de la población carente de la característica estudiada(1-p); d= error o precisión deseada, la cual se fijó en 10% (0.1).
; n= tamaño de muestra; Zα2 = valor de Z en una distribución normal Zα = 1.96 cuando α= 0.05; p= proporción de la población con la característica estudiada (si se desconoce se emplea 0.5, como en este caso); q= proporción aproximada de la población carente de la característica estudiada(1-p); d= error o precisión deseada, la cual se fijó en 10% (0.1).




