INTRODUCCIÓN
La mastitis
bovina es una enfermedad compleja con alta prevalencia a nivel mundial, por lo
que ésta continua siendo un problema para la industria lechera, debido a las
pérdidas económicas por la reducción en la producción de leche, desecho del
producto, descarte temprano de animales enfermos y servicios veterinarios(1).
Se han asociado a la enfermedad diversas especies de microorganismos, sin
embargo, solo un grupo reducido de especies bacterianas son los agentes
causales más comunes de la enfermedad, que de acuerdo con su origen se
clasifican como patógenos contagiosos o ambientales(2). Los
primeros, son aquellos adaptados a sobrevivir dentro de la glándula mamaria y
pueden causar infecciones asintomáticas. Los principales miembros de este grupo
son Staphylococcus aureus, Streptococcus agalactiae y Mycoplasma
spp(2,3). Por otro lado, los patógenos ambientales son oportunistas
y no están adaptados a sobrevivir en la glándula mamaria. Este grupo incluye a Streptococcus
spp no agalactiae, Enterococcus spp, Staphylococus
coagulasa-negativos y bacterias coliformes. De estos últimos el patógeno más
importante es Escherichia coli(2,3).
Generalmente, E.
coli es considerada una bacteria no patógena que habita en el intestino de
humano y animales. No obstante, se han descrito varios patotipos clasificados
como patógenos intestinales o extraintestinales(4). Para el caso de E.
coli causante de mastitis, a la fecha no se han identificado cepas o
factores de virulencia específicos, pero se ha propuesto que forman un nuevo
patotipo denominado Mammary Pathogenic E. coli (MPEC)(5). Sin
embargo, se ha observado gran diversidad tanto genética como en los factores de
virulencia entre esas bacterias(6,7). De estos últimos factores,
muchos son comunes con los patotipos de humanos e incluyen toxinas, adhesinas,
producción de cápsula, la habilidad para resistir el complemento sérico y
sistemas de adquisición de hierro(7-9).
Entre las
estrategias para el control de la mastitis, la principal es la terapia con
antibióticos, aunque frecuentemente el tratamiento falla debido a la creciente
resistencia bacteriana a dichos compuestos(10). Además, el uso
indiscriminado de antibióticos para el tratamiento de infecciones y como
promotores de crecimiento en animales, ha favorecido el desarrollo de
resistencia bacteriana, que puede ser transmitida a patógenos de animales o
humanos(11). Por esto, el ganado bovino es considerado un reservorio
importante de bacterias que llevan genes de resistencia para antibióticos
beta-lactámicos de espectro extendido, tetraciclinas, quinolonas,
aminoglucósidos, entre otros(10). Por lo anterior, la mastitis
bovina es considerada un problema de salud pública debido a la contaminación de
la leche con bacterias patógenas resistentes a los antibióticos(12).
Entre
los principales mecanismos de resistencia bacteriana a los antibióticos se
encuentran la producción de beta-lactamasas de espectro extendido (BLEE),
especialmente en bacterias Gram negativas. Las principales familias de BLEE son
las de tipo CTX-M, TEM, CMY, SHV y OXA(13,14). De acuerdo con lo
anterior, se ha observado incremento en la detección de beta-lactamasas en
bacterias de origen animal(12). También, la resistencia a
antibióticos en bacterias Gram negativas incluye la presencia de sistemas de
expulsión, como los codificados por los genes tetA-G, asociados con
resistencia a tetraciclina(15). De igual forma, la resistencia puede
ser debida a la presencia de enzimas que modifican e inactivan a los
antibióticos, como aquellas que confieren resistencia a estreptomicina
codificada por los genes strA-strB(16). Frecuentemente los
genes de resistencia están asociados a plásmidos conjugativos e integrones, que
son los principales elementos involucrados en la diseminación de la resistencia
a antibióticos entre bacterias Gram negativas(17).
La región
occidente de Michoacán, México, es una zona productora de leche muy importante,
sin embargo, a la fecha no hay información sobre los principales patógenos
causantes de la mastitis bovina, así como de los perfiles fenotípicos y
genotípicos de su resistencia a los antibióticos. Por tanto, el objetivo del
presente trabajo fue estudiar los patrones de resistencia a los antibióticos de
E. coli aisladas de mastitis bovina, analizar la prevalencia de genes de
virulencia y de resistencia para beta-lactámicos, tetraciclina, estreptomicina
y quinolonas, así como su asociación con integrones clase 1.
MATERIAL Y MÉTODOS
Detección
de mastitis y toma de muestras
Este
trabajo se desarrolló durante 2013, como parte de un programa regional para el
estudio de la mastitis en el occidente de Michoacán, México. En el estudio se
incluyeron nueve establos con un tamaño de 7 a 64 vacas. La mastitis se detectó
mediante la aplicación de la prueba de California (PC) (Sanfer, México),
siguiendo las instrucciones del fabricante. La PC se realizó en los cuatro
cuartos de cada vaca, y de los cuartos que dieron positivo a la prueba se colectó
una muestra de 30 ml de leche en tubos estériles. Previo a la colecta de la
muestra, los cuartos se desinfectaron con algodón humedecido con alcohol al 70
%, desechando los primeros chorros de leche. Las muestras colectadas se
transportaron a 4 °C en hielo al laboratorio y se procesaron el mismo día.
Aislamiento de E. coli
De
cada una de las muestras se inocularon 100 µl en agar MacConkey (Difco, USA) y
se incubaron por 24 h a 37 °C. A partir de las muestras en que se observó
crecimiento, se seleccionó al azar una colonia fermentadora de lactosa para su
análisis, la cual fue estriada en agar Eosina Azul de Metileno (Difco, USA) y
se le realizó tinción de Gram. De estas últimas se seleccionaron 34 colonias
típicas de E. coli y se identificaron bioquímicamente de acuerdo a la
literatura(18). Las principales pruebas que se realizaron fueron
fermentación de glucosa y lactosa, así como producción de gas y H2S
en agar Kligler con Hierro, IMViC (indol, rojo de metilo, Voges-Proskauer y
citrato), oxidasa, catalasa, ureasa y producción de fluorescencia en medio
EC-MUG.
Extracción de ADN
El ADN se
preparó a partir de 500 µl de un cultivo de toda la noche. Las células se
centrifugaron a 13,000 rpm por 2 min. Después, el sobrenadante se descartó y
las células se resuspendieron en 1 ml de agua destilada estéril y se llevaron a
ebullición por 10 min. La mezcla resultante se centrifugó por 5 min a 13,000
rpm y el sobrenadante se utilizó directamente como DNA molde o se almacenó a
-20 °C hasta su uso(19).
Análisis molecular de los
aislados de E. coli
Para
confirmar la identidad de los aislados se amplificaron fragmentos de los genes uidA
y lacZ con los primers uidA-F/uidA-R y lacZ-F/lacZ-R, respectivamente (Cuadro
1). Las amplificaciones por PCR se realizaron de acuerdo a la metodología
descrita(20). Por otro lado, mediante PCR multiplex con los primers
enlistados en el Cuadro 1, se analizó la presencia de genes de virulencia
característicos de las dos principales variantes de E. coli patógenas de
humanos, E. coli enteropatógena (EPEC) y E. coli
enterohemorrágica (EHEC). Los genes analizados fueron eaeA, que codifica
para la intimina y es común a EPEC y EHEC, bfpA, que codifica para la
pilina del pilus tipo IV de EPEC y stx2, que codifica para la toxina
Shiga tipo 2 de EHEC. Las reacciones de PCR se realizaron como se describió
previamente(21).
Para
la detección de los genes de resistencia en E. coli, los extractos de
ADN total se utilizaron para amplificar por PCR fragmentos de los genes blaTEM,
blaSHV, blaCTX-M, blaCMY
y blaOXA(22). También, se amplificaron los genes
de resistencia a tetraciclina tetA y tetB con primers descritos
previamente(22,23), así como genes de resistencia a estreptomicina strA
y strB(24) y de resistencia a quinolonas qnrA y qnrB(25).
Los genes se seleccionaron debido a los altos índices de resistencia de las E.
coli a los antibióticos beta-lactámicos, tetraciclina y estreptomicina.
Además, los genes de resistencia se han asociado frecuentemente a integrones
clase 1, por lo cual se analizó la presencia del gen que codifica para la
integrasa intI1(26). Las características de las secuencias de
los primers se enlistan también en el Cuadro 1. En general, las mezclas de
reacción para las amplificaciones se realizaron en un volumen de 25 µl conteniendo:
1X de Master mix (Promega, USA), 0.5 µM de cada primer y 1 µl de extracto total
de ADN. Las reacciones de PCR se realizaron en un termociclador C1000 (Bio-Rad, México). La amplificación se inició con un
ciclo de desnaturalización a 95 °C por 5 min seguido de 35 ciclos a las
siguientes temperaturas: desnaturalización a 95 °C por 40 seg, alineamiento a
la temperatura indicada en el Cuadro 1 para cada par de oligonucleótidos por 40
seg, extensión a 72 °C por 1 min y un ciclo final de extensión a 72 °C por 5
min. Los fragmentos 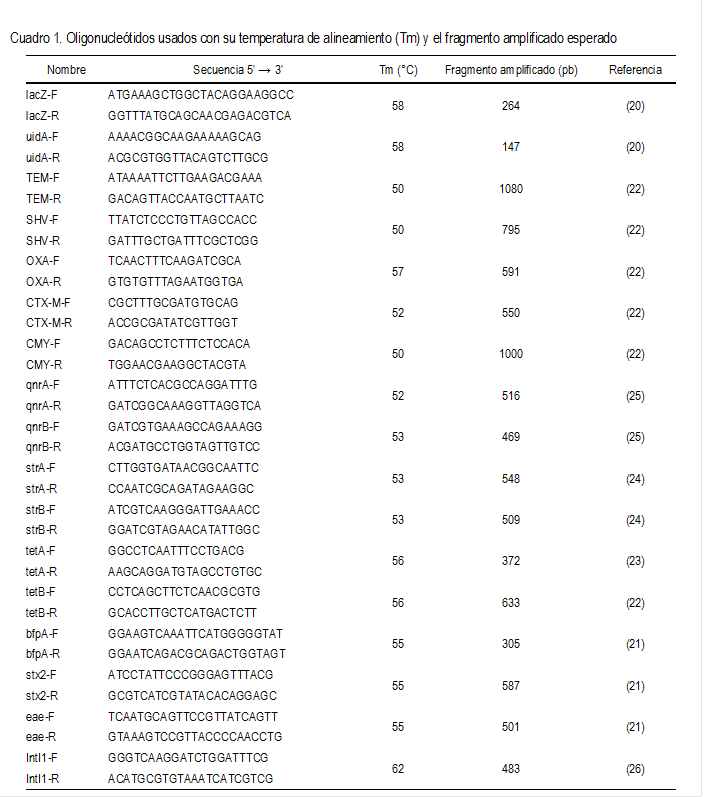
amplificados se
separaron por electroforesis en geles de agarosa al 1%, se tiñeron con bromuro
de etidio y se visualizaron en un fotodocumentador Gel Doc Universal Hood II
(Bio-Rad, México).
Pruebas
de resistencia a los antibióticos
|
 |
Las pruebas de
susceptibilidad se realizaron por ensayos de difusión en disco en agar Mueller-Hinton
(Difco, USA), para lo cual se preparó una suspensión bacteriana equivalente a
la turbidez del tubo 0.5 de la escala de McFarland (1-2 x 108
UFC/ml). Después, se distribuyeron 200 μl de la suspensión bacteriana
sobre la superficie del agar Müeller-Hinton (Difco, USA). Posteriormente, se
colocaron los multidiscos para Gram negativos II (Bio-Rad, México), con los
siguientes antibióticos a las cantidades indicadas: amikacina (30 µg),
ampicilina (10 µg), levofloxacina (5 µg), cefalotina (30 µg), cefotaxima (30
µg), ceftriaxona (30 µg), cloramfenicol (30 µg), gentamicina (10 µg),
netilmicina (30 µg), nitrofurantoina (300 µg), cefepima (30 µg) y trimetoprim-sulfametoxazol
(25 µg). Las zonas de inhibición se midieron, y de acuerdo a las instrucciones
del fabricante los aislados se clasificaron como resistentes (R), intermedios
(I) o sensibles (S). La resistencia a tetraciclina, kanamicina y estreptomicina
(Sigma Aldrich, México) se analizó mediante el método de microdilución y se
interpretó de acuerdo a los criterios del CLSI(27). Los aislados se
clasificaron como resistentes si las concentraciones mínimas inhibitorias (CIM)
fueron ≥16 µg/ml para tetraciclina, ≥25 µg/ml para kanamicina y
≥32 µg/ml para estreptomicina.
RESULTADOS
De acuerdo a
la PC, la prevalencia de mastitis subclínica en el área estudiada fue de 49 %,
ya que 148 de 302 vacas dieron positivo a la prueba en al menos uno de los
cuartos. En total se colectaron 120 muestras de leche de las vacas con
mastitis, 28 no se pudieron obtener por dificultades técnicas durante la toma
de muestras. De las muestras colectadas, en 51 (42.5 %) se obtuvo crecimiento
en agar MacConkey, de éstas se seleccionó una colonia representativa
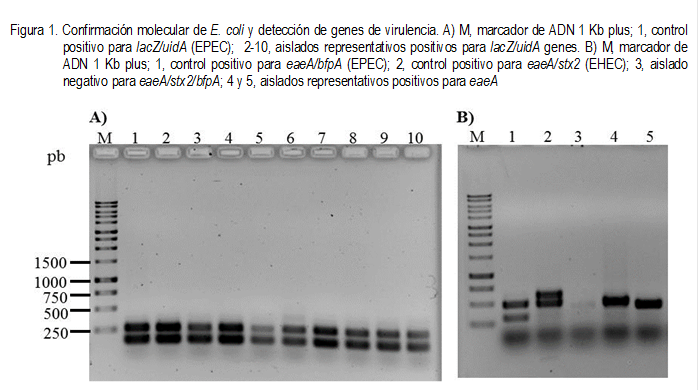
fermentadora de lactosa y se confirmó como Gram negativa. Treinta y cuatro
(66.7 %) colonias fermentadoras de lactosa se identificaron bioquímicamente
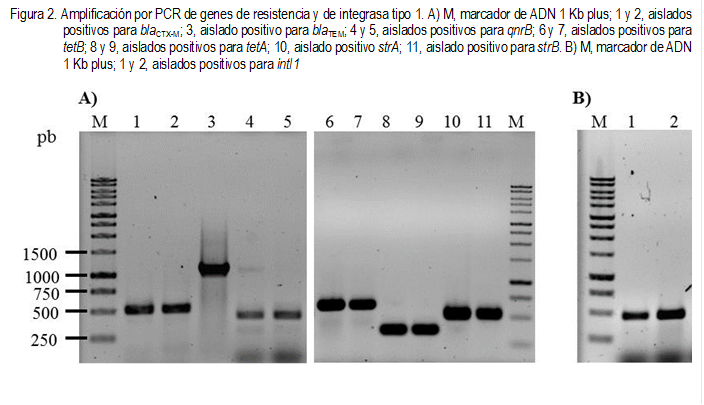
como E. coli y en todas ellas se amplificaron fragmentos de los genes uidA
y lacZ (Figura 1A). Además, en 4/34 (11.8 %) aislados de E. coli,
se detectó el gen de virulencia para la intimina (eaeA), pero en ningún
aislado se identificaron los genes de virulencia bfpA o stx2
(Figura 1B).
Perfiles de
resistencia a los antibióticos
Los ensayos de
susceptibilidad a los antimicro-bianos de las E. coli (n= 34) revelaron
que todas fueron resistentes a uno o más antibióticos (Cuadro 2). Los
compuestos a los cuales mostraron mayores índices de resistencia fueron a
tetraciclina (88.2 %), ampicilina (76.5 %) y cefalotina (73.5 %). Índices de
resistencia menores que van de 38.2 al 2.9 % se observaron en los restantes 12
antibióticos. Además, se observó resistencia intermedia principalmente para
cefotaxima (64.7 %), nitrofurantoína (52.9 %) y ceftriaxona (47 %). Por otro
lado, se observaron altos índices de susceptibilidad para
trimetoprim-sulfametoxazol (97 %), levofloxacina (91.2 %), kanamicina (88.2 %),
cefepima (85.3 %), netilmicina (85.3 %), gentamicina (82.4 %), amikacina (73.5
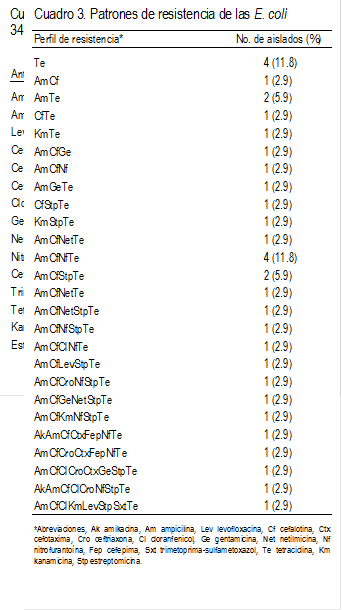
%), cloranfenicol y estreptomicina (61.8 %), respectivamente. Se observaron 26 patrones
de resistencia distintos entre los 34 aislados de E. coli, siendo los
patrones observados con mayor frecuencia, los de resistencia a tetraciclina y
ampicilina-cefalotina-nitrofurantoína-tetraciclina con cuatro aislados cada
uno. Adicionalmente, 25 (73.5 %) aislados se clasificaron como
multirresistentes, ya que toleraron tres o más antibióticos diferentes, de los
cuales, 12 (35.3 %) presentaron resistencia de 5 a 8 antibióticos (Cuadro 3).
Presencia de genes de
resistencia e integrones clase 1 en E. coli
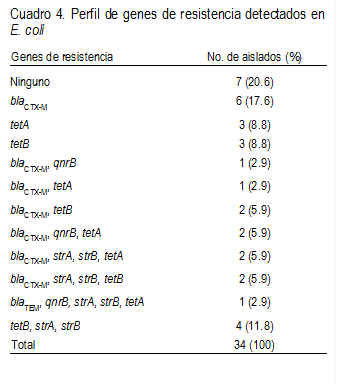
De los 11
genes de resistencia analizados, se logró detectar la presencia de 7 en 27
(79.4 %) E. coli (Figura 2A). El más frecuente fue blaCTX-M
presente en 16 (47 %) aislados, seguido por tetB en 11 (32.3 %), tetA,
strA, strB en 9 (26.5%), respectivamente; qnrB en 4
aislados y blaTEM en 1 (2.9 %) aislado (Cuadro 4).
Adicionalmente, los genes de resistencia identificados se presentaron solos (12
aislados) o en diferentes combinaciones (15 aislados). De estos últimos, 10
aislados positivos
para blaCTX-M
presentaron seis arreglos distintos con
los genes strA,
strA, tetA, tetB y qnrB; mientras que el arreglo tetB-strA-strB
se encontró en cuatro aislados y blaTEM-tetA-strA-strB
en un aislado. Sin embargo, de las 34 E. coli, solo dos (5.9 %) aislados
multirresistentes contienen el gen de la integrasa para integrones clase 1 (Figura
2B). De los cuales, uno presentó el gen tetB, mientras que en el segundo
se identificó el arreglo blaCTX-M-tetA-strA-strB.
DISCUSIÓN
La mastitis
bovina es la enfermedad más importante del ganado bovino a nivel mundial. Su
prevalencia, tipo de microorganismos causales y sus características son variables
de un lugar a otro. E. coli es un patógeno ambiental importante causante
de mastitis; cuando esta bacteria infecta la glándula mamaria se muestran
síntomas de inflamación, reducción en la producción y cambios físico-químicos
en la leche(28).
Las E. coli
aisladas en este trabajo pertenecen a 34
muestras distintas, lo cual representa un 28.3 % de muestras positivas
para esta bacteria. Datos similares en la frecuencia de aislamiento de E.
coli han sido reportados(29), donde el 27.2 % de las muestras
colectadas de vacas con mastitis fueron positivas para E. coli. Además,
en cuatro de las E. coli se detectó el gen de virulencia de la intimina,
aunque no se detectaron los genes bfpA o sxt2. Lo anterior
sugiere que esas bacterias pueden ser E. coli enteropatógena atípicas
(aECEP) o E. coli enterohemorrágica (ECEH) con otra variante del gen stx(4).
Sin embargo, el repertorio de genes de virulencia analizado en este trabajo fue
limitado y se requieren más estudios para estudiar el catálogo completo de
genes en las E. coli causantes de mastitis. En este sentido, se sabe que
el ganado bovino es un reservorio importante de variantes de E. coli
patógenas de humano, y varios estudios han reportado que los patotipos de
humano también están asociados con mastitis bovina(30-32). Por
ejemplo, se han identificado diferentes arreglos de los genes stx1,
stx2 y eaeA en E. coli aislada de mastitis
bovina(30). También se han descrito aislados de E. coli
asociados a mastitis que tienen el gen eaeA sólo o en combinación con
otros genes de virulencia, pero esos aislados fueron negativos para stx1
o stx2(32). Aunque muchos de los genes
identificados en E. coli aislada de mastitis bovina no son específicos
para la enfermedad, se ha sugerido que esos y otros elementos aun no
identificados pueden ser responsables de la mastitis(6). En este
sentido, varios estudios se han enfocado en la asociación de factores de
virulencia específicos de E. coli causante de mastitis, pero no lo han
logrado(6,33,34).
Normalmente,
para el control de la mastitis bovina se emplea la terapia con antibióticos;
sin embargo, muchas veces el tratamiento falla debido al incremento en la
resistencia bacteriana a los antimicrobianos(10). En este sentido,
las E. coli aisladas de mastitis bovina han desarrollado resistencia a
los antibióticos comúnmente usados en medicina veterinaria para tratar la
enfermedad(35). En este estudio se encontraron elevados niveles de
resistencia para tetraciclina (88.2 %), ampicilina (76.5 %) y cefalotina (73.5
%), principalmente. Lo anterior puede estar relacionado con el hecho de que
esos compuestos pertenecen a los principales grupos de antibióticos usados para
el tratamiento de infecciones en ganado o se utilizan como promotores de
crecimiento animal(36). En este sentido, se ha encontrado que E.
coli aislada de mastitis bovina presentó resistencia a ampicilina (47.1 %),
kanamicina (37.1 %) y estreptomicina (32.9 %)(7). Mientras que otros
autores han encontrado altos índices de resistencia a ampicilina (98.4 %),
estreptomicina (40.3 %) sulfisoxazol (34.1 %) y tetraciclina (24.8 %), además
de otros antibióticos(37). Asimismo, 64.7 % de las E. coli
analizadas en este estudio fueron multirresistentes, ya que fueron resistentes
a tres o más antibióticos. Datos similares se han reportado(10),
donde el análisis de 70 aislados de E. coli resistentes a los
antibióticos mostró que el 62.8 % fueron multirresistentes.
La
resistencia bacteriana a los antibióticos es debida a la presencia de genes que
le confieren la capacidad de tolerar esos compuestos a través de mecanismos
como expulsión, modificación, entre otros. Asimismo, el ganado bovino se
considera un reservorio de E. coli, las cuales frecuentemente tienen
genes de resistencia a antibióticos que se pueden diseminar a través de los
alimentos de origen animal(11). De acuerdo con lo anterior, en este
estudio se encontraron genes que codifican para beta-lactamasas, tetraciclina y
estreptomicina en el 79.4 % de las E. coli aisladas de mastitis bovina y
todas ellas presentaron resistencia o resistencia intermedia a esos
antibióticos. Lo anterior demuestra que en el área de estudio de este trabajo,
el ganado bovino es reservorio de bacterias resistentes a los antibióticos, con
la alta prevalencia de genes de resistencia a los antibióticos y que aunado a
la presencia de genes de virulencia, representa un riesgo a la salud por la
contaminación de la leche. Otros autores han encontrado genes para BLEE en el
26 y 1.5 % de enterobacterias aisladas de excremento y leche de vacas con
mastitis, respectivamente. En ese trabajo, se detectaron los genes blaCTX-M,
blaSHV y blaTEM en 92, 5.5 y 2.2 % de
las bacterias positivas para BLEE(38). Además, diversos estudios han
encontrado que E. coli multirresistentes causantes de mastitis contienen
los genes blaCTX-M y blaTEM(39,40).
También, el
análisis genético de los determinantes de resistencia a tetraciclina y
estreptomicina en E. coli aislada de mastitis bovina, reveló que todas
contenían uno o más genes de resistencia a esos compuestos en diferentes
combinaciones(37). Además, en 33 E. coli analizadas en este
trabajo presentaron al menos un plásmido grande (>50 kb, datos no
mostrados), sugiriendo que los genes de resistencia pueden estar asociados con
estos. De acuerdo con lo anterior se ha asociado a plásmidos >35 kb con la
presencia y movilización de genes de resistencia a antibióticos(41).
Además, dos aislados presentaron integrones clase 1, cuya presencia se
correlaciona comúnmente con aislados multirresistentes, sugiriendo que los
genes pueden estar asociados a esos elementos. La asociación de genes de
resistencia con integrones ha sido observada en otros estudios de E. coli
causante de mastitis con altos índices de resistencia a los antibióticos(42).
CONCLUSIONES E
IMPLICACIONES
Se aislaron e
identificaron E. coli asociadas a mastitis bovina y resistentes a
antibióticos; aunque en baja frecuencia, éstas contienen genes de virulencia
relacionados con patógenos entéricos de humano. También presentaron genes
responsables de la resistencia a compuestos beta-lactámicos, tetraciclina,
estreptomicina y quinolonas. Hasta donde se sabe, ésta es la primera vez que se
determina la presencia de dichos genes de resistencia a antibióticos en E.
coli asociada a mastitis bovina en México. Aunque se requieren más estudios
para caracterizar el repertorio de genes de virulencia
y de resistencia e investigar su movilidad por su posible asociación con
plásmidos conjugativos. El estudio de las principales características de
las bacterias patógenas asociadas a mastitis bovina, en el futuro podría
contribuir a mejorar la situación de la enfermedad en los hatos ganaderos del
estado, así como los riesgos a la salud asociados.
AGRADECIMIENTOS
Se
agradece a Carlos Cervantes y Jesús Silva los comentarios al presente trabajo.
Este trabajo fue financiado en parte por el apoyo del programa de desarrollo
profesional docente (PRODEP), Fortalecimiento de Cuerpos Académicos (proyecto
IDCA-11106) y por la Universidad de La Ciénega del Estado
de Michoacán de Ocampo (UCEM).
LITERATURA
CITADA
1. Seegers H, Fourichon
C, Beaudeau F. Production effects related to mastitis and mastitis economics in
dairy cattle herds. Vet Res 2003;34(5):475-491.
2. Thompson-Crispi K, Atalla H, Miglior F, Mallard BA. Bovine mastitis:
Frontiers in immunogenetics. Front Immunol 2014;5:1-10.
3. Contreras
GA, Rodríguez JM. Mastitis: comparative etiology and epidemiology. J Mammary
Gland Biol Neoplasia 2011;16(4):339-356.
4. Kaper JB,
Nataro JP, Mobley HL. Pathogenic Escherichia coli. Nat Rev Microbiol
2004;2(2):123-140.
5. Shpigel NY,
Elazar S, Rosenshine I. Mammary pathogenic Escherichia coli. Curr Opin
Microbiol 2008;11(1):60-65.
6. Blum SE,
Leitner G. Genotyping and virulence factors assessment of bovine mastitis Escherichia
coli. Vet Microbiol 2013;163(3-4):305-312.
7. Liu Y, Liu
G, Liu W, Ali T, Chen W, Yin J, et al. Phylogenetic group, virulence
factors and antimicrobial resistance of Escherichia coli associated with
bovine mastitis. Res Microbiol 2014;165(4):273-277.
8. Blum SE,
Heller ED, Sela S, Elad D, Edery N, Leitner G. Genomic and phenomic study of
mammary pathogenic Escherichia coli. PLoS One. 2015;10(9):1-24.
9. Hogan J,
Larry Smith K. Coliform mastitis. Vet Res 2003;34(5):507-519.
10. Saini V,
McClure JT, Léger D, Keefe GP, Scholl DT, Morck DW, et al. Antimicrobial
resistance profiles of common mastitis pathogens on Canadian dairy farms. J
Dairy Sci 2012;95(8):4319-4332.
11. Phillips I,
Casewell M, Cox T, De Groot B, Friis C, Jones R, et al. Does the use of antibiotics
in food animals pose a risk to human health? A critical review of published
data. J Antimicrob Chemother 2004;53(1):28-52.
12. Li XZ,
Mehrotra M, Ghimire S, Adewoye L. beta-Lactam resistance and beta-lactamases in
bacteria of animal origin. Vet Microbiol 2007;121(3-4):197-214.
13. Jacoby GA,
Munoz-Price LS. The new beta-lactamases. N Engl J Med 2005;352(4):380-391.
14. Bush K,
Jacoby GA. Updated functional classification of beta-lactamases. Antimicrob
Agents Chemother 2010;54(3):969-976.
15. Nguyen
F, Starosta AL, Arenz S, Sohmen D, Dönhöfer A, Wilson DN. Tetracycline
antibiotics and resistance mechanisms. Biol Chem 2014;395(5):559-575.
16. Sunde M,
Norström M. The genetic background for streptomycin resistance in Escherichia
coli influences the distribution of MICs. J Antimicrob Chemother
2005;56(1):87-90.
17. Gillings MR.
Integrons: past, present, and future. Microbiol Mol Biol Rev 2014;78(2):257-277.
18. Scheutz
F, Strockbine N. Escherichia. Bergey's Manual of Systematics of Archaea
and Bacteria. 2015:1-49.
19. Madico G, Akopyants NS, Berg DE. Arbitrarily primed PCR DNA
fingerprinting of Escherichia coli O157:H7 strains by using templates
from boiled cultures. J Clin Microbiol 1995;33(6):1534-1546.
20. Horakova K,
Mlejnkova H, Mlejnek P. Specific detection of Escherichia coli isolated
from water samples using polymerase chain reaction targeting four genes:
cytochrome bd complex, lactose permease, beta-D-glucuronidase, and
beta-D-galactosidase. J Appl Microbiol 2008;105(4):970-976.
21. Vidal M,
Kruger E, Durán C, Lagos R, Levine M, Prado V, et al. Single
multiplex PCR assay to identify simultaneously the six categories of
diarrheagenic Escherichia coli associated with enteric infections. J
Clin Microbiol 2005;43(10):5362-5365.
22. Ahmed
AM, Shimamoto T. Molecular characterization of multidrug-resistant avian
pathogenic Escherichia coli isolated from septicemic broilers. Int J Med
Microbiol 2013;303(8):475-483.
23. Guillaume
G, Verbrugge D, Chasseur-Libotte M, Moens W, Collard J. PCR typing of
tetracycline resistance determinants (Tet A-E) in Salmonella enterica
serotype Hadar and in the microbial community of activated sludges from
hospital and urban wastewater treatment facilities in Belgium. FEMS Microbiol
Ecol 2000;32(1):77-85.
24. Gebreyes
WA, Altier C. Molecular characterization of multidrug-resistant Salmonella
enterica subsp. enterica serovar Typhimurium isolates from swine. J Clin
Microbiol 2002;40(8):2813-2822.
25. Robicsek A,
Strahilevitz J, Sahm DF, Jacoby GA, Hooper DC. qnr prevalence in
ceftazidime-resistant Enterobacteriaceae isolates from the United
States. Antimicrob Agents Chemother 2006;50(8):2872-2874.
26. Mazel
D, Dychinco B, Webb VA, Davies J. Antibiotic resistance in the ECOR collection:
integrons and identification of a novel aad gene. Antimicrob Agents Chemother
2000;44(6):1568-1574.
27. CLSI.
Methods for dilution antimicrobial susceptibility tests for bacteria that grow
aerobically. Ninth
ed. Approved Standard M07-A9. Clinical and Laboratory Standards Institute.
2012.
28. Blum SE,
Heller ED, Leitner G. Long term effects of Escherichia coli mastitis.
Vet J 2014;201(1):72-77.
29. Momtaz H,
Safarpoor Dehkordi F, Taktaz T, Rezvani A, Yarali S. Shiga toxin-producing Escherichia
coli isolated from bovine mastitic milk: serogroups, virulence factors, and
antibiotic resistance properties. Scient World J 2012;2012:1-9.
30. Kobori D,
Rigobelo EC, Macedo C, Marin JM, Avila FA. Virulence properties of Shiga
toxin-producing Escherichia coli isolated from cases of bovine mastitis
in Brazil. Revue
d'élevage et de medcine vétérinaire des pays tropicaux 2004;57(1-2):15-20.
31. Lira WM,
Macedo C, Marin JM. The incidence of Shiga toxin-producing Escherichia coli
in cattle with mastitis in Brazil. J Appl Microbiol 2004;97(4):861-876.
32. Suojala L,
Pohjanvirta T, Simojoki H, Myllyniemi AL, Pitkälä A, Pelkonen S, et al. Phylogeny,
virulence factors and antimicrobial susceptibility of Escherichia coli
isolated in clinical bovine mastitis. Vet Microbiol 2011;147(3-4):383-398.
33. Fernandes
JB, Zanardo LG, Galvão NN, Carvalho IA, Nero LA, Moreira MA. Escherichia
coli
from clinical mastitis: serotypes and virulence factors. J Vet Diagn Invest
2011;23(6):1146-1152.
34. Kaipainen T,
Pohjanvirta T, Shpigel NY, Shwimmer A, Pyörälä S, Pelkonen S. Virulence factors
of Escherichia coli isolated from bovine clinical mastitis. Vet
Microbiol 2002;85(1):37-46.
35. Suojala L,
Kaartinen L, Pyörälä S. Treatment for bovine Escherichia coli mastitis -
an evidence-based approach. J Vet Pharmacol Ther 2013;36(6):521-531.
36. Dibner JJ,
Richards JD. Antibiotic growth promoters in agriculture: history and mode of
action. Poult Sci. 2005;84(4):634-643. doi: 10.1093/ps/84.4.634.
37. Srinivasan V, Gillespie BE,
Lewis MJ, Nguyen LT, Headrick SI, Schukken YH, et al. Phenotypic and
genotypic antimicrobial resistance patterns of Escherichia coli isolated
from dairy cows with mastitis. Vet Microbiol 2007;124(3-4):319-328.
38. Geser N, Stephan R, Hächler
H. Occurrence and characteristics of extended-spectrum β-lactamase
(ESBL) producing Enterobacteriaceae in food producing animals, minced
meat and raw milk. BMC Vet Res 2012;8(1):1-9.
39. Pehlivanoglu F, Turutoglu H,
Ozturk D. CTX-M-15-Type Extended-Spectrum Beta-Lactamase-Producing Escherichia
coli as causative agent of bovine mastitis. Foodborne Pathog Dis
2016;13(9):477-482.
40. Timofte D, Maciuca IE, Evans
NJ, Williams H, Wattret A, Fick JC, et al. Detection and molecular
characterization of Escherichia coli CTX-M-15 and Klebsiella
pneumoniae SHV-12 β-lactamases
from bovine mastitis isolates in the United Kingdom. Antimicrob Agents
Chemother 2014;58(2):789-794.
41. Freitag C, Michael BG, Kadlec K, Hassel M, Schwarz
S. Detection of plasmid-borne extended-spectrum β-lactamase (ESBL)
genes in Escherichia coli isolates from bovine mastitis. Vet
Microbiol 2017;200:151-156.
42. Wang
GQ, Wu CM, Du XD, Shen ZQ, Song LH, Chen X, et al. Characterization of
integrons-mediated antimicrobial resistance among Escherichia coli
strains isolated from bovine mastitis. Vet Microbiol
2008;127(1-2):73-78.